来源:BV1q34y1m7FC
讲者介绍

什么是单细胞测序(主要讨论RNA的测序)
为什么要做单细胞测序
假如你正在研究一份血液样本,想看看血液成份的表达谱,血液是许多不同类型细胞的复杂混合物,比如B细胞、T细胞、巨噬细胞和中性粒细胞,但是如果直接测血液内的RNA含量,显然是极为困难的(因为各种细胞都混在一起了)
就像一杯混合水果果汁一样,水果果汁里面有树莓、橙子、菠萝、香蕉、草莓五种水果,而你只对树莓和橙子感兴趣;在直接喝果汁的情况下,显然是很难去确定哪些味道是来自于树莓的
单细胞测序可以区分出细胞的种类

- 上图展示了一个来自血液样本的数据,在这张图中,成千上万的单个细胞被并行处理,每个单细胞的转录本都进行了测序和分析,可以检测到存在于人类基因组中的2万个以上的不同的基因。
- 然而,从2万个维度去描述数据是非常困难的,所以我们需要对这些数据进行降维处理,使用一些方法把它们压缩成二维的数据,我们有很多选择(比如PCA、tSNE、UMAP等),最终会得到上面的图
- 上面的图中,每一个小点代表了一个细胞,对于聚在一起的小点(细胞),它们距离越小,彼此之间的相似程度就越高,我们把这种图称为细胞群图,每个群代表了不同种类的细胞
- 这张图的意义:这张图本身的意义不大,能让我们知道我们的样本中有哪几种细胞类型,但你没办法知道他们具体是什么
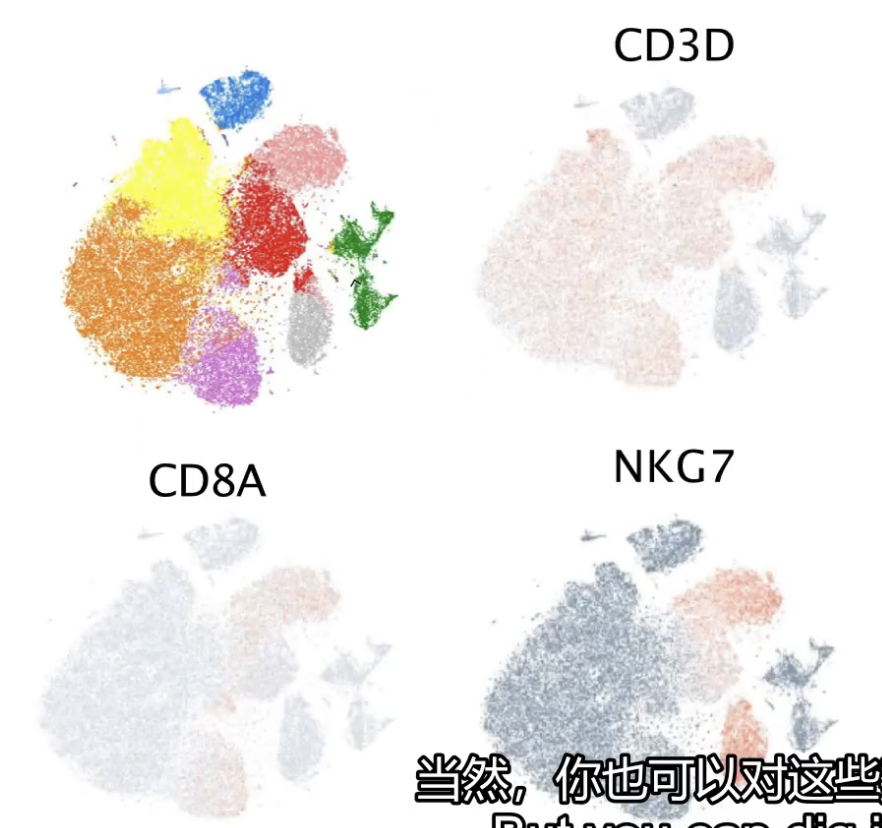
- 当然,我们可以对数据进行深入研究,比如我们可以针对某一群细胞,去探究他们具体在哪个转录本上有差异(使得两个亚群的细胞可以被区分开),我们对许多基因这样做了之后,就可以用这些信息(某一群细胞表达某些基因、而不表达另一些基因)来把细胞们分成不同的集群,就像下图这样
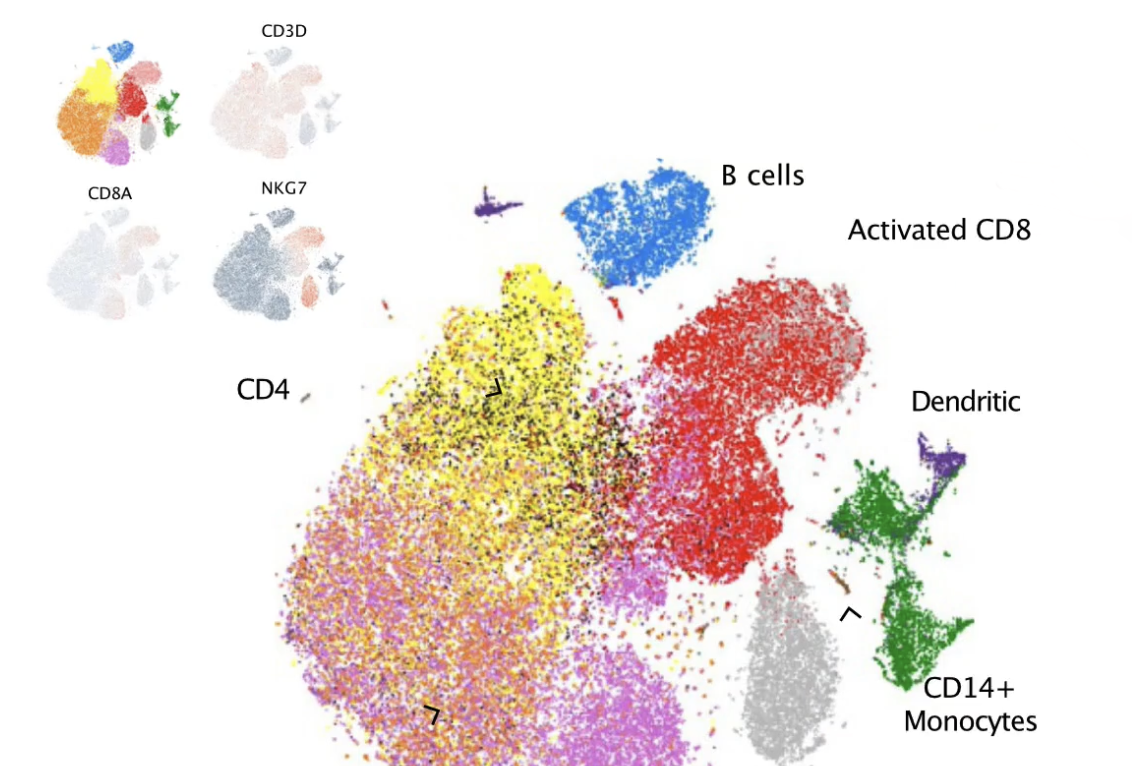
- 我们会通过观察每一群细胞的转录谱,来识别和观察很多不同的细胞类型
流式细胞学v.s.单细胞测序
- 流式细胞学也可以获得相同的结果,那么为什么我们还要进行单细胞测序呢?
- 其中一个原因是这些细胞群可以进一步细分,通过不同的基因进行重新组合分析,因此,我们可以对可能存在于样本中的多达2万个差异表达的基因进行再次度量,而使用流式细胞学和质谱流式细胞学可能使得我们受限于几十个标记中
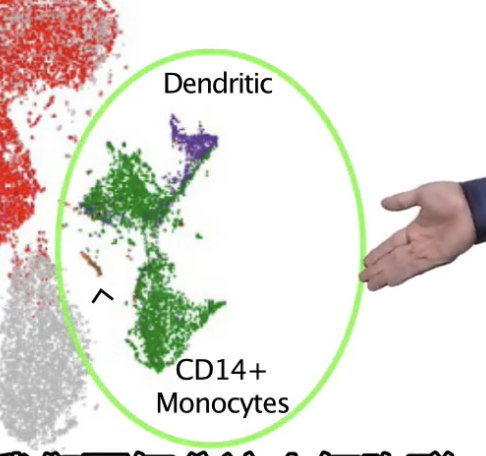
- 举例,如果我们要细分上图中这个绿色的圈里的细胞群,忽略其他的细胞,并且把这群细胞的特点分得再详细一点,放大这些特点并且重新归类,我们会把这群绿色的细胞单独拿出来,然后根据他们之间的差异,将他们进一步、更细致地重新分群

- 这个过程其实可以无限重复下去,如果我们找到了感兴趣的细胞,就可以深入研究一下,看看这群细胞中是否有明显的基因表达特征,可以进一步细分亚群,这才是单细胞测序真正的力量,我们可以得到一组非常高纬度的数据,这是其他方法无法获得的
单细胞测序在过去的10年使用得越来越多
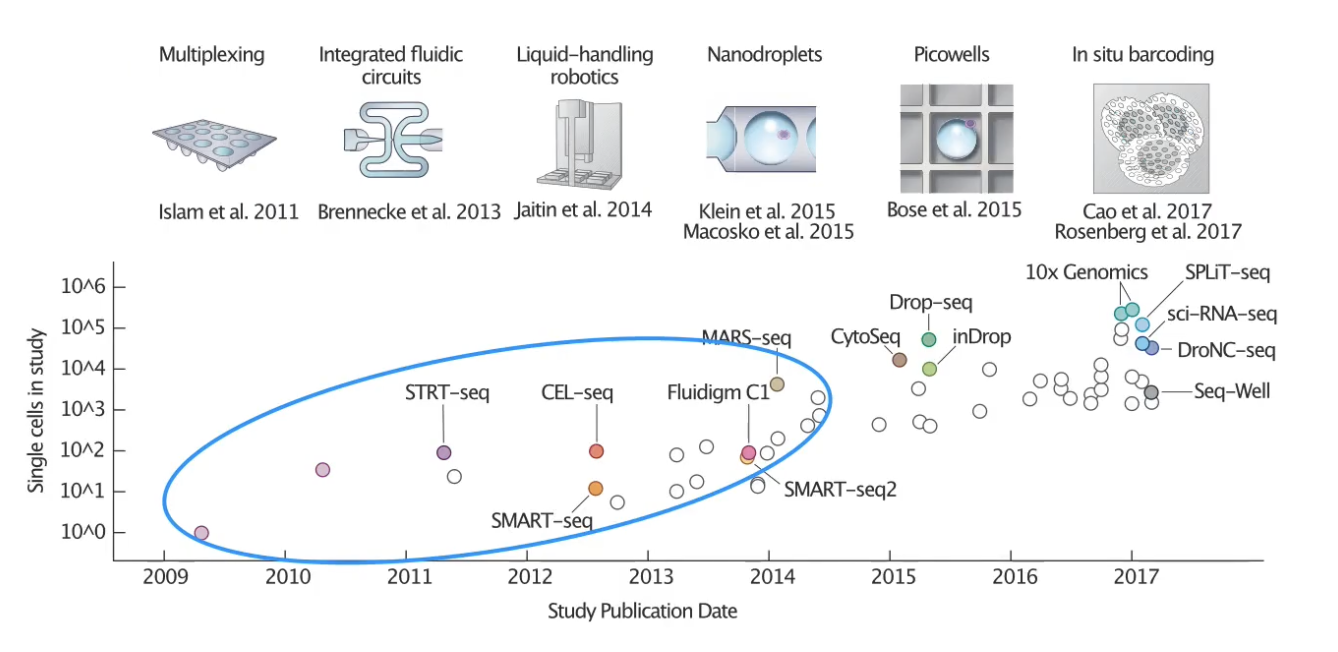
- 2009年,世界上第一次有人进行了单细胞测序,他测序了1个细胞
- 多年来,在不同的研究中,细胞的数量呈现指数级的增长,上图里的每一个圈都对应着不同的出版物和分析的细胞数量
平板法plate-based methods
- 把每个细胞分到平板的不同孔中,对每个细胞单独建库,然后进行测序
- 这种方法也可以叫基于平板的SMART-seq计数,plate-based SMART-seq
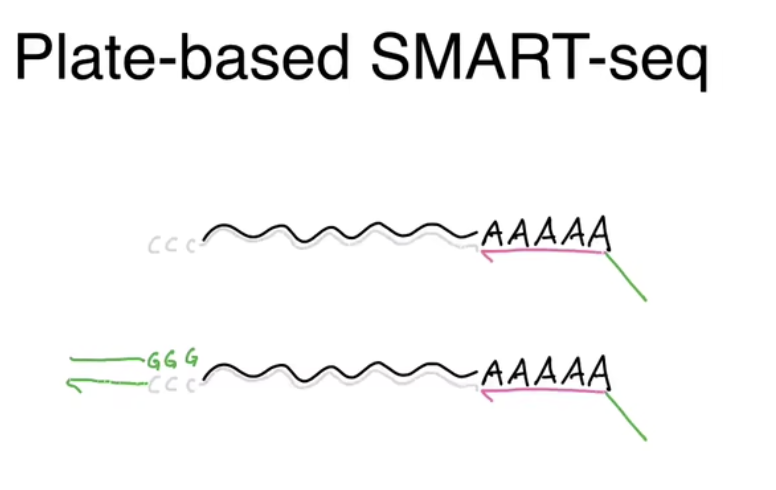
- 使用流式细胞仪对意向细胞进行分类,把一个细胞放到96或者384孔板的每个孔中,然后裂解细胞,再使用SMART-seq进行逆转录反应建库
- 在这种建库方法中,我们有一个oligo-dT(多聚T重复的寡核苷酸),用于引发逆转录反应(与有poly-A尾的mRNA),oligo-dT的尾端有一个把手(oligo-dG)
- 逆转录酶使用RNA作为模版合成DNA,这种合成的DNA末端会在酶的作用下,添加几个非模版的C碱基,这些C碱基会被用来与末端带有G的模版转换寡核苷酸结合
- 接下来,当我们在反应体系中加入模版转换用的oligo-dG上,逆转录酶就能使用这种模版转录核苷酸作为进一步聚合的模版,最后我们就得到了两边都有PCR把手的cDNA
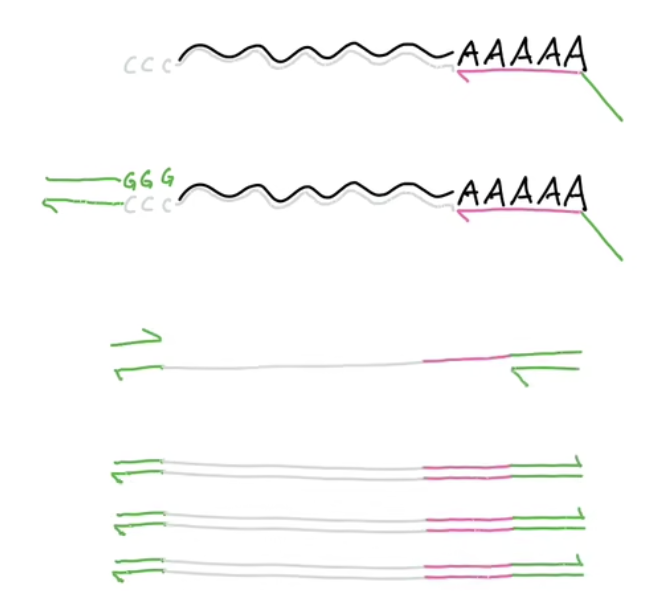
- 接着,我们进行PCR反应后,每种cDNA就可以得到很多很多个拷贝,然后我们可以开始进入样品制备,这个方法叫Nextera DNA文库制备法
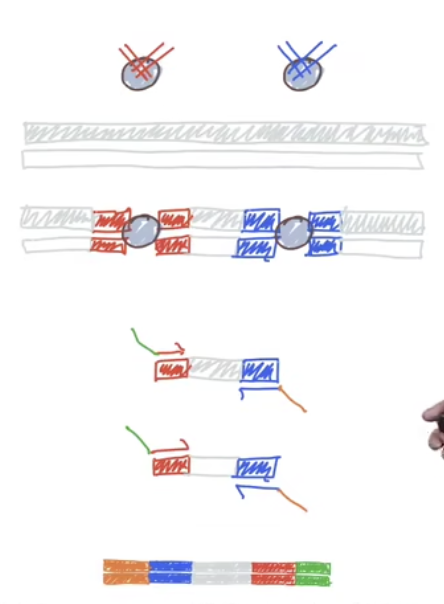
- 最下面的DNA链实际有两条
- Nextera转座酶会将cDNA片段化为更小的片段,添加部份的illumina接头,然后通过PCR扩增这些物质,并完成文库的准备过程
- 这种方法的上限是几百和几千个样本,但这会有很多工作量,因为每个细胞都要分别处理,有大量的移液、加样、点样的过程,这些过程都是手动的;而且,其实这种方法相当昂贵,因为相当于每个细胞都要进行单独的DNA库样品制备
微流体法microfluid-based methods
Dropseq
- 本质是使用微流体芯片,或者说非常小的液滴,并在这些液滴中进行分子生物学过程
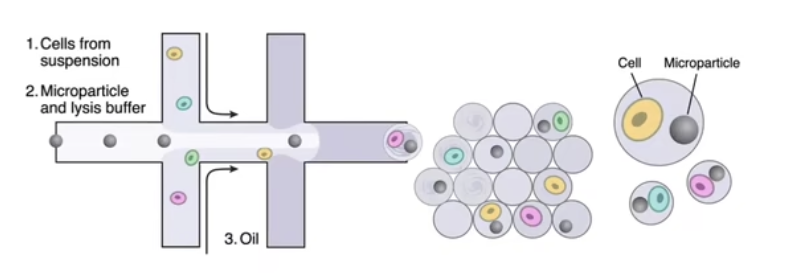
- 在芯片(反应场所)中,磁珠beads从左边流入,细胞从第一个连接处进入,这时细胞和磁珠会有不均匀的混合,混合物在下一个入口会遇到流入的油,油的加入会使水流截断并形成乳液,也就是油包水,水里可能有磁珠和/或细胞
- 我们最终会得到很多液滴,这些液滴中大部份是空的,有些液滴会包含有磁珠,有些液滴会包含细胞,一小部份液滴中会同时有一个磁珠和一个细胞
- 磁珠的特别之处1:磁珠表面含有大量的oligo-dT,这些oligo-dT可以用于捕获和磁珠在一个液滴中的细胞的mRNA
- 磁珠的特别之处2:每个磁珠的oligo-dT后会含有特殊的一段序列,被称为bardode
- 接着RNA被捕获到磁珠上,我们会把液滴打破,然后进行逆转录反应(下图1到下图2),这样每个磁珠上逆转录出的cDNA都会有相同的barcode
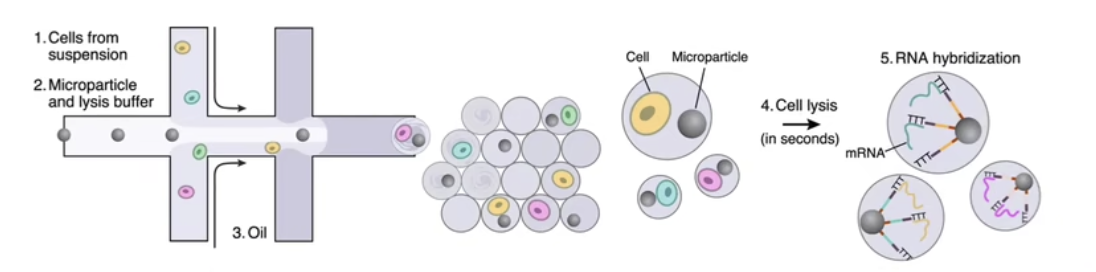
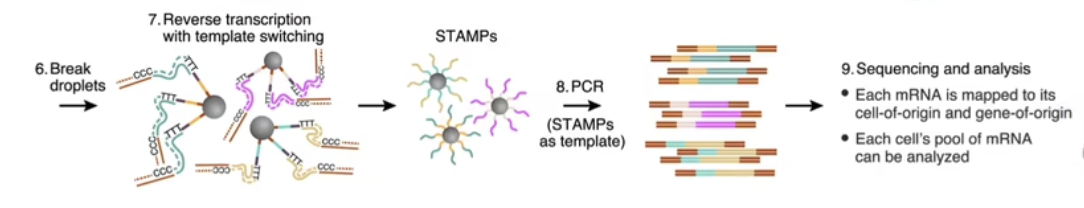
- 接着,我们可以准备DNA测序库,对所有转录本的末端进行测序;由于我们已经对每一个转录本都做了对应的标记,而且都带着用于对应一个细胞的barcode
- 用这种方法,我们可以去并行处理很多很多管细胞,可以处理上千甚至上万的细胞
这种方法的缺点
- 只有液滴中的一少部份含有磁珠,绝大部分液滴都不含有任何物质
- 细胞和磁珠是随机封装在这些液滴中的,所以也会有一个液滴里封装了2+个细胞和/或磁珠的情况,而且这种情况会随着你增加细胞和磁珠的浓度来而增加,而且没有办法将它们在测序的过程中区分开来
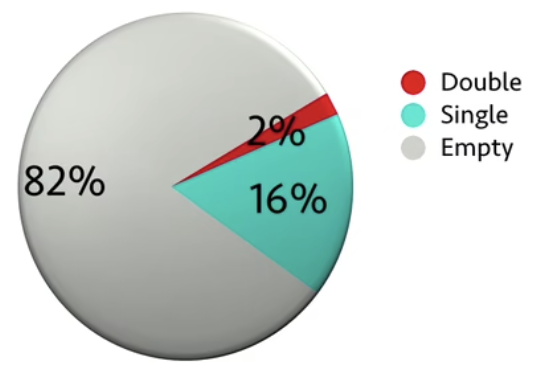
- 总结来说,在使用这种方法时,我们还是要使用比较低的细胞和磁珠浓度,来避免同一个液滴里封装了2+个细胞的情况。
10✖️ Genomics
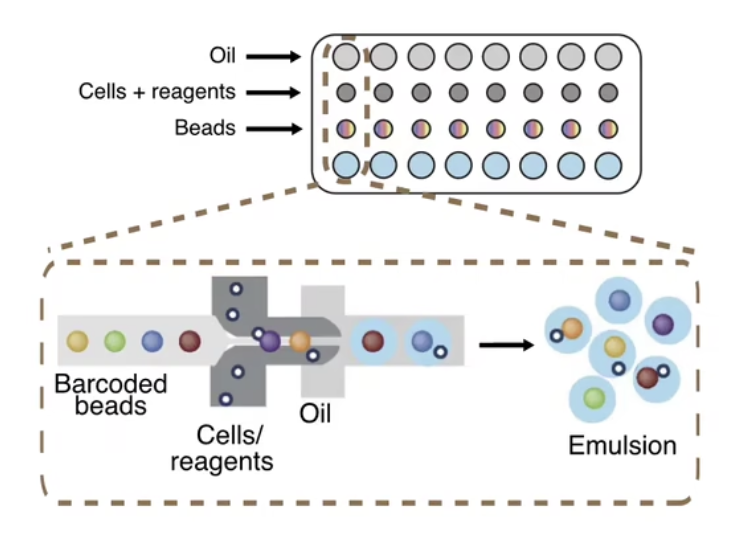
- 和上面一家公司的操作差不多,有从侧面流入的,含有barcode的磁珠,细胞和试剂首先与磁珠混合,然后油进入,产生乳液
- 在上图中,大多数磁珠都只含有一个细胞,这是因为这家公司设计了特别的微流体装置和磁珠,在这些特殊的设备作用下,90%的液滴会包含一颗磁珠,而且只含有一颗磁珠,这大大提高了效率
- 这种技术的另一个不同:在液滴中不仅可以捕获细胞,而且可以进行逆转录
- 在逆转录反应结束后,我们在单管中打破乳液,提取cDNA并且进行扩增,然后在单管中构建测序库
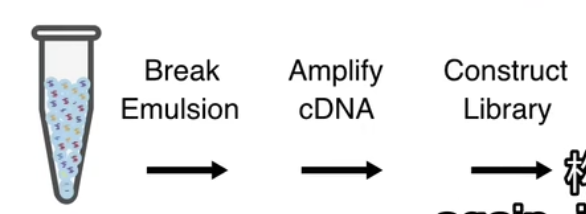
- 一个芯片的每个通道上可以测25,000个细胞,一个芯片有8个通道,所以是合计20万个细胞(理论上),而这个过程可以在一天之内完成。
微孔法microwells
- 无需液滴的平行单细胞测序
- 我们有非常小的微孔阵列,每个微孔可能只有几十到几百微米,有一些公司,比如SeqWell、Celsee、BD在做这样的产品
- 工作原理:微孔阵列中放入磁珠,这些磁珠经过特殊设计,以至于一个微孔中只能放进一颗磁珠,在放入磁珠后这个混合物会有点像一板单独隔开的磁珠,然后把细胞铺在这个板上

- 比如在上图中,有细胞的部份被标记为红色,有磁珠的区域是蓝色,细胞会落入有磁珠的孔中
- 接着,我们去裂解细胞,然后进行所有酶促反应-建库
- 这种方法的通量会更高,可以有数十万个孔,甚至是数百万个孔在一个芯片上
组合索引法combinational indexing methods
- 进行非常多的原位反应(使用微孔作为隔间)
- 进行多轮的barcoding和混合,因为多轮的barcode会生成不同的混合,最终会获得单个细胞的数据
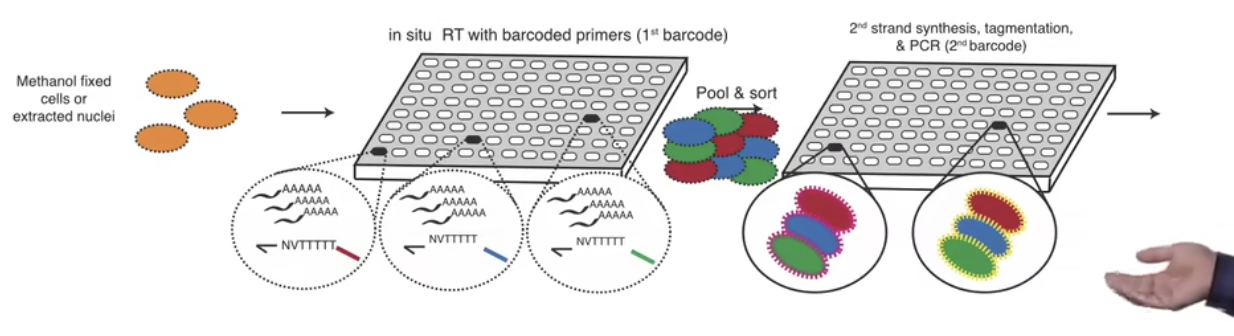
- 图片的最左边,我们会看到固定和透化(permeabilized)的细胞,以便试剂可以流入和流出细胞
- 平板的每个孔中仍然有很多个细胞,每个孔中有不同barcode的oligo-dT引物,在第一轮逆转录PCR之后,你会筛选出图中的三个孔中的细胞(分别用红色、绿色和蓝色标记),我们将这些细胞混合在一起,或者将它们打乱,然后随机分配,加到一组新的96孔板中
- 第二次混合时,我们会将逆转录出的cDNA提取出,用特殊的引物去添加第二个barcode,进行PCR
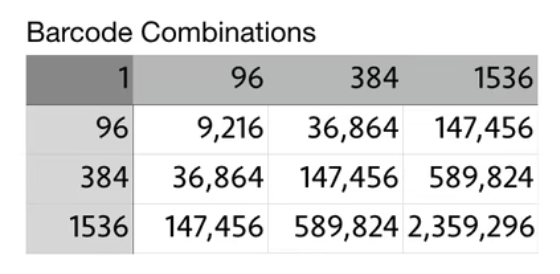
- 对于两轮反应:我们有96✖️96,大概10000种选择,比如说如果我们要测序1000个细胞,那我们就不太可能遇到两个细胞出现在一个孔中的情况
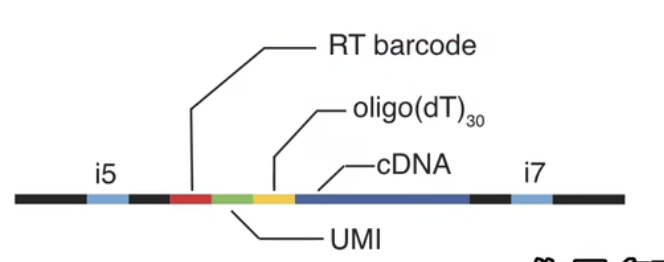
- 最终的产物如上图所示
- 这种技术的魅力:非常高的通量,可以有接近15万个细胞(而且在现实研究中,使用多达384孔板实际上并不常见
其他单细胞测序法(非RNA)
利用CITE-seq技术量化蛋白质
- 使用抗体与目标蛋白结合(这个抗体与用于流式细胞学的抗体类似),这个抗体不会连着一个荧光基团(用于流式读取),或是同位素(例如CyTOF),而是结合在DNA barcode上
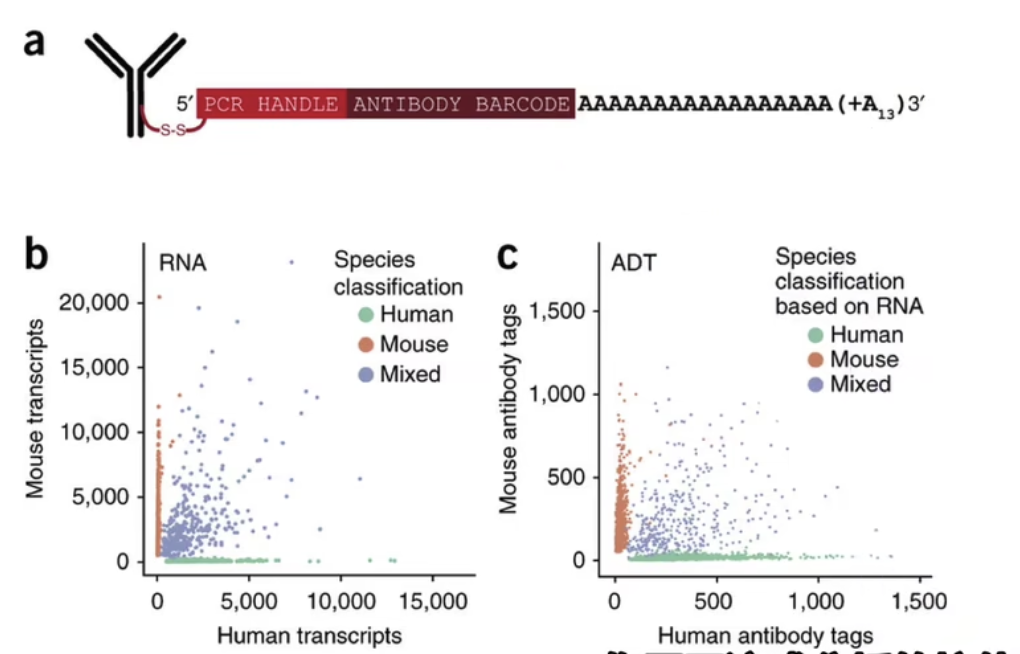
- 就像上图a所示,我们可以看到抗体的C段连着一个用于启示PCR反应的PCR handle,然后连着一段用于鉴定这个抗体信息的barcode,最后是poly-A尾(就像mRNA上的poly-A尾一样,允许这段DNA序列被单细胞测序方法捕获)
- 有大概100种左右的抗体可以和单细胞测序结合使用,很厉害的一点是我们不仅可以获得蛋白质丰度(对于有抗体的蛋白质),同时还可以获得这个细胞转录组的情况
- 也就是说,这种测序方法本质是一种多组学方法,可以同时分析蛋白质和mRNA
技术的局限:上样浓度loading concentration
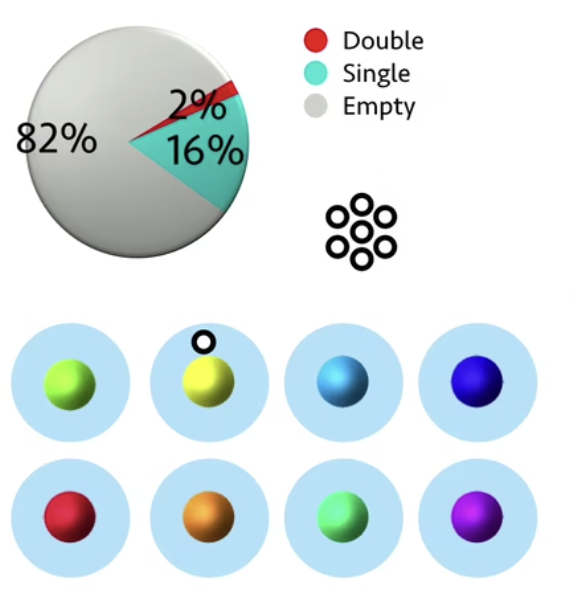
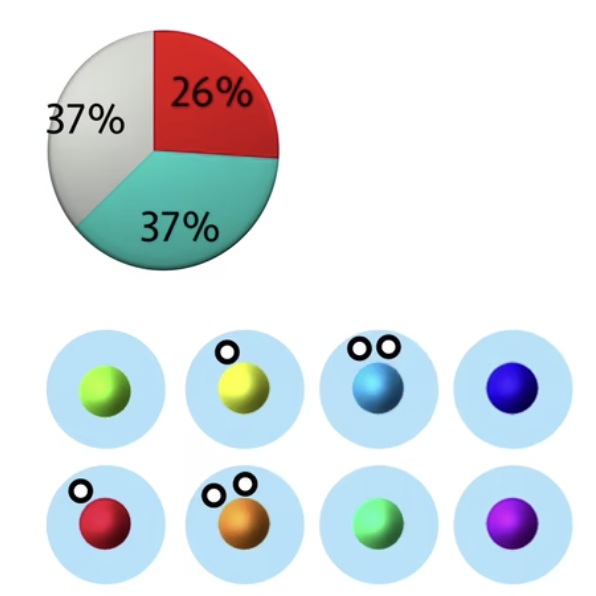
- 和上面我们说过的内容一样,我们希望使用液滴法的时候,磁珠和细胞的浓度都不希望太高,以避免一个液滴中出现两个以上细胞(和/或磁珠)
- 我们确实可以使用一些计算的方法,来将一液滴复数细胞(和/或磁珠)从我们的数据分析中删除
demuxlet
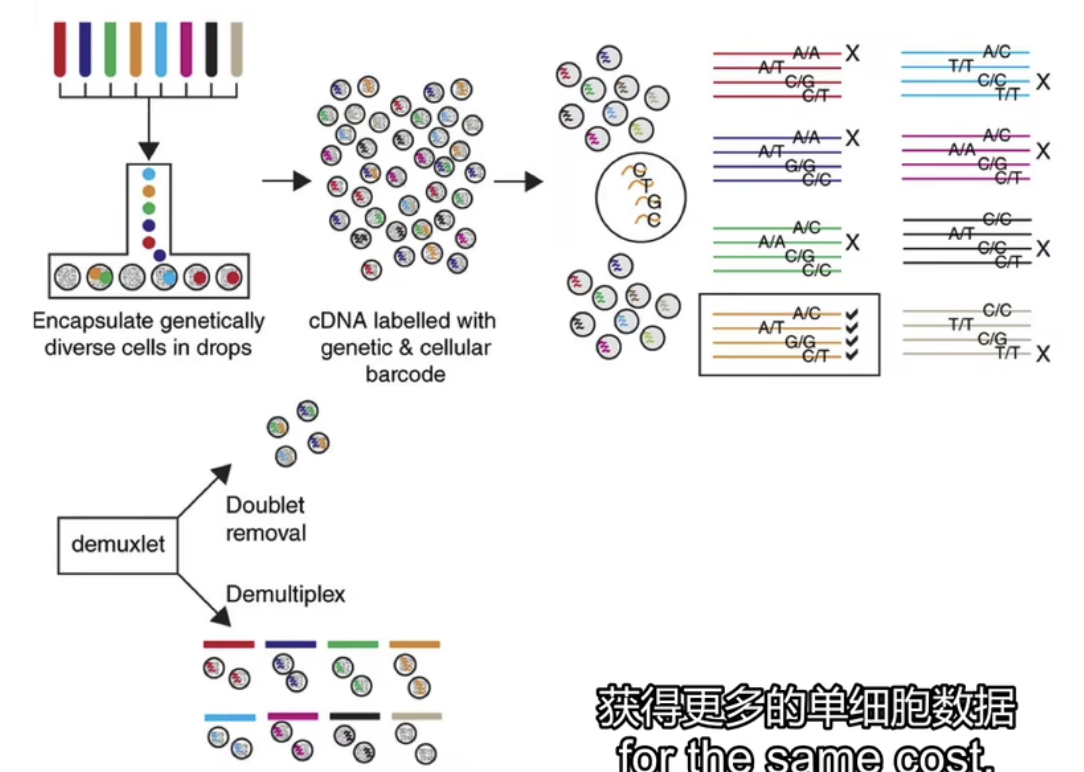
- demuxlet技术会使用双重barcode,在细胞上有一条barcode(多样本上样进一个芯片,然后在样本处理阶段引入第一条barcode,即细胞barcode),然后在测序阶段引入第二条barcode(磁珠barcode),然后进行测序
- 在测序的阶段,如果我们发现了一个液滴中含有两个细胞(在这种情况下,一个液滴中的两个细胞通常是不同来源的,也就是带有不同的细胞barcode),那这样我们就可以在数据分析阶段,通过相同的磁珠barcode+不同的细胞barcode,来剔除这部份数据
非遗传的的multiplexing策略
- non-genetic multiplexing strategies
- multiplexing:笔者的理解是多样本单次检测,节省成本的同时提高通量
基于抗体的multiplexing策略
- demuxlet的方法存在局限性,具体来说,我们会使用细胞barcode来区分来源不同细胞的基因组,但是,如果每个细胞的基因组都是一致的,这种方法就不再有效了
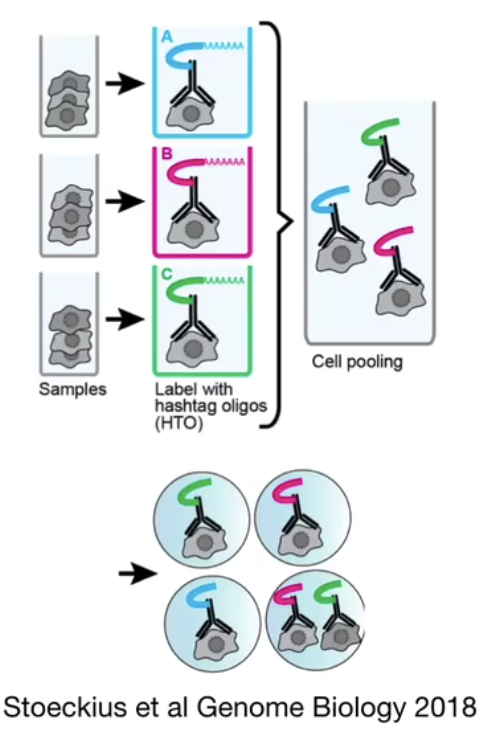
- 这种技术是由开发了前面说过的CITE-seq技术的人开发出来的
- 思路是通过使用与细胞表面某种通用抗原结合的抗体,将一大份抗体分成几等分,然后每个部份给对应的一个barcode,然后每个barcode抗体加入不同来源的样本,然后上机测序。
- 这种方法也能解决单一液滴中复数细胞的问题,如果你在一个液滴中检测到了复数barcode的样品,你就会知道你的样品混入了两个不同来源的细胞,进而将它们剔除
基于杂合DNA的multiplexing策略
- 使用嵌入细胞膜的脂化DNA
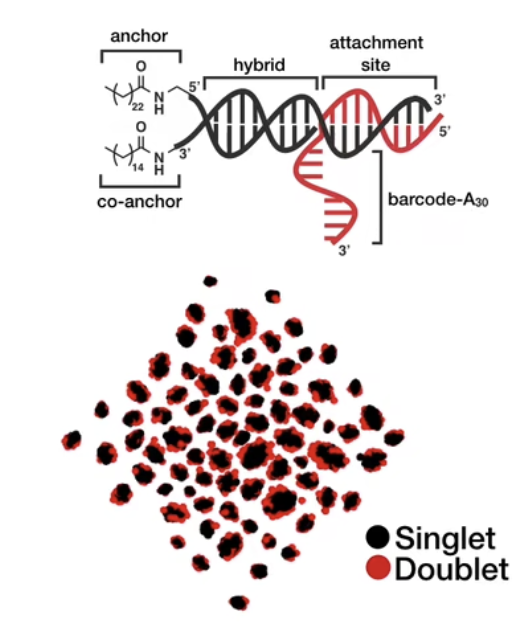
- 在每个样本池中加入不同的barcode,然后将它们汇集在一起
- 在上图的例子中,我们可以标记96种不同的样本,将它们汇集在一起,同时还能区分一个液滴内含有复数中细胞的情况。
总结
各种各样的单细胞测序方法
- DNA、RNA、蛋白质
- 染色质可及性
- ……
- 每隔几周就会有新的方法
单细胞测序会产生高维度数据
- 特别是在研究RNA时,我们实际上在探究一个细胞内所有2万种不同基因的数量或表达水平
单细胞测序技术的缺点
- 相比于流式细胞学或质谱流式细胞学
- 产量较低
- 价格较高
不基于单细胞的测序方法
- seqFISH、MERFISH、CODEX
- 基于显微镜
- 更低的产量,但是会带有空间信息



