
Visium与Xenium产品的比较
概述
- Visium产品非常适合用于探索,比如各位老师还没有感兴趣的biomarker或者细胞类型
- 对于想要了解组织中发生了什么变化的各位老师来说,Visium HD是非常合适的产品
- Xenium适合各位老师已经有了感兴趣的biomarker(至少是一部份)和细胞类型,因为Xenium可以根据各位老师感兴趣的biomarker进行panel的定制服务
通过10x的预印本文章去学习各种技术如何交相呼应(orchestrate)
- 各位老师可以通过探索10x发布的预印本文章,去了解各种技术(单细胞,空间与原位)是如何相互配合来回答生物学问题和探究样品的生物学特征的
Visium HD is like the start of the show, while Chromium and Xenium are helping out and validating a lot of what Visium HD is revealing

补充:各位老师可以使用10x single-cell FLEX产品去处理FFPE样本,来获得单细胞数据
如果各位老师对免疫相关的数据有兴趣,可以使用10x提供的5’ single cell immune profile来获得TCR和/或BCR的数据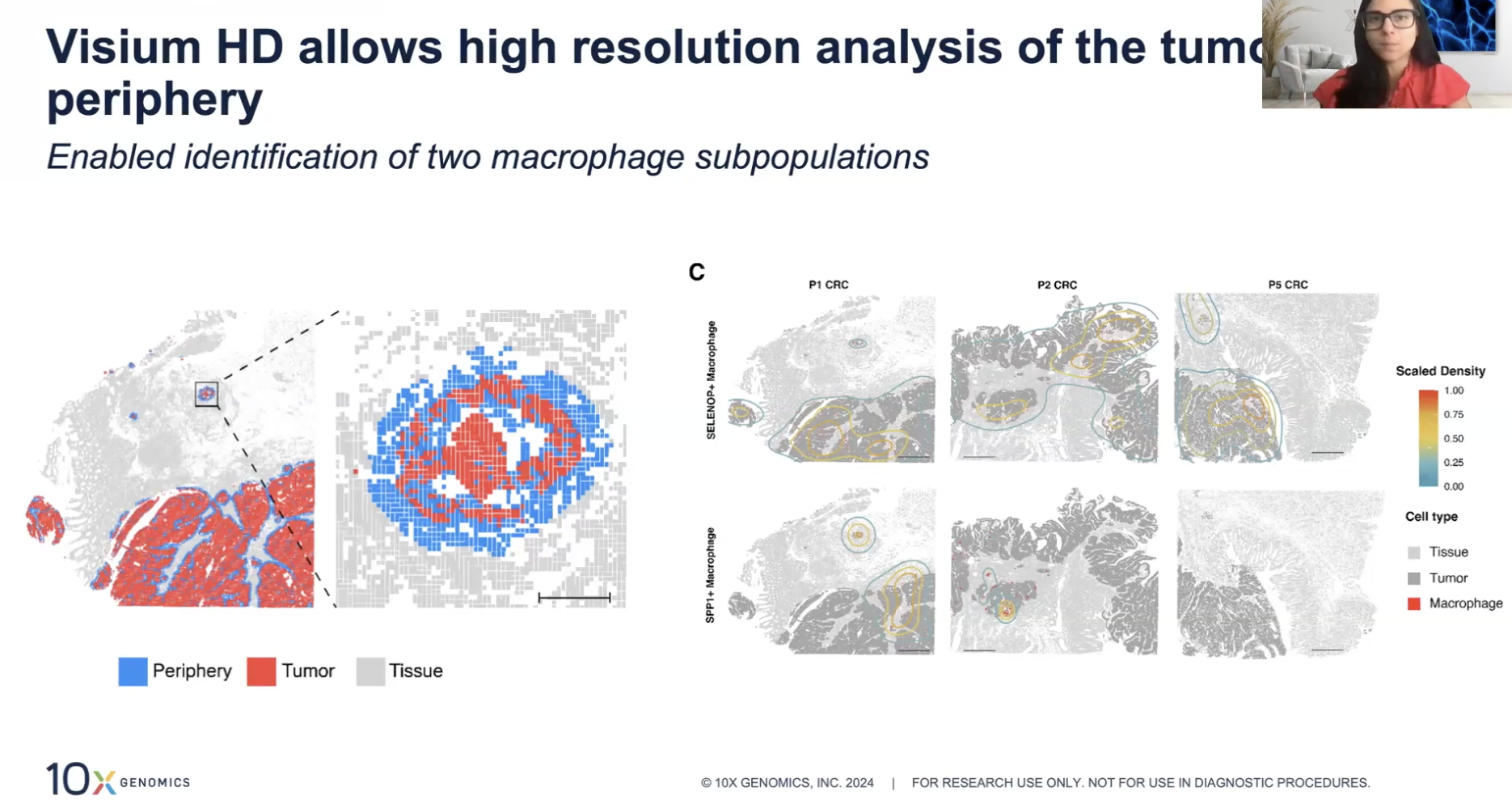
- Visium HD技术可以很好地揭示肿瘤的中心和外围区域,并且发现这个区域有2种巨噬细胞亚群的富集,这两种巨噬细胞在肿瘤外围区域中的空间定植情况也是不一样的。得出的解释是两种不同的巨噬细胞介导的免疫互作可能对肿瘤组织的异质性做出了贡献
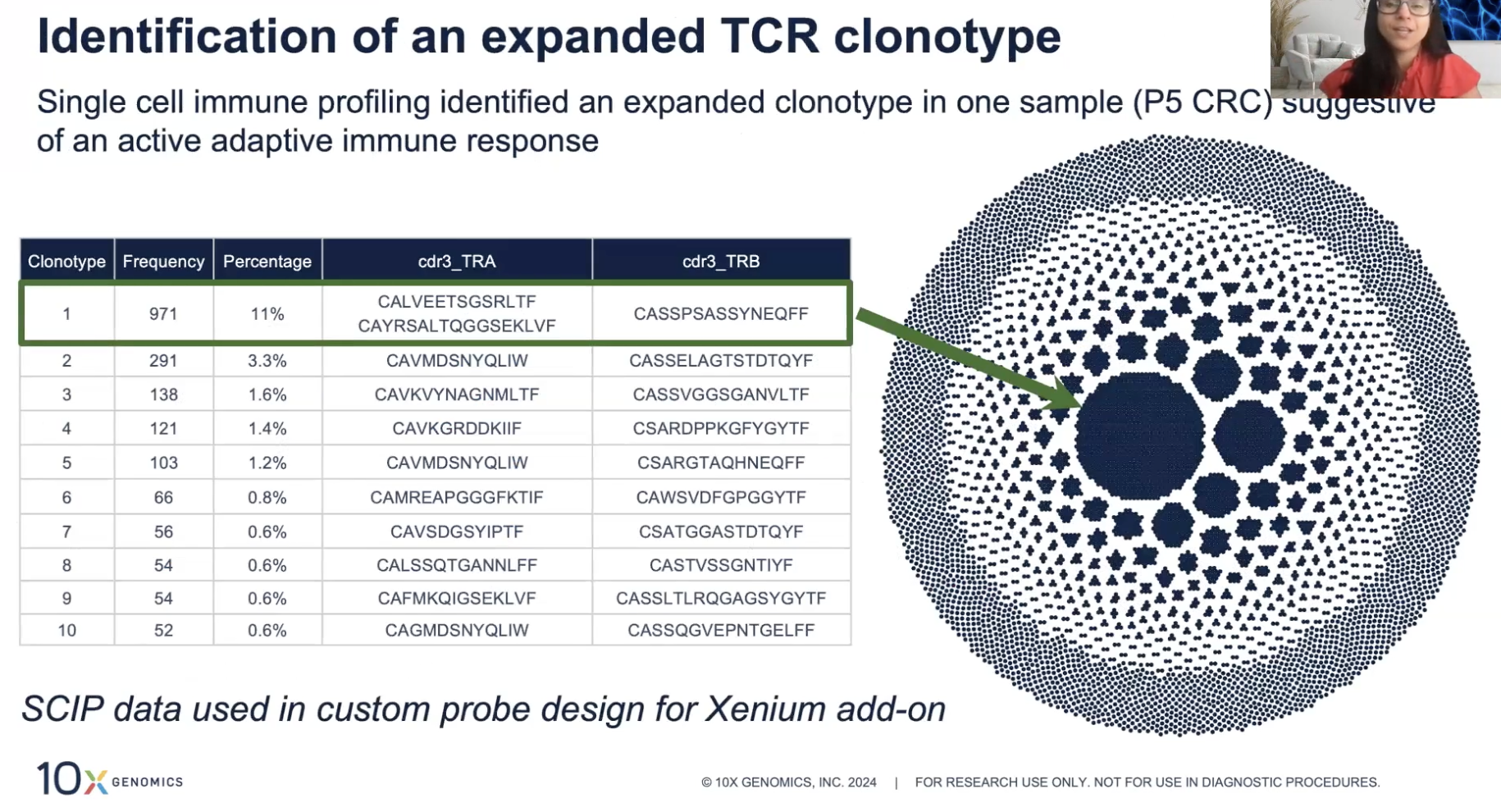
- 文章中的另一发现是5’-single cell assay和TCR贡献的,这种技术可以探究肿瘤免疫组织中参与和/或扩增的细胞亚群,也就是所谓的”single cell immune profiling”,上图(蜂巢图)可以在Loupe Browser中产生,图中越大的球表示对应的细胞克隆占总细胞克隆的比例越大
- 可以根据5’-single cell assay和TCR测序的结果,去生成Xenium Panel使用的探针,然后探究这些细胞在空间中的分布情况
- 文章中Xenium部分的结果见下图
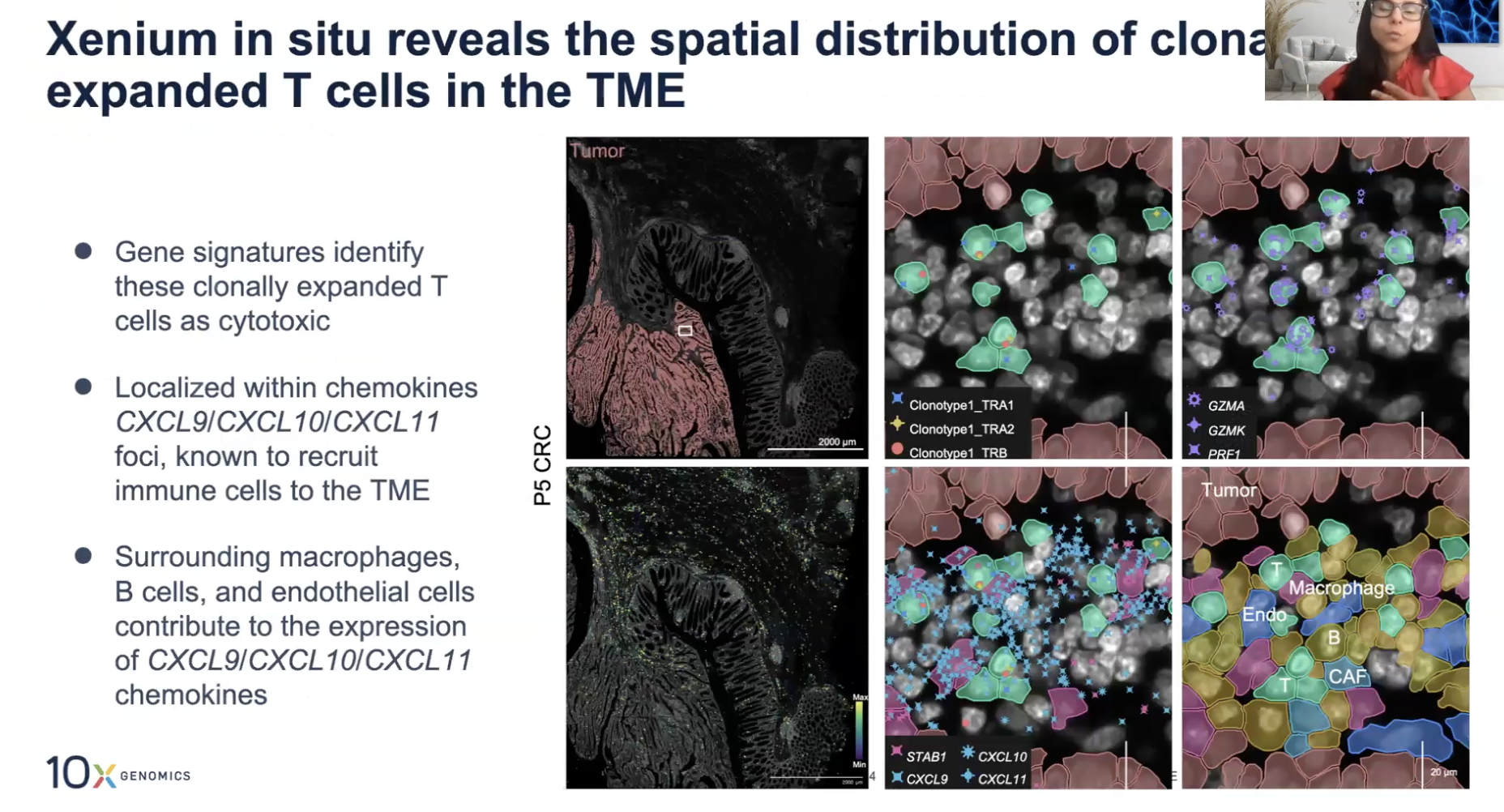
- 使用了针对巨噬细胞的基因(在Visium平台上被发现有表达差异)去设计Xenium Panel,也观察了5’-single cell assay中发现的各种细胞类型在空间上的定位情况
- 发现5’-和TCR的结果中扩增的T细胞表现出细胞毒性的表型,这些细胞与趋化因子CXCL9-11共定位,这些趋化因子负责招募免疫细胞进入免疫微环境;可以看到cytotoxic T cells与B细胞,巨噬细胞,其他内皮细胞等趋化因子邻域共定位。
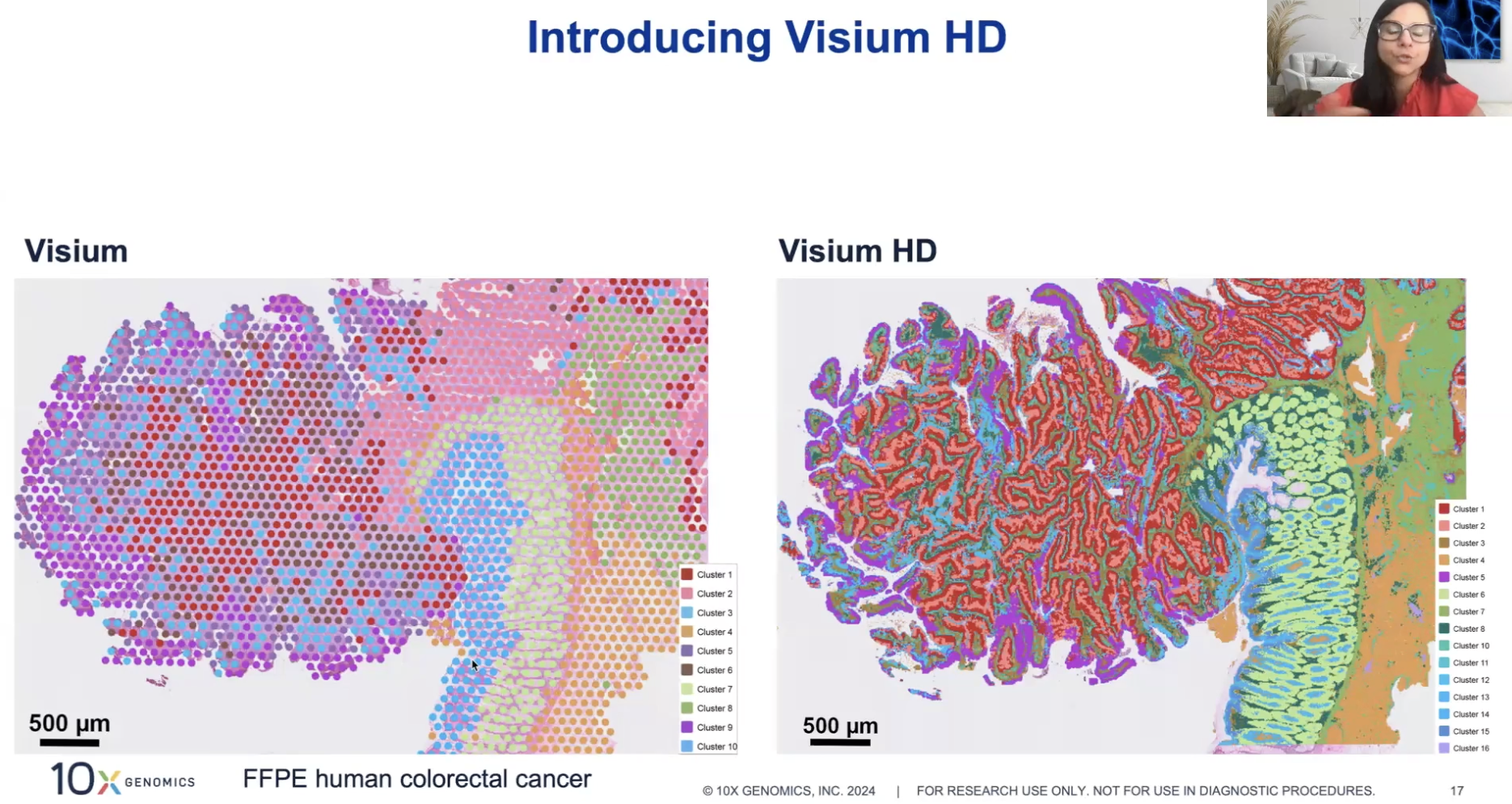
Visium HD产品的介绍
- 前所未有的空间探索——全转录组基因表达谱与形态学分析(HE或免疫荧光,在Visium分析钱)
- 单细胞尺度的分辨率——探针捕获区域是连续的2✖️2微米,这个分辨率大于绝大多数哺乳细胞的体积
- Visium HD运行在CytAssist平台上,并且兼容FFPE样本

- CytAssist平台相比于手动操作能获得更高质量的数据

- CytAssist平台甚至允许跨大洲运输的样品进行处理
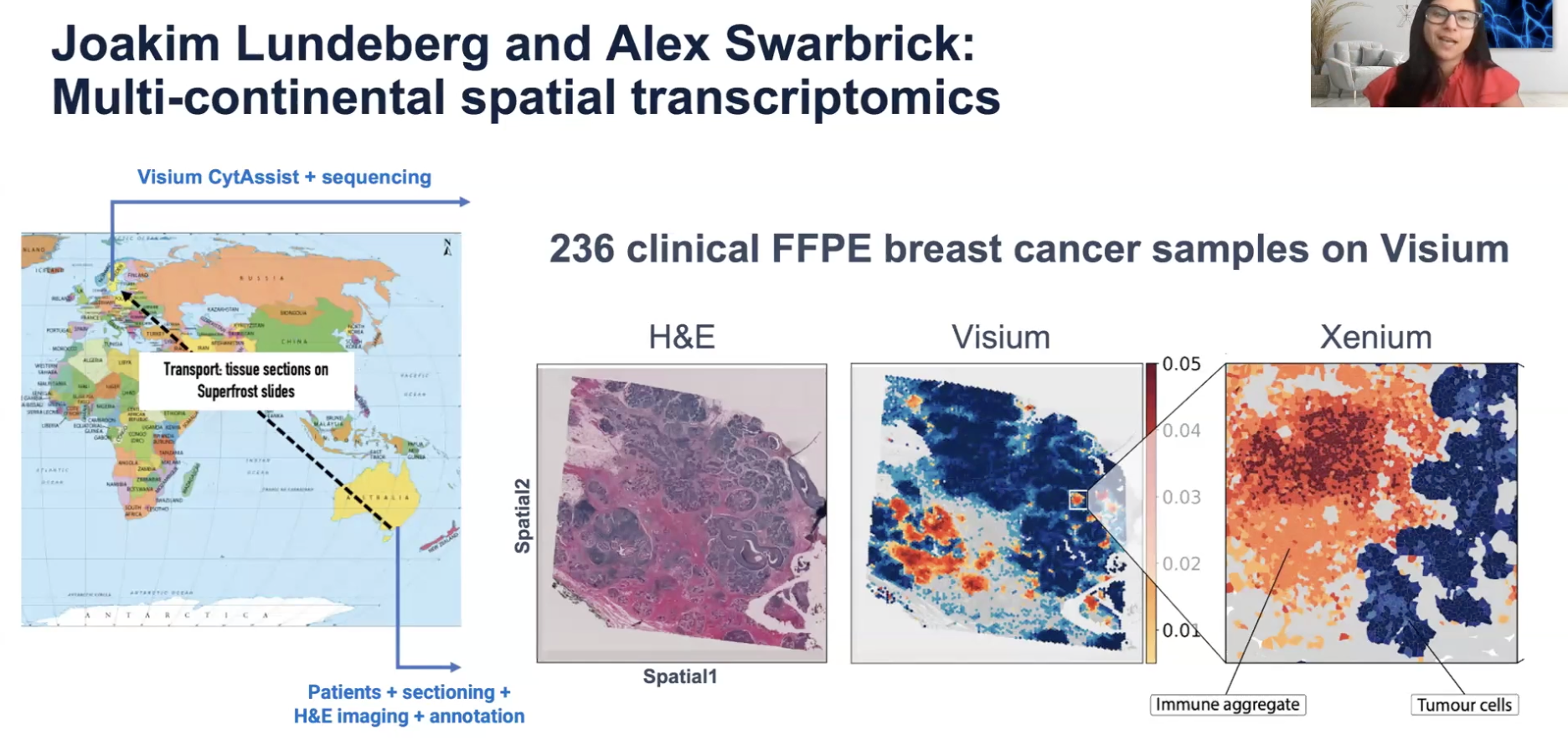
- 对于含有外源序列的样本(例如被新冠病毒感染的人肺样本),可以针对样本特异性地设计探针
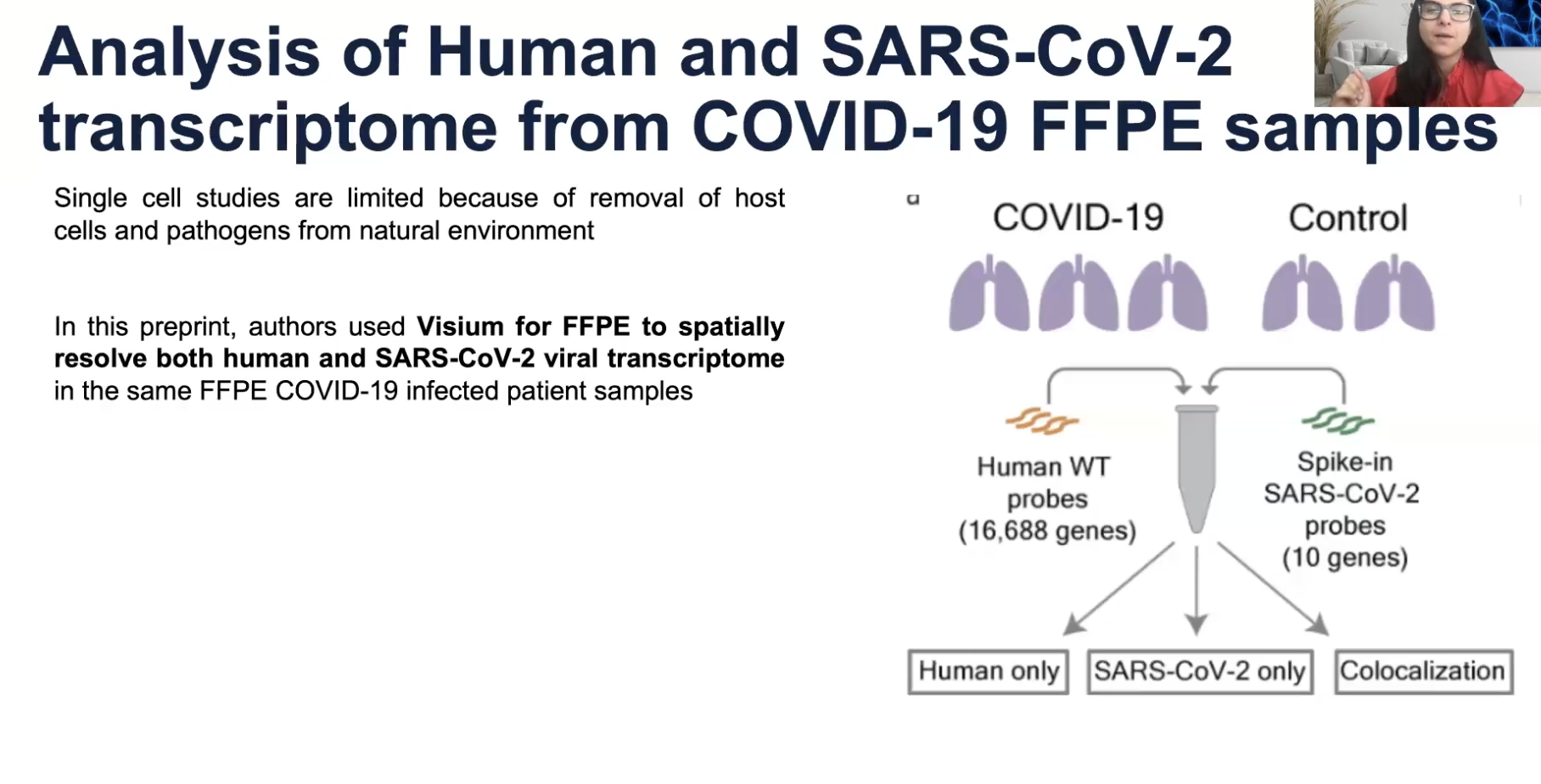
- 在上面的研究中,研究者针对新冠患者的FFPE样本选择了Visium的人全转录组panel的基础上,还增加了对于COVID-19的全转录组探针
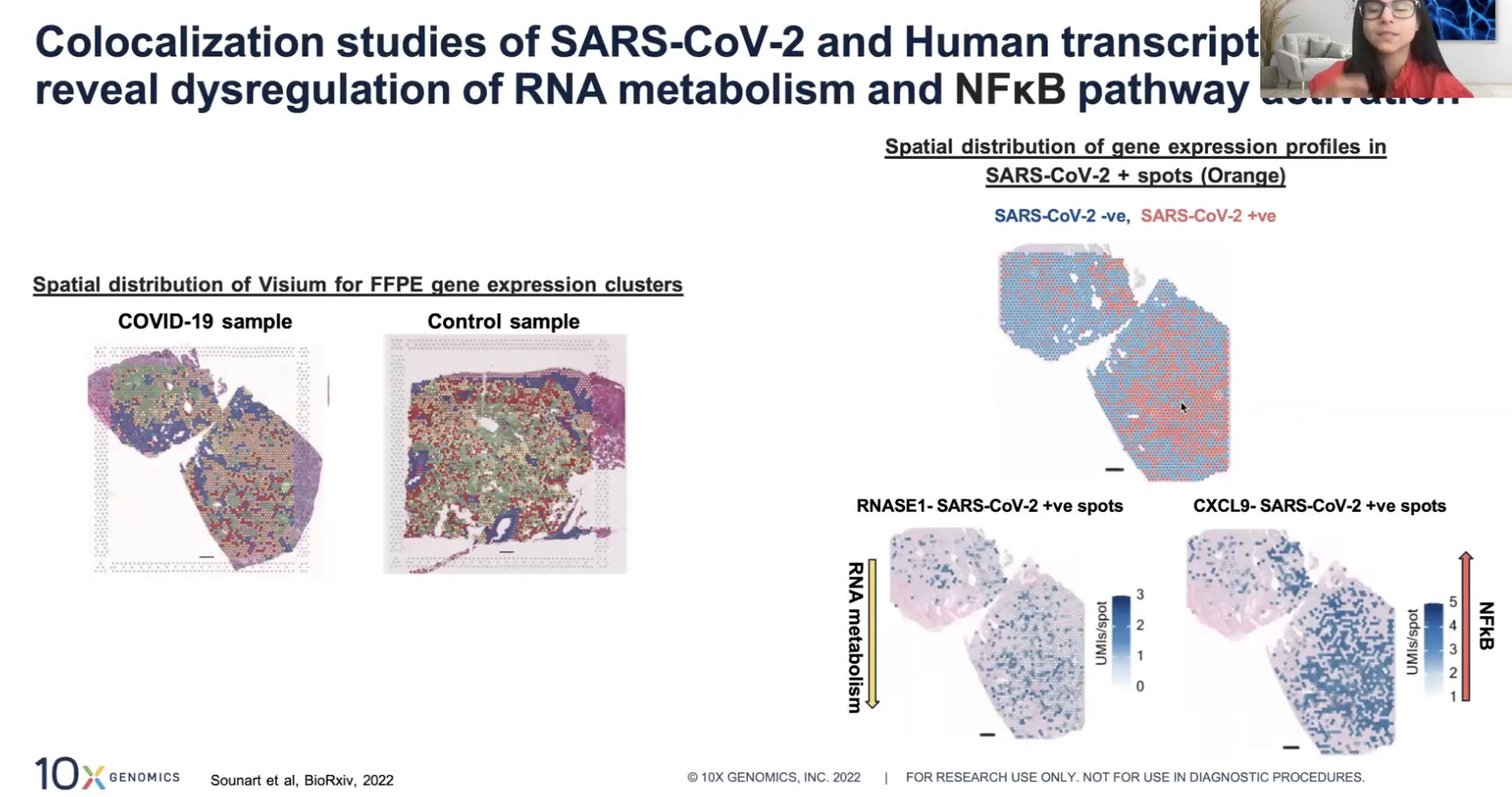
- 可以看上图右上部分,蓝色的区域没有检测到COVID-19,红色的区域有,因此可以发现在被COVID-19感染了的区域,RNA代谢水平下降,NF-κB信号通路表达增加
- 上图中使用的Visium V2每个孔spot的尺寸是55μm,因此还没有单细胞分辨率,但是Visium HD每个bin的尺寸是2✖️2μm,即便分析中会合并成8✖️8μm去分析,也能实现单细胞尺度的解析度(见下图)
Visium HD的工作流简单介绍
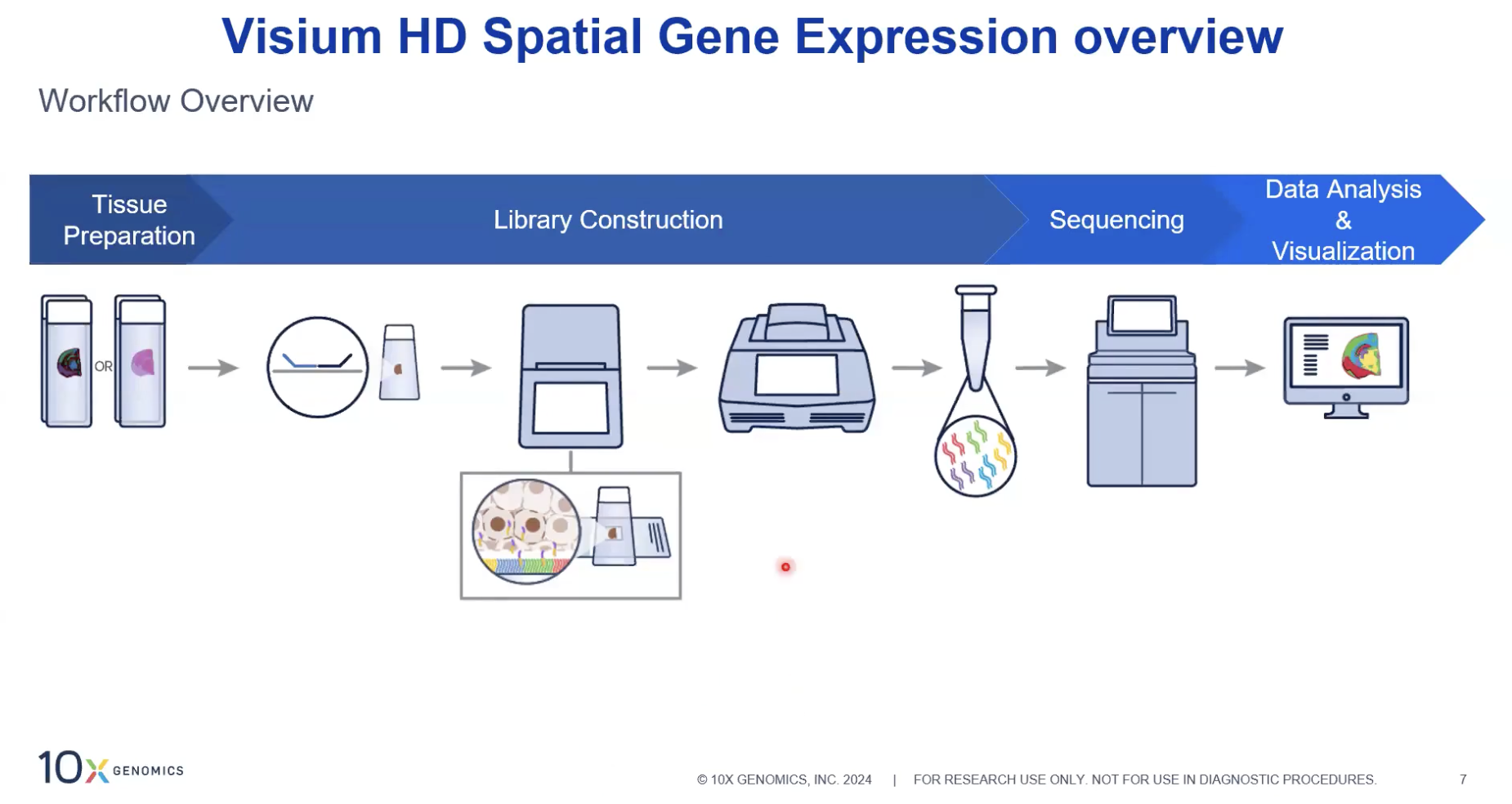
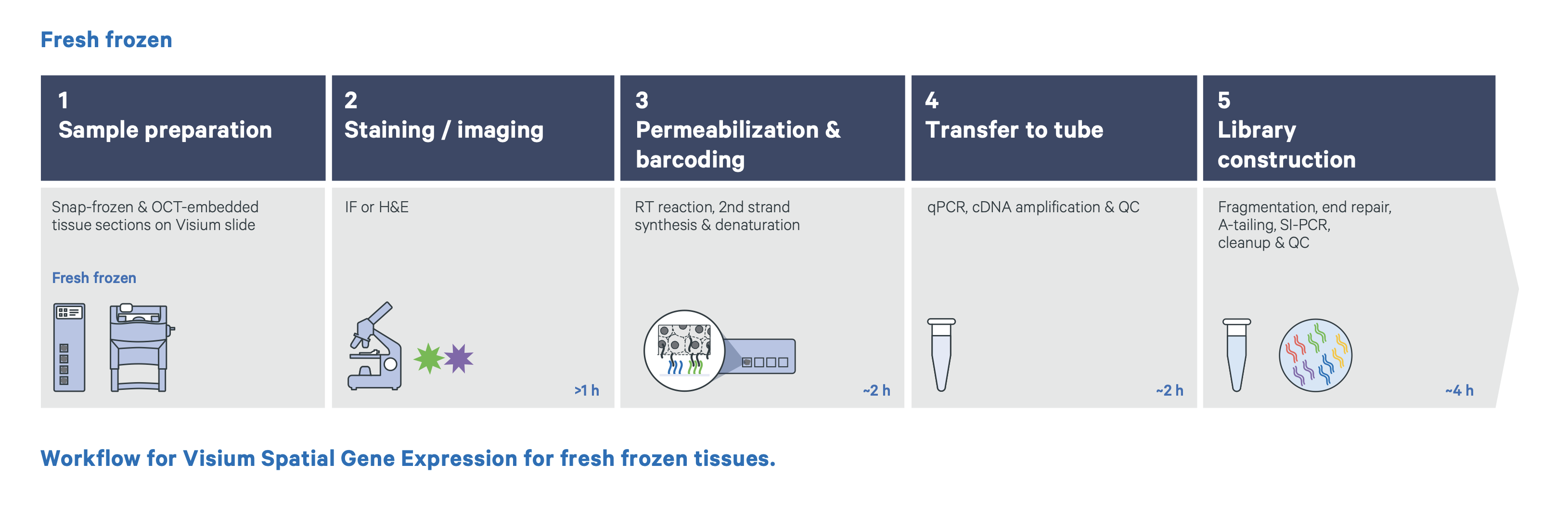

- 可以把切片放在普通的载玻片上,对于已经切好的切片,各位老师也可以将其转移到CytAssist上(不是这一步)
- 进行染色(HE或免疫荧光,取决于老师对什么感兴趣)
- 可以多次切片,直到选中了我们想要的组织
- 染色后,老师们可以将组织进行对应的处理(FF透化,FFPE解交联),然后与探针共孵育
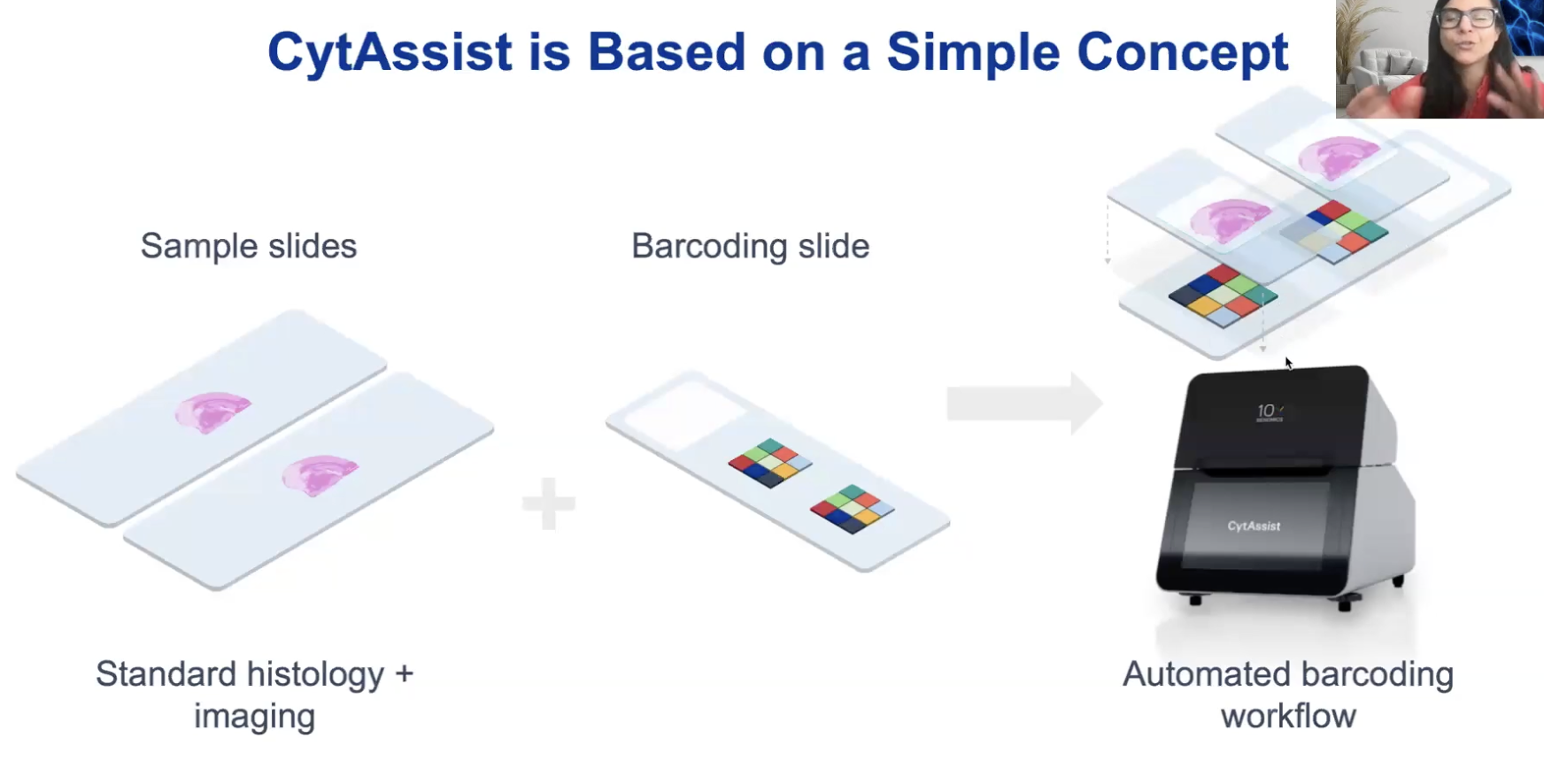
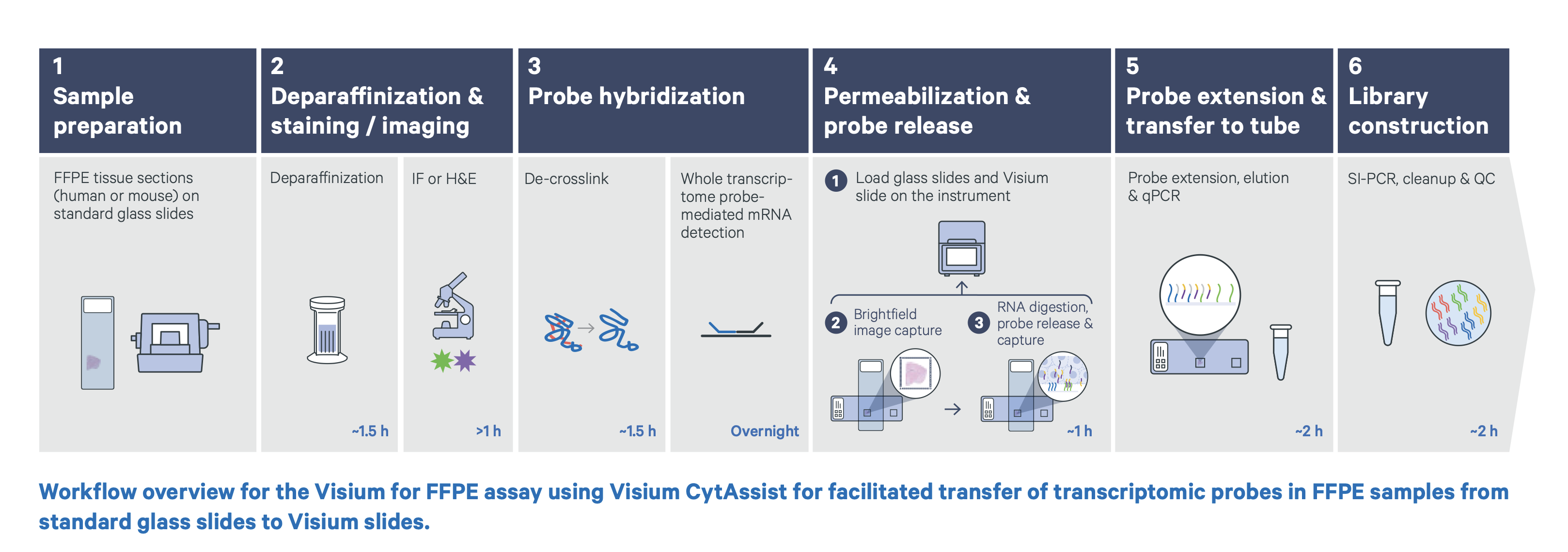
- 之后将样品的切片与携带有Visium Barcode的切片一起放置在CytAssist上即可
- 在CytAssist上,机器会拍下一张高清图片(以确定捕获区相对于我们组织的位置),然后使上一步在组织上的RNA上的探针被洗脱下来,释放到Visium芯片上
- 每次运行机器(以下称为循环cycle)可以进行两个反应,因为一个Visium Barcode切片上有两个捕获区,每个捕获区内可以放复数个组织
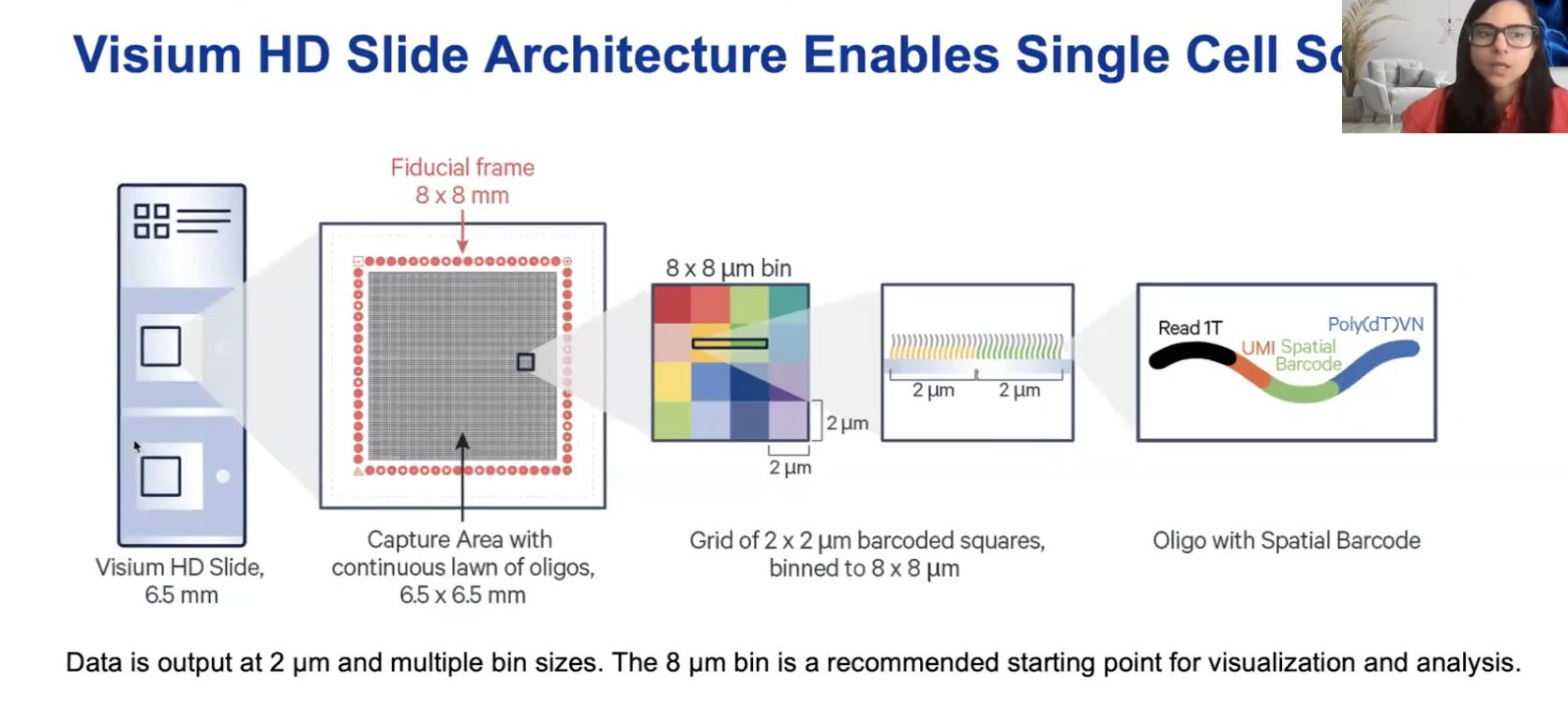
- 两个捕获区,每个捕获区的大小是6.5✖️6.5mm,每个捕获区内是由连续的2✖️2μm的小格子构成的,在数据分析中,各位老师可以把数个格子整合在一起,形成4✖️4μm,6✖️6μm或8✖️8μm(推荐,适用于绝大多数哺乳动物样本)的矩阵(bin),来简化数据分析的需要,尽管各位老师仍然可以获取到2✖️2μm的样本数据,甚至可以拿到16✖️16μm的bin size
对于探针的介绍

- Read 1T 是来自 Illumina TruSeq 测序技术中的一部分,这一部分的主要作用是标记探针序列的开始位置,确保在后续的测序过程中能够正确地识别该探针的开始位置,从而准确地进行后续数据分析


Visium HD兼容多种样本类型
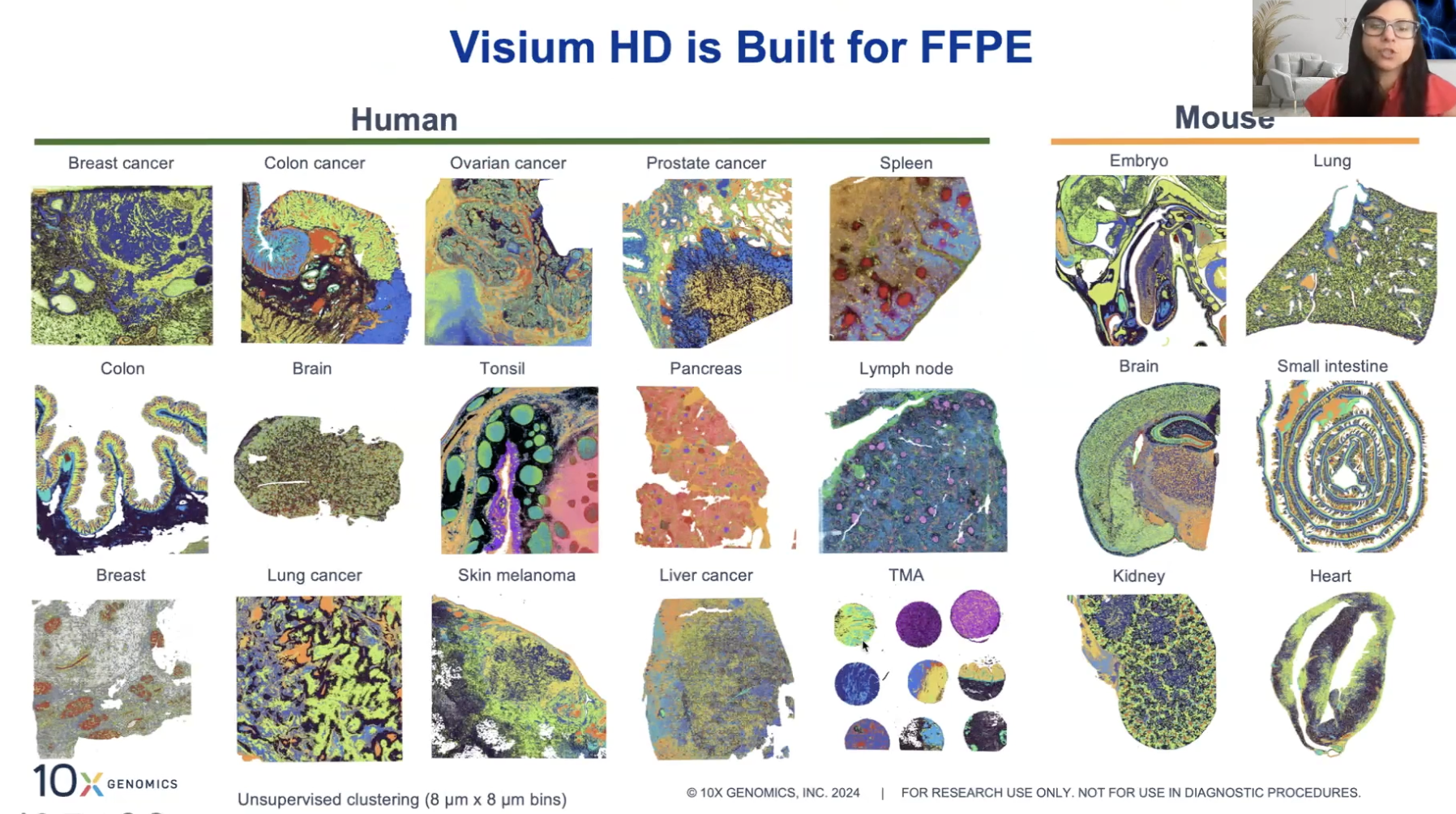
- 比如FFPE和TMA(组织微阵列Tissue Microarray)
Visium HD的结果展示
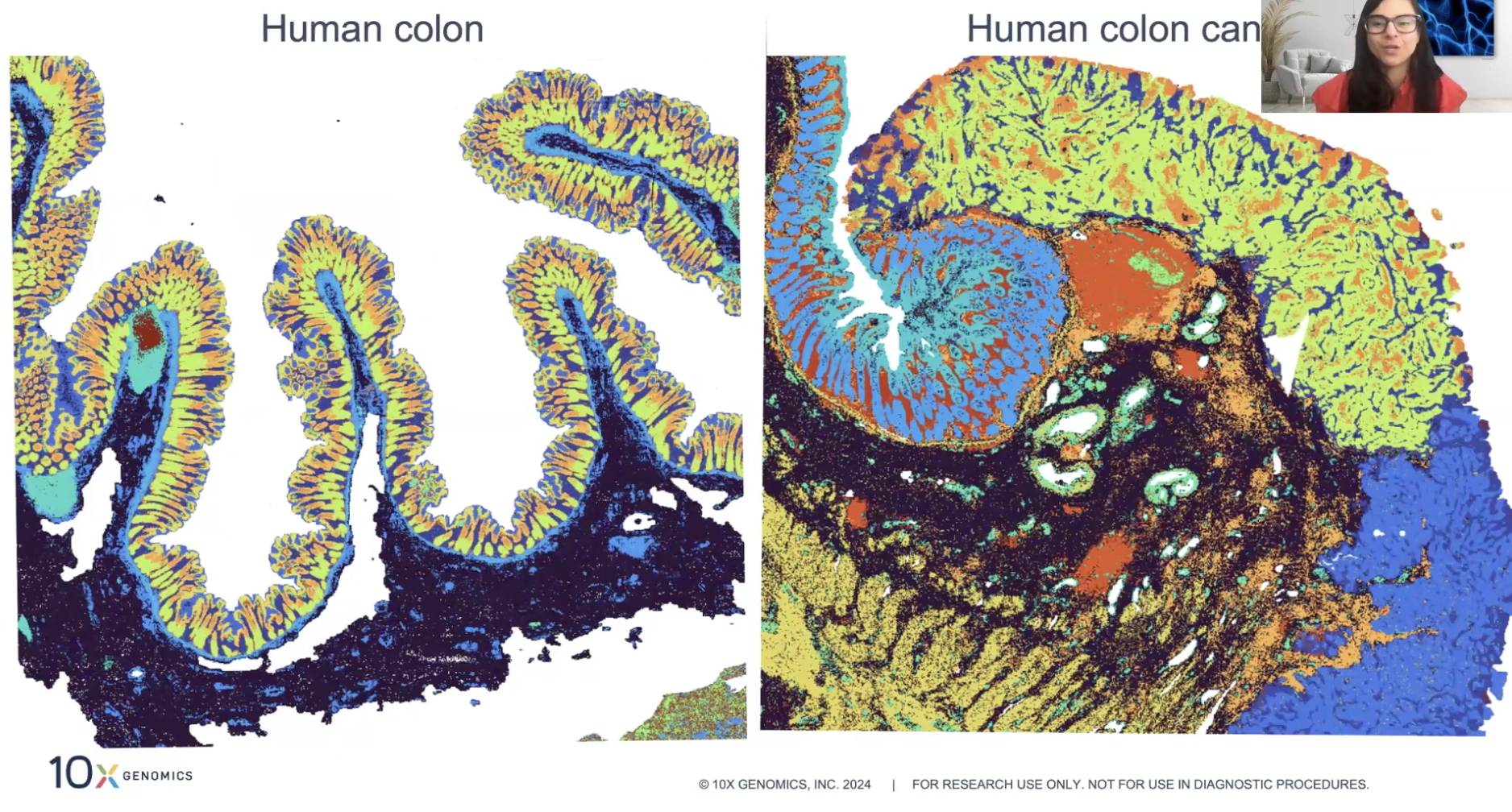
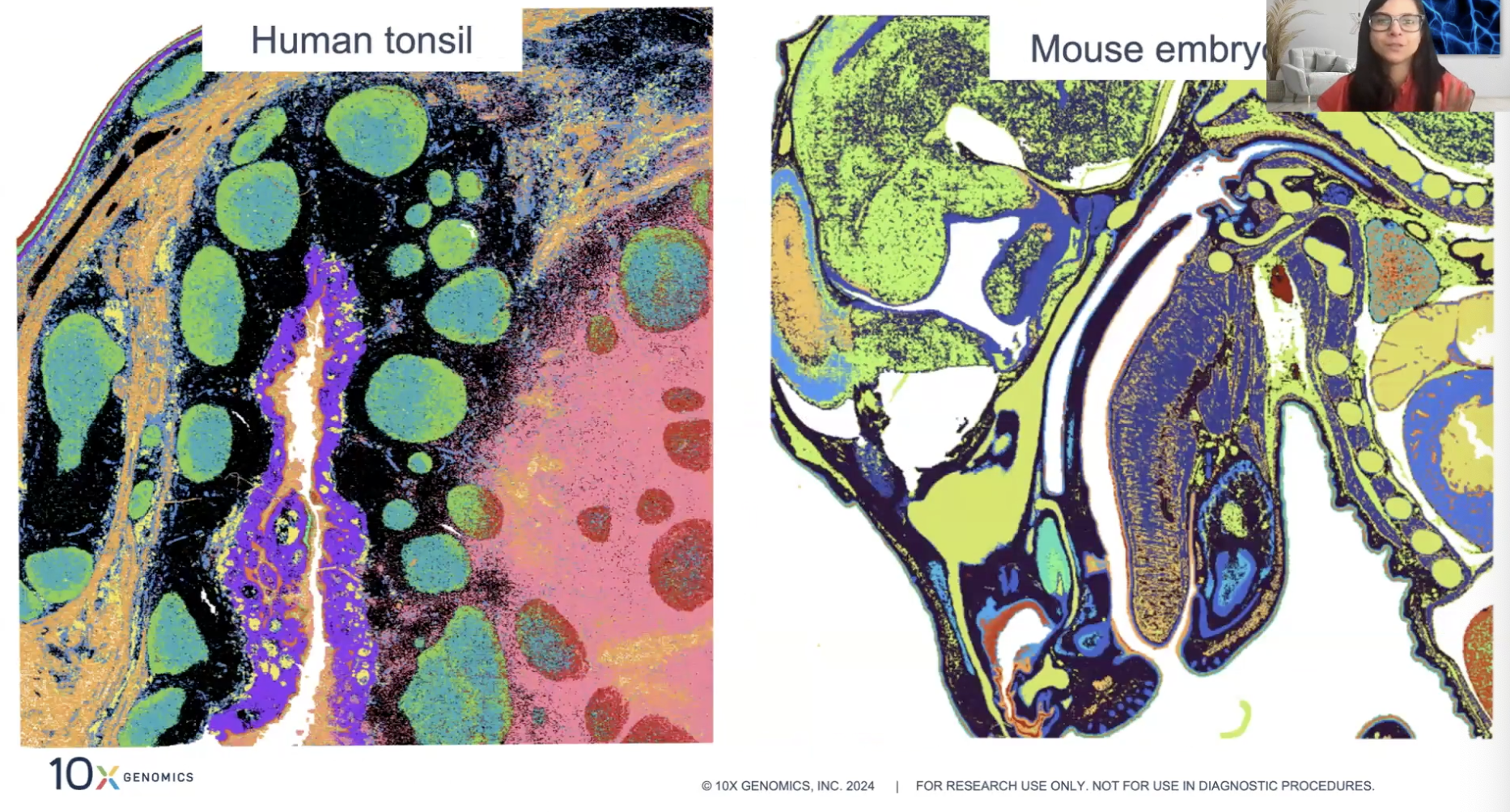
Visium HD同时实现高分辨率和高特异性
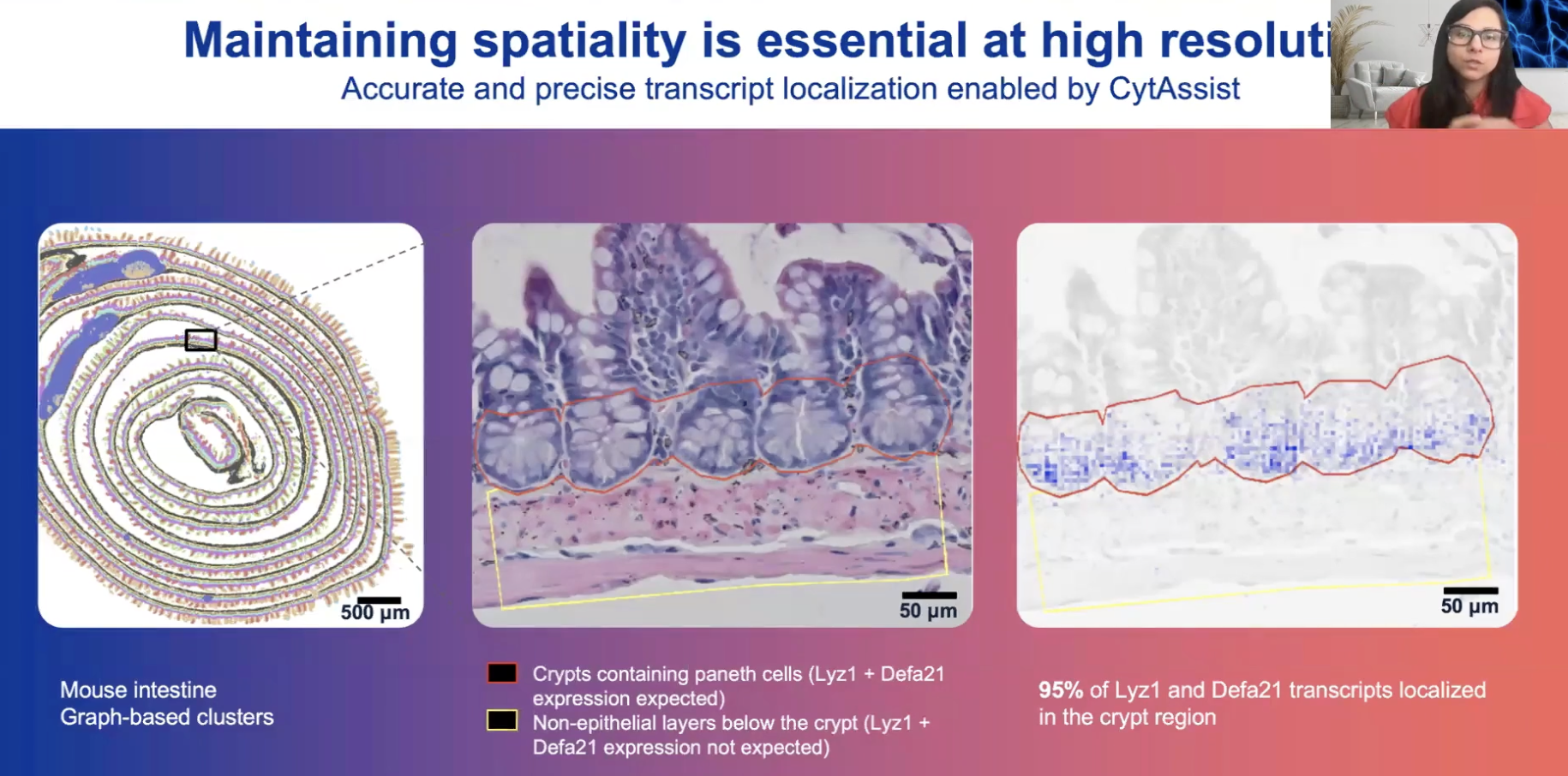
- Visium HD数据进行Graph-based clustering产生的细胞clustering结果与形态学一致
- 例如上图中间是小鼠肠道上皮的样本,样本中包含两类细胞,各自有不同的转录组特征,而右图展示的是Visium HD数据进行Graph-based clustering的结果,可以看到Visium HD捕获到的转录本95%都落在了与形态学相符的区域内
Visium HD在不同处理的样本中都能获得高质量数据
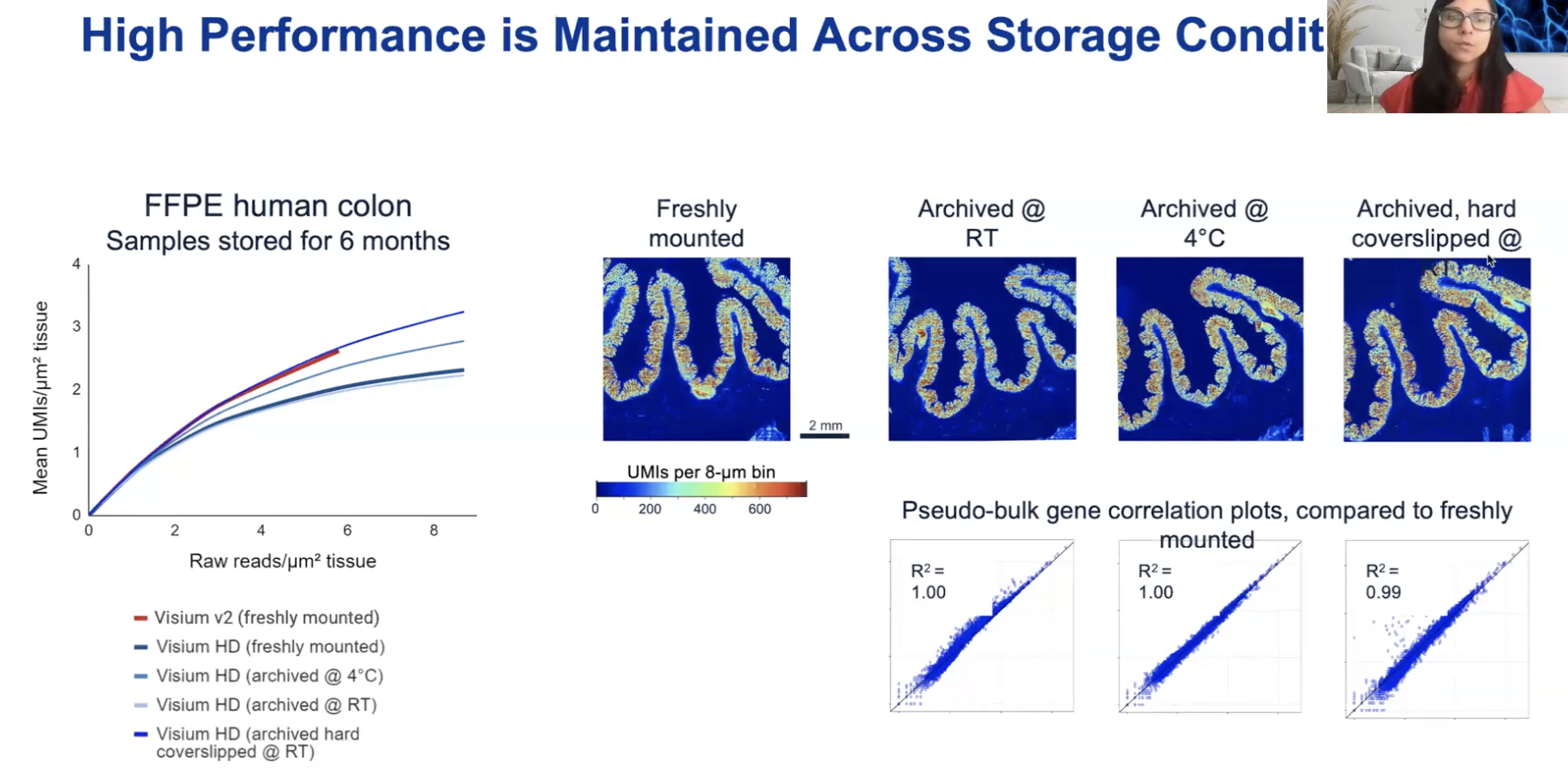
- 对于人结肠的FFPE样本,可以看到在不同的处理条件下(室温、4度、硬玻片覆盖)都能捕捉到足够数量的转录本,也就是数据的质量足够高
- Visium HD能保存到这么高质量数据的其中一个原因是使用了CytAssist机器
结合单细胞数据对HD数据进行反卷积
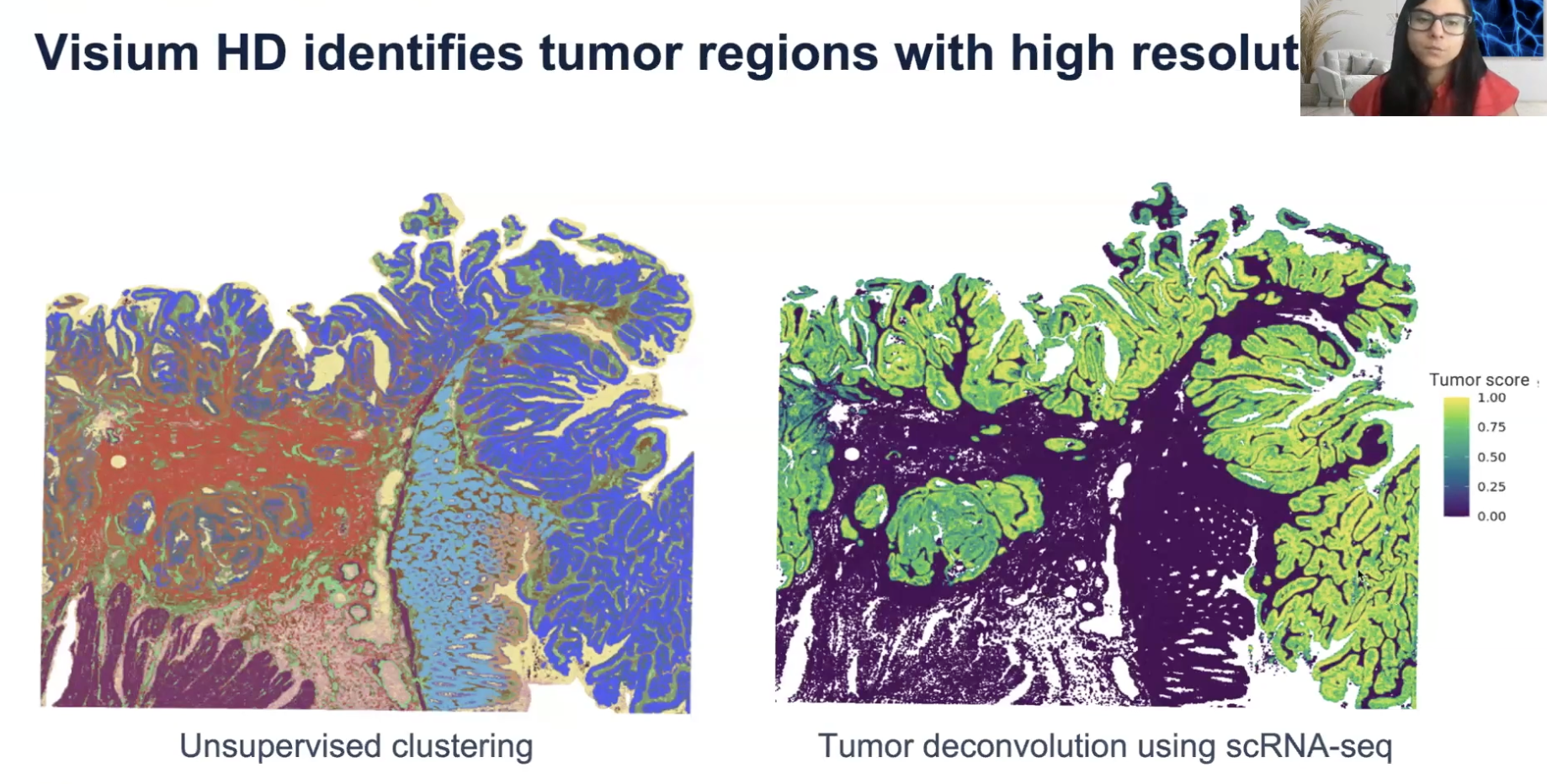
- 左图是Visium HD数据进行无监督聚类分析的结果,深蓝色的区域为肿瘤细胞
- 右图是结合了单细胞数据对HD结果进行反卷积分析,设置了一个“肿瘤分数”,分数越高越可能是肿瘤,可以看到绿色的区域内是肿瘤细胞
- 启示是引入single-cell的数据对Visium HD数据进行反卷积分析,可以得到更深入的洞察
- 另一个使用单细胞数据去辅助Visium HD数据进行分析的示例
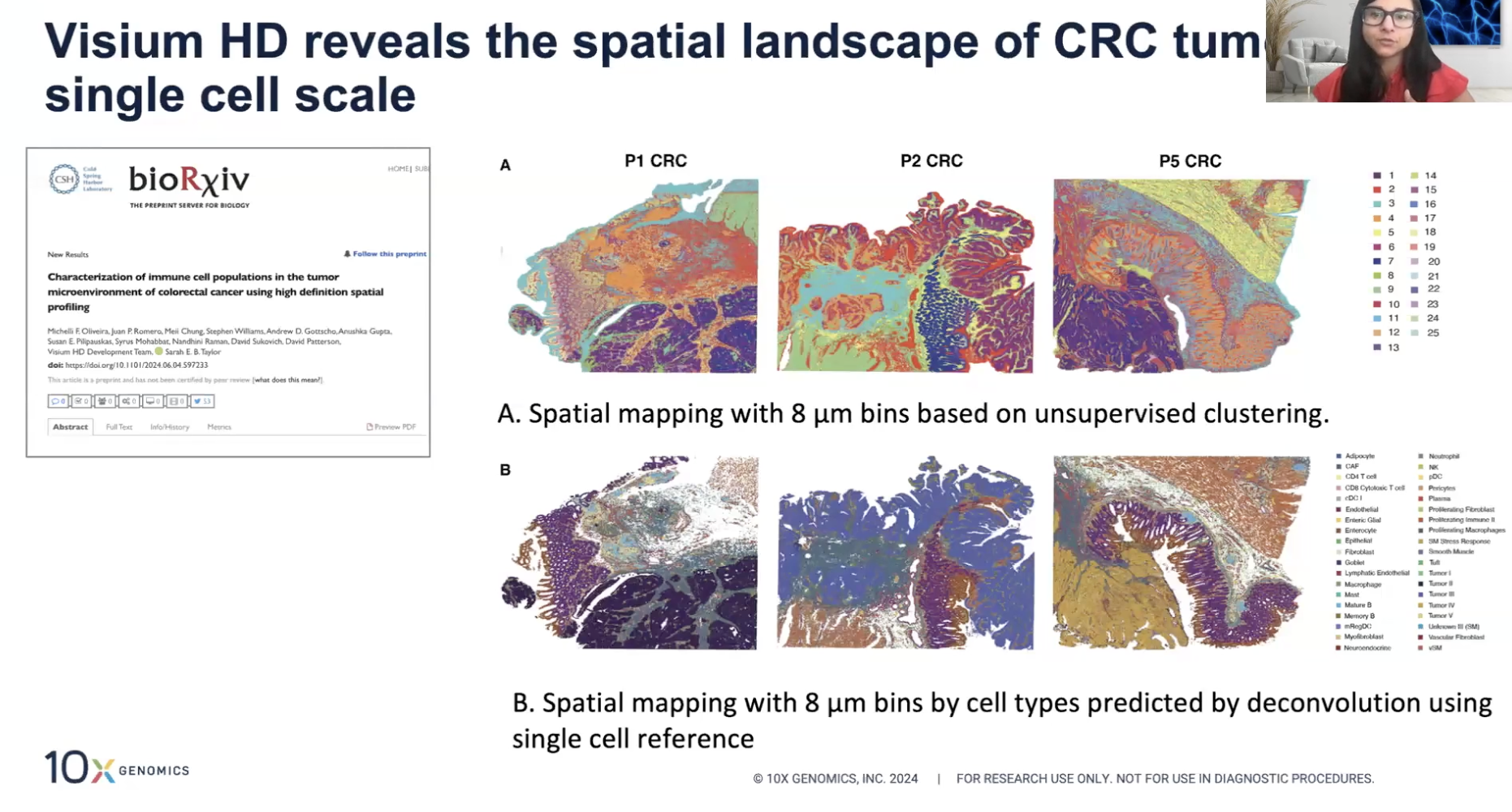
- 上图A是仅使用Visium HD数据进行无监督聚类得到的结果,而上图B是使用单细胞数据(24万5千个细胞,连续切片使用single-cell FLEX技术)生成了图谱,然后使用图谱对Visium HD数据进行注释得到的结果
- 得到的结果:无监督聚类的结果与单细胞数据进行反卷积,然后注释细胞类型的结果是非常相似的;但是在不同样本间比较的时候,单细胞数据注释后的结果可以给我们更好的insight
割核算法——用Visium HD数据得到类似于单细胞核分析的结果

- 补充:教程文章在这里
- 把不同的2✖️2μm的区域分配到细胞核所在的位置上(细胞核的位置可以通过HE获得。)具体来说,我们使用的算法是Stardist,这个算法会探测HE图像,然后把对应的2✖️2μm的捕获区域分配到细胞核所在的位置上。
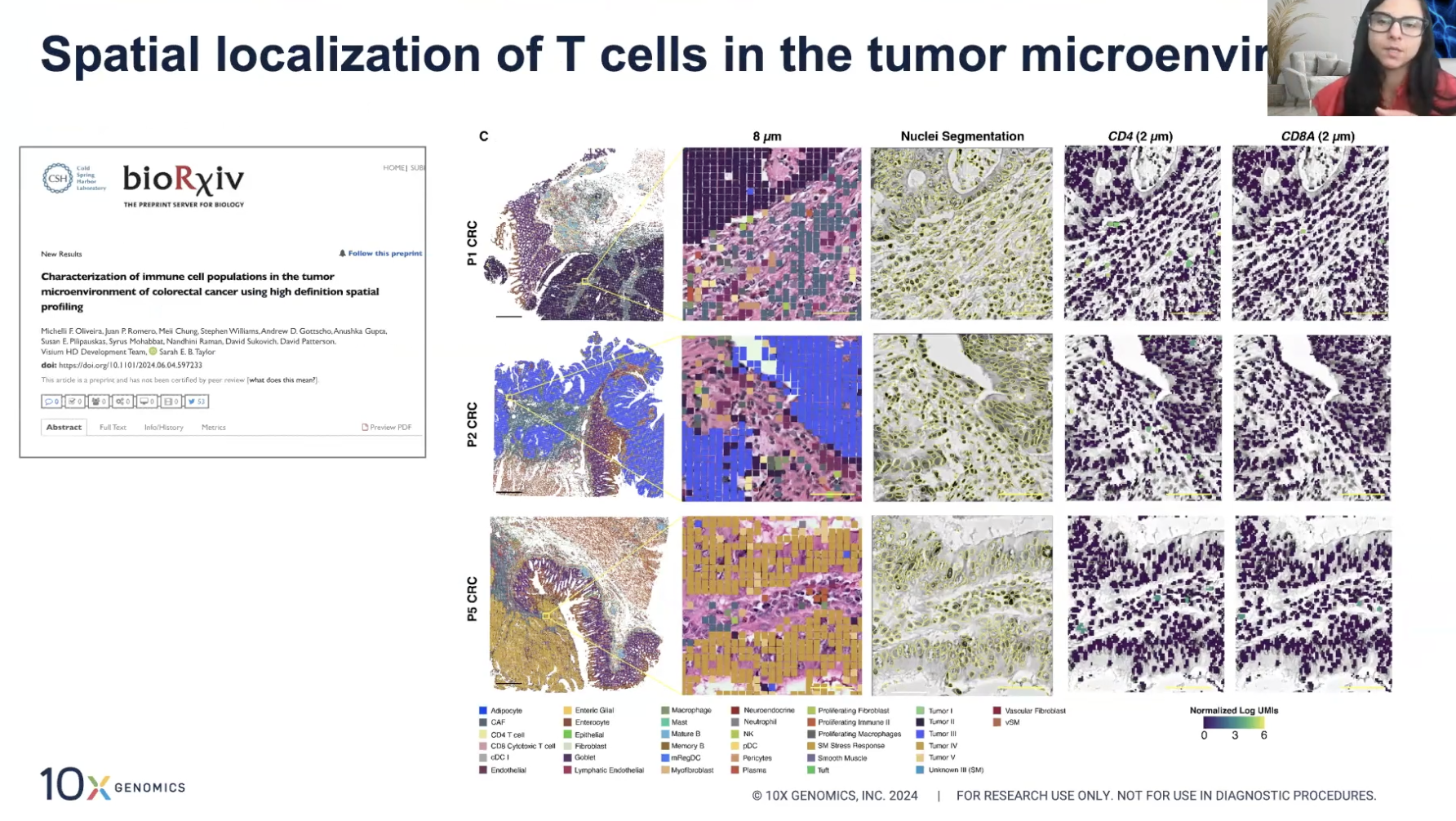
- 细胞核分割实现的结果:在原本的分析中,选用的8✖️8μm bin只包含一种细胞类型,但这意味着那些含有多种细胞类型的8✖️8μm bin所携带的信息被忽略了
- 在这篇预印本的分析中,作者发现在肿瘤的外围区域,有许多T细胞处于异质性的环境中,而这些细胞在分析中被忽略了,这时就可以使用割核算法去补回这一部份的信息
- 割核算法帮我们补回了CD4+和CD8+的T细胞,它们倾向于疏松分布,但是割核算法能帮我们把这些细胞找回来
Visium HD与 Visium V2的对比
- 都使用CytAssist机器
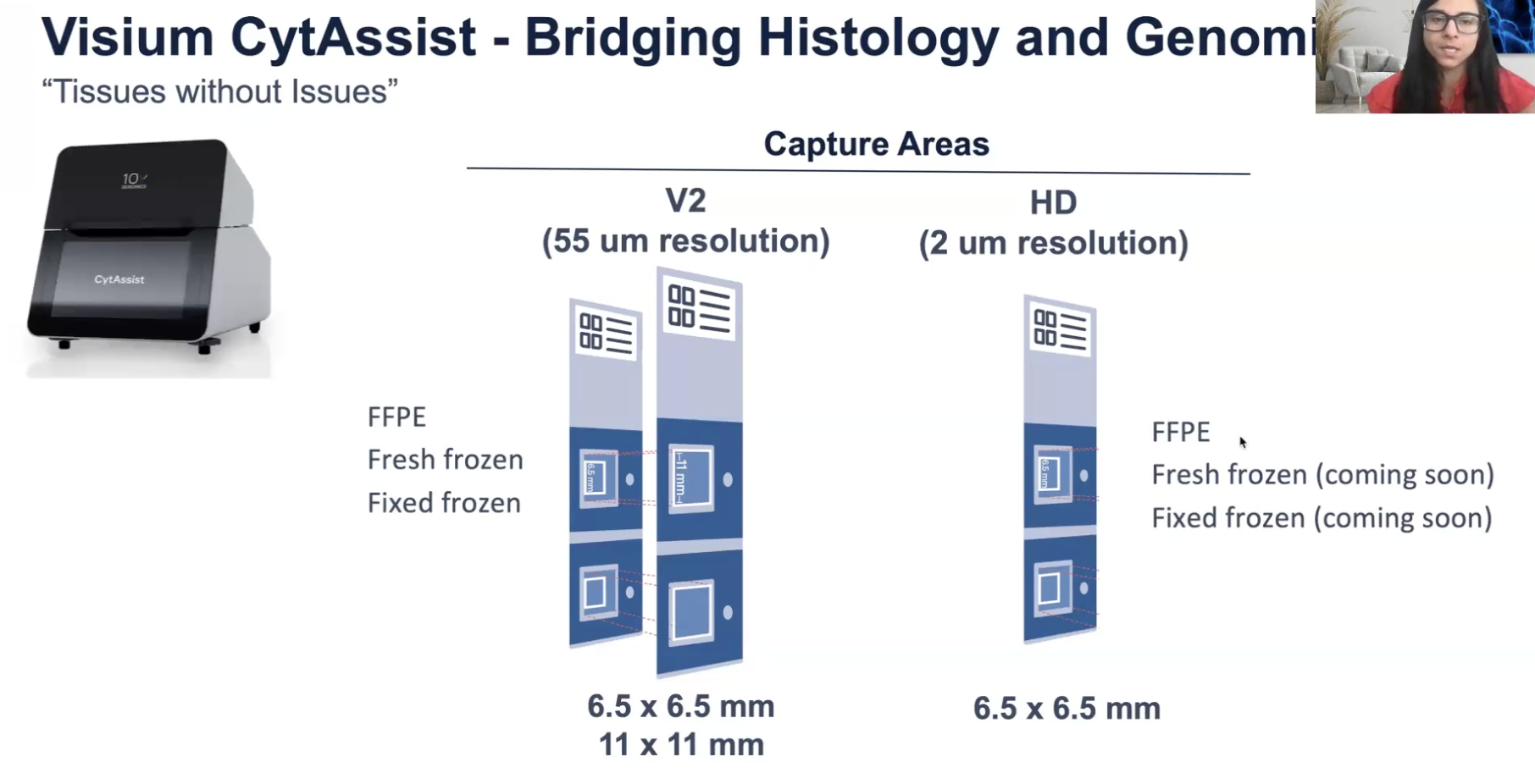
- Visium V2的分辨率相对小(最小为55μm,而且不连续),但是可以实现更大的捕获区域,具体来说是11✖️11mm,V2兼容的样本类型更多(新鲜冰冻、固定冰冻、FFPE)
- Visium HD的分辨率更大(2μm,通常简化为8μm分析),但是捕获区域只有6.5✖️6.5mm的版本,暂时只兼容FFPE样本
Visium HD总结



