单细胞实验简介
单细胞数据可以回答什么生物学问题

- 研究样本里面,每一个基因类型特异性表达的基因是什么
- 所研究的样本里面有哪些细胞亚型,是否研究样本中存在新的或稀有的细胞亚群
- 在不同的细胞亚群中,有哪些信号通路被激活了?
- 不同样本之间的差异(药物处理前后的)
- 通过trajectory分析,了解到发育或疾病发展过程中,细胞状态的变化
单细胞数据分析的流程
- 分析流程:
实际操作中,我们会在各个步骤中往复进行,也就是需要“来回横跳”,来获得更好的分析结果
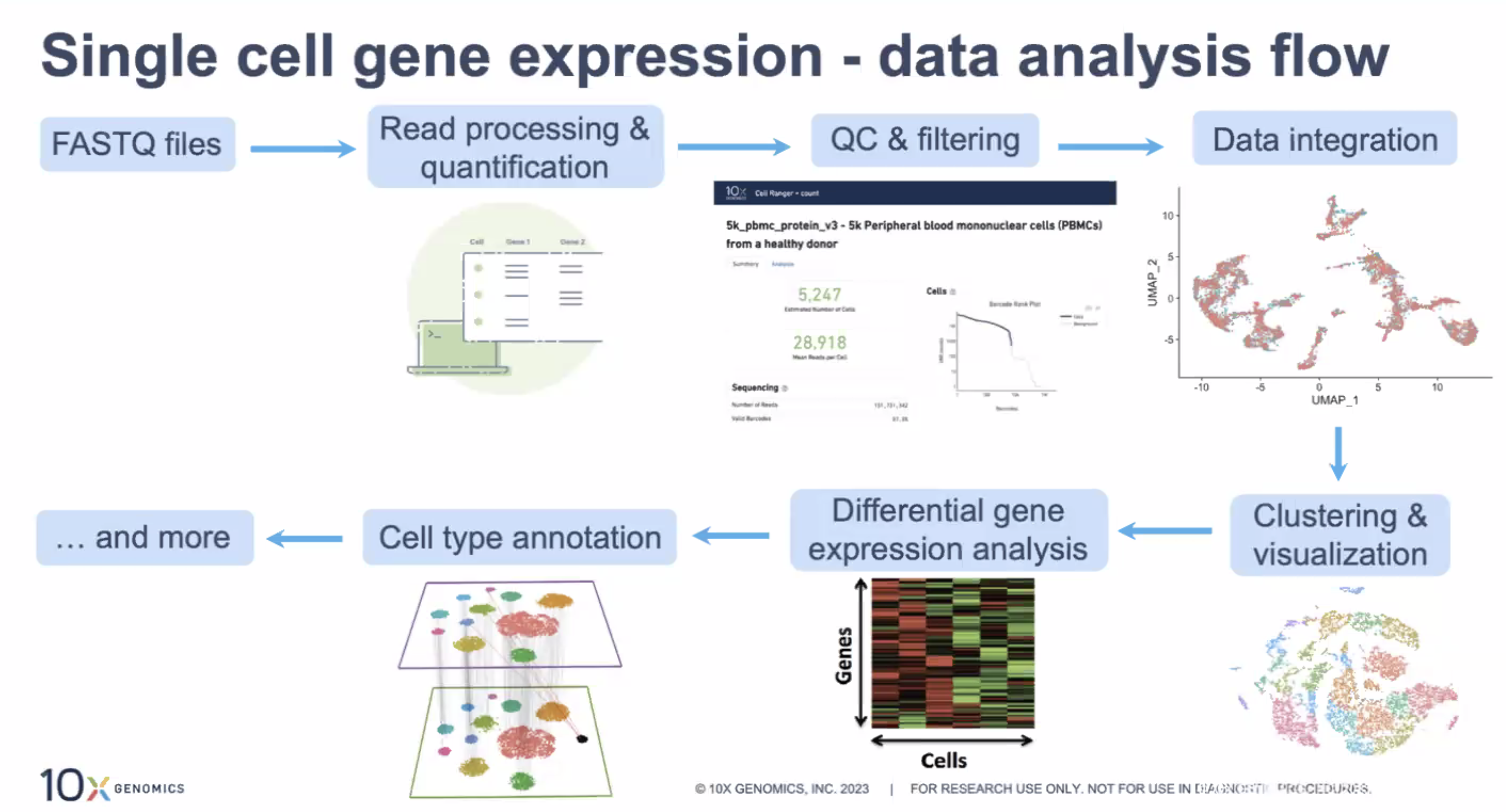
- 下机数据中得到的:原始的测序数据,通常是fastq文件
- 对原始测序数据进行处理,并计算出每一个基因的表达量
- 数据质控和细胞筛选
- 数据整合
- 聚类与可视化
- 差异表达基因分析
- 细胞注释分析
- 更高阶的分析……
单细胞分析的主要软件工具

cell ranger
- 10x研发的软件集,用于处理10x的单细胞数据
Loupe R & Loupe Browser
- Loupe R是一个R的软件包,可以把Seurat数据转换成Loupe Browser文件
- Loupe Browser是一个桌面软件,可以提供简单易用的界面,对10x的单细胞数据进行分析和可视化
有用的第三方软件工具
- 以下简称“三方工具”,是一个意思
- 独立运行的软件包
- 虽然10x对这些第三方工具不提供技术支持,但是这些第三方软件大大拓宽了我们进行细胞分析的广度和深度
细致的单细胞分析流程
数据预处理
- 首先,对原始测序数据的read进行预处理,用到的工具是cell ranger
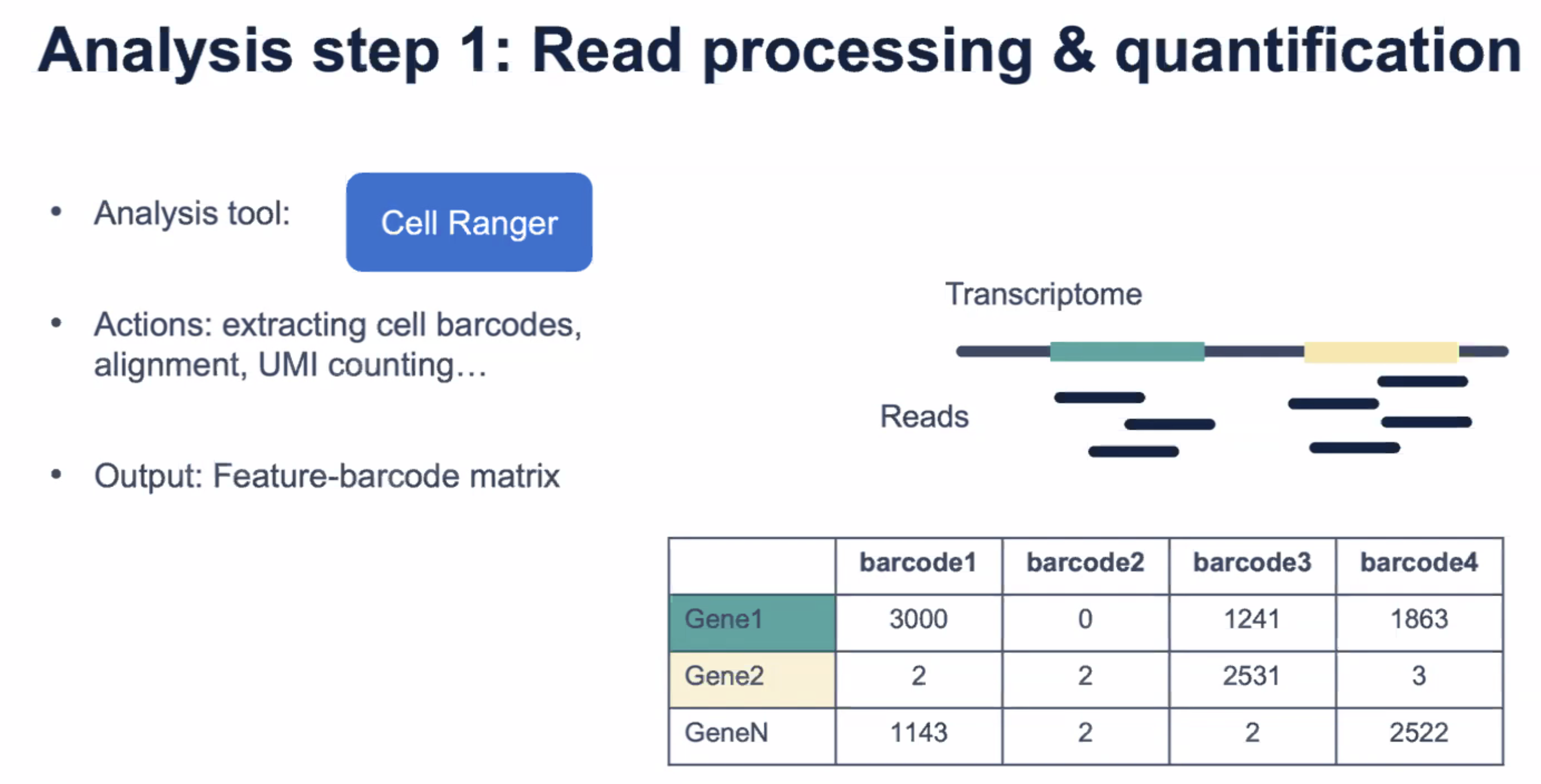
- cell ranger可以获取细胞的barcode信息,并将read比对到参考基因组上,以此计算出每个barcode内,每个基因捕获的RNA数目,也就是UMI
- 在这一步之后,我们可以得到一个feature barcode的矩阵,这个矩阵包含着每个barcode,每个基因对应的UMI数(也就是转录本的数目)
- 但是,有一些barcode不一定包含细胞,而只是背景噪音而已
质量控制与数据筛选
- 对原始的feature barcode数据质控,过滤掉没有细胞的barcode
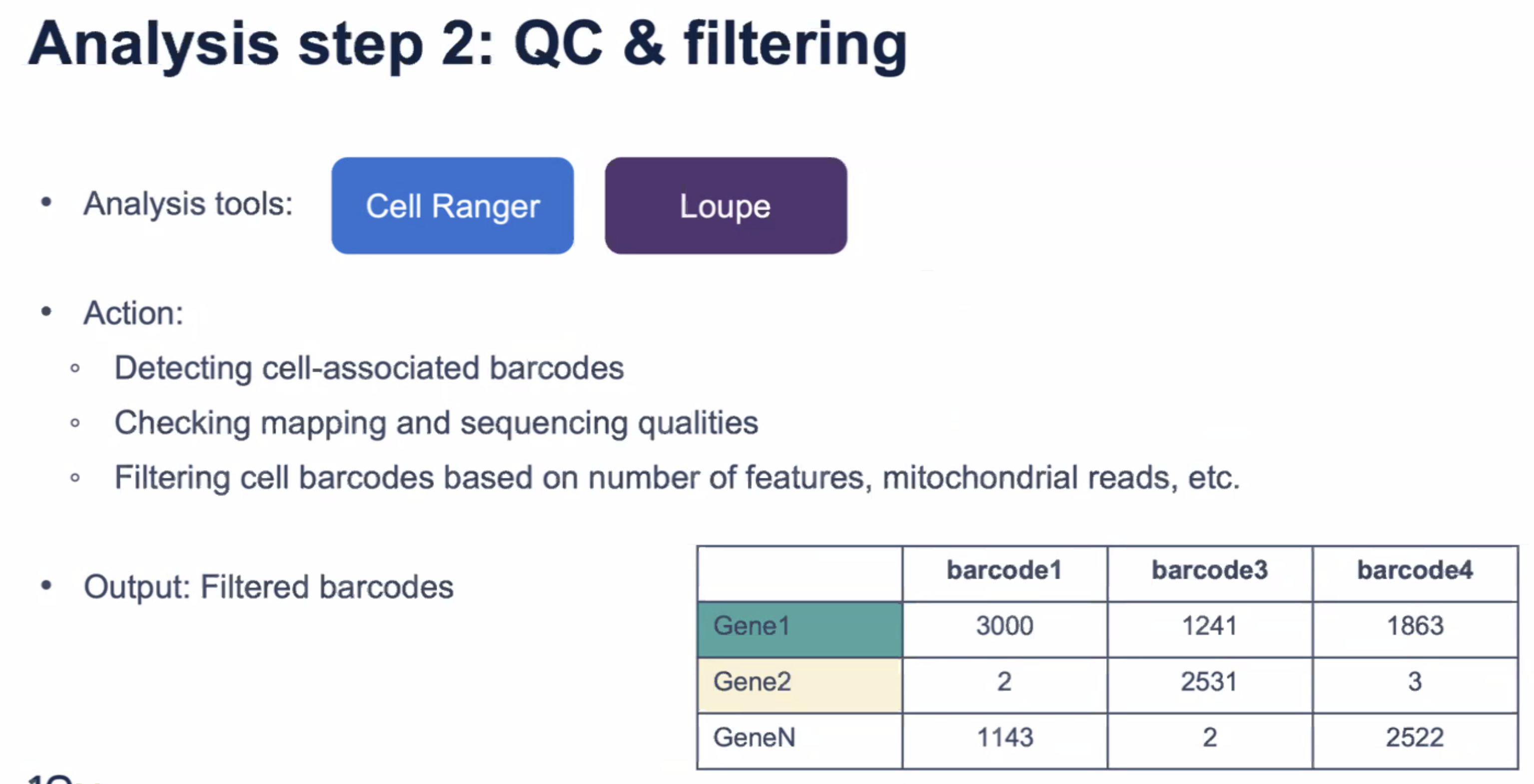
- cell ranger可以进行基本的质控分析,提供一些重要的指标,帮助分析样本和数据的质量,去掉没有细胞的barcode,剩下有细胞的barcode,并给出测序和比对等重要的质量指标
- Loupe browser可以去掉低质量细胞的barcode,过滤的标准包括每个barcode里面的基因总数,以及线粒体基因表达的百分比
在质控和过滤到低质量的barcode之后,我们将得到高质量的细胞群,进行下一步的数据分析
(可选)数据整合与去批次效应
- 数据整合
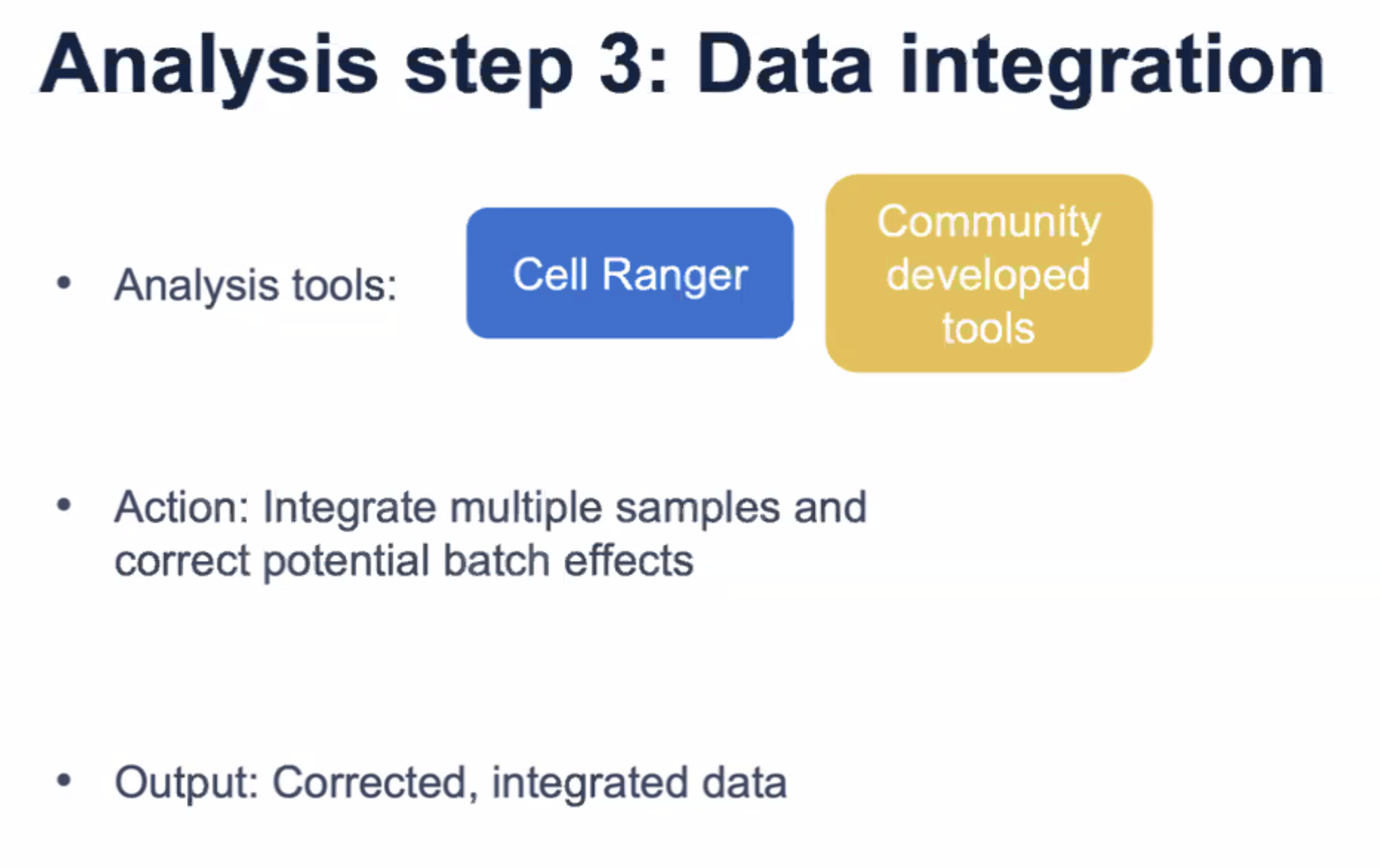
- 当我们有很多样本的时候,通常我们可以考虑将这些样本整合起来,进行分析,这一步可以使用Cell Ranger,也可以使用三方软件
- 目的:整合不同的样本得到的单细胞数据
- 如果有批次效应(多种技术要素造成),还要去批次

- 如果不同的样本间在tSNE或UMAP图上能显著的区分开(上图1),那就提示具有明显的批次效应,我们就需要对这两个样本进行去批次效应来整个分析
- 批次效应是由多种技术因素造成的,如果对相同的生物样本使用相同的10x试剂,并同时处理,这样的批次效应可能就很小
- 然而,如果样本是在不同的时间,由不同的人员进行处理,批次效应可能就比较显著
聚类和可视化分析(tSNE和UMAP哪个更好没有定论)
- 根据细胞的基因表达谱对细胞进行分组,并在二维的图上进行数据的可视化(单细胞数据很复杂,我们需要降低维度,以便我们能在二维平面上看到单细胞数据)
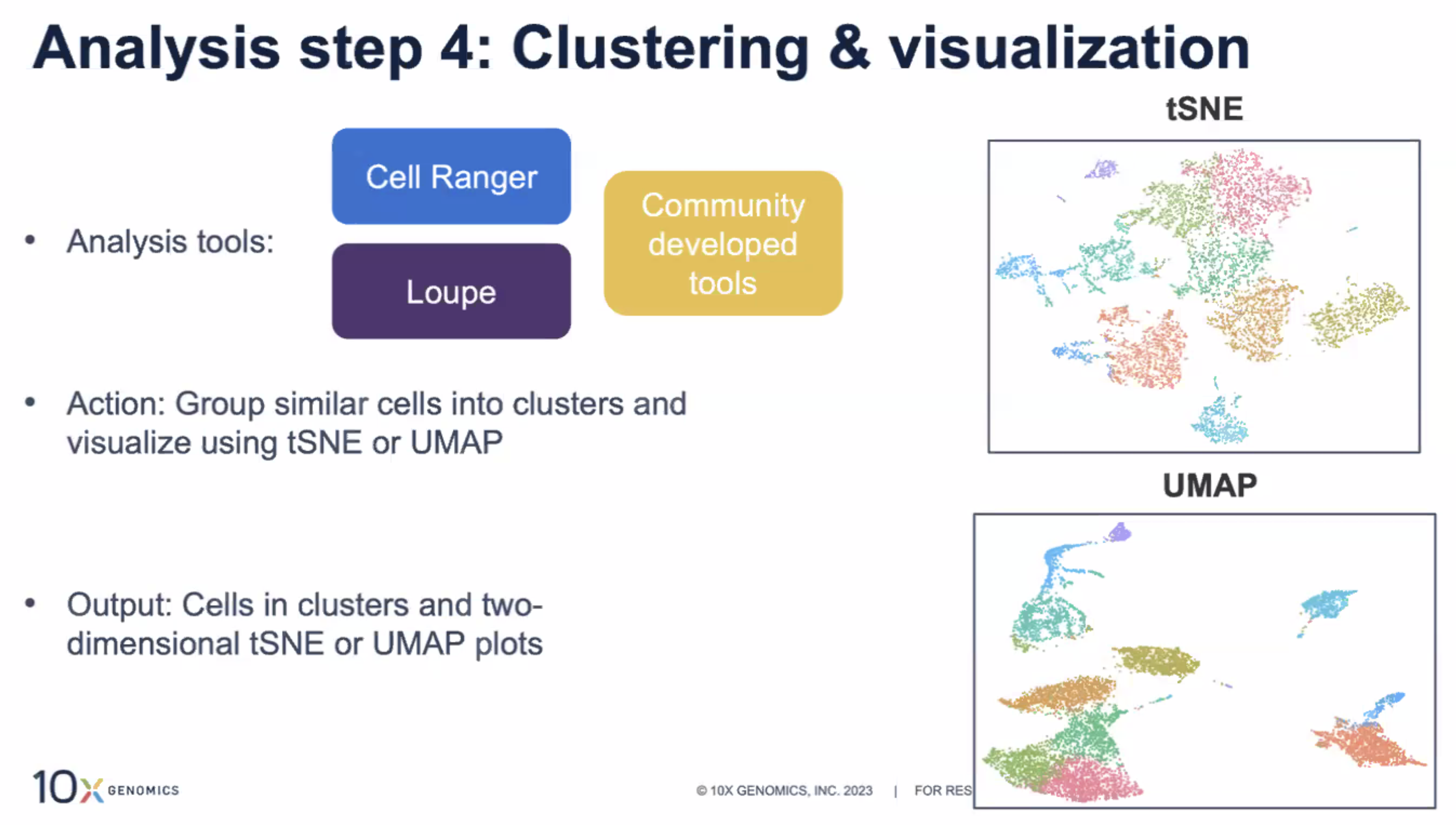
- 常见的可视化方式是tSNE和UMAP,对于相同的样本,这两种方法可以提供略有不同的,集群群体的结构
- 单细胞数据分析是一个非常活跃的领域,tSNE和UMAP到底哪一个可视化效果更好,还没有明确的结论,可以根据自己的数据和参考文献来选择分析方法
差异基因表达差异分析

- 对每个聚类进行差异基因表达分析,这个步骤可以在cell ranger的默认参数下进行,并且自动得到相应的结果
- 也可以使用Loupe Browser和三方软件来实现同样的目的,使用这些软件进行差异基因表达的分析可以是高度定制化的
- 在这个步骤中,我们经常要做的就是把每个cluster的细胞和其他cluster的细胞进行比较,然后找到他们之间的基因表达的差异,输出的结果是每个cluster中富集的基因列表,以及这些基因对应的倍数变化,还有调整后的p值
- 在使用Loupe和三方软件的过程中,我们可以定制在两个特定的细胞群之间进行差异分析
细胞注释
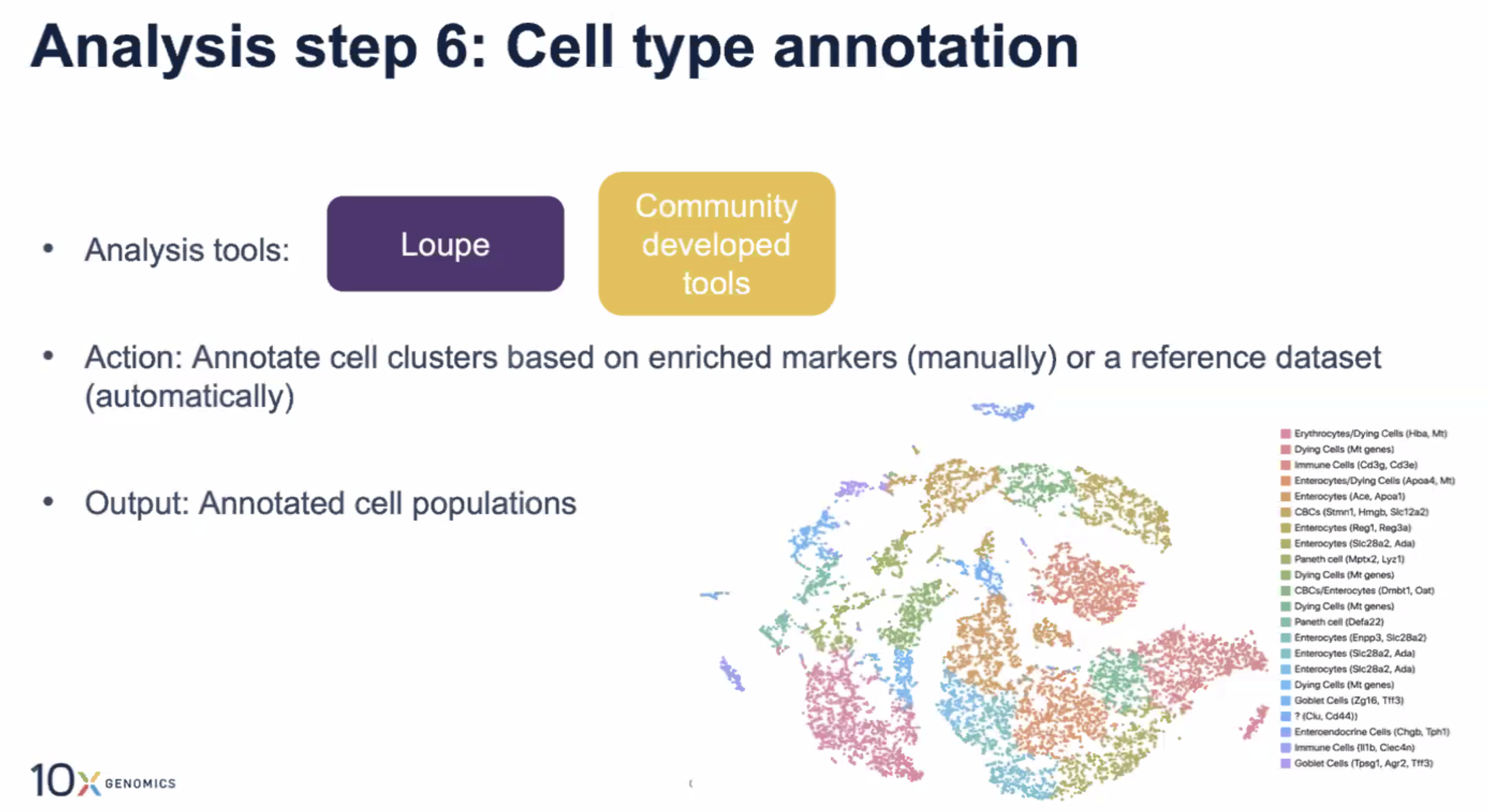
- 找到每个cluster,并且对每个cluster进行细胞注释
- 进行这个步骤由很多选择,我们可以根据上一步发现的,每个cluster的marker基因进行手动注释,也可以用一些三方软件进行自动的细胞注释
- 在这一步结束之后,所有的cluster都会被注释为特定的细胞类型
一些三方软件介绍

- 非常流行、有用的分析工具:Seurat和Scanpy,都可以对单细胞数据进行分析和可视化
- 更高阶的数据分析可以通过三方软件进行
- 信号通路分析,找到不同cluster中富集了哪些基因
- trajectory/RNA速率分析,判断细胞状态之间的转变
- 可以通过scRNA-tools数据库来探索用于单细胞数据分析的工具目录
- 10x的官网上也有不同主题的分析指南,包括single-cell, Visium(含HD)和Xenium,可以看analysis guide,这里有致力于解决高阶数据分析问题的内容,这些内容也将不断被丰富
Loupe R
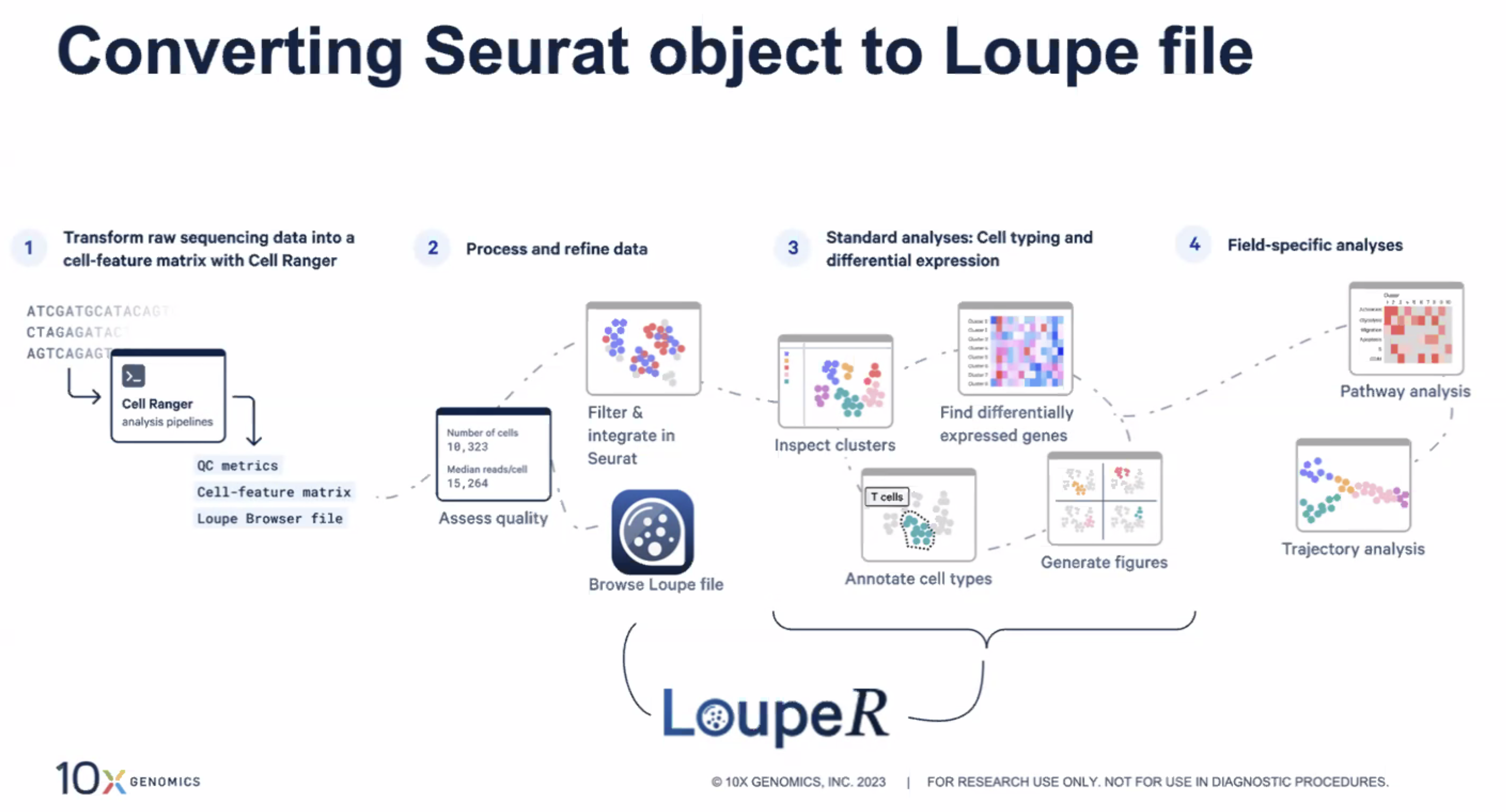
* 虽然Loupe Browser提供了非常直观的分析界面和强大的分析功能,但一些高级分析,比如细胞的自动注释,或者批次矫正仍然无法在Loupe Browser中完成,而Seurat等三方工具可以完成这些分析
* 现在,用户可以使用Loupe R这个软件包,将两个平台进行结合,Loupe R允许用户将Seurat数据转化为Loupe Browser文件
* 比如,在使用Seurat进行批次矫正之后,我们可以使用Loupe Browser进行更多的下游的数据分析
各种软件的运行要求
Cell Ranger需要的技能

- Linux系统执行各种程序
- 了解实验设计
- Linux系统中的命令行
- 所在机构的数据管理系统
- Linux系统出现问题的时候,我们要联系谁
Loupe R & Loupe Browser
- Loupe R:基本的R编程的语言
- Loupe Browser:苹果或微软操作系统
- 实验设计与生物学知识
- 诠释差异表达基因的结果
三方软件:例如Seurat和Scanpy

- 实验设计与生物学知识
- R与python的编程能力
- 软件包的默认参数
- 诠释数据分析的结果
- 10x support官网有更多说明


