beadchip 表达谱芯片的特点
- 上样量低:每个芯片在一轮反转录的情况下仅需50-200ng RNA;
- 重复性高且检测通量大:
- 微珠芯片(beadchip)技术和“无序自组装”方式,每种探针的微珠有的30倍重复;
- “片中片”的设计等特点保证微珠芯片的高重复性和低批次差异性
- 特异性高
- 数据准确性高
- 检测的费用相对低廉
beadchip 表达谱芯片制备的基本原理和步骤
- 这里我们讨论的主要是Sentrix Beadchip,但还有另外一种Sentrix Array Matrix,SAM

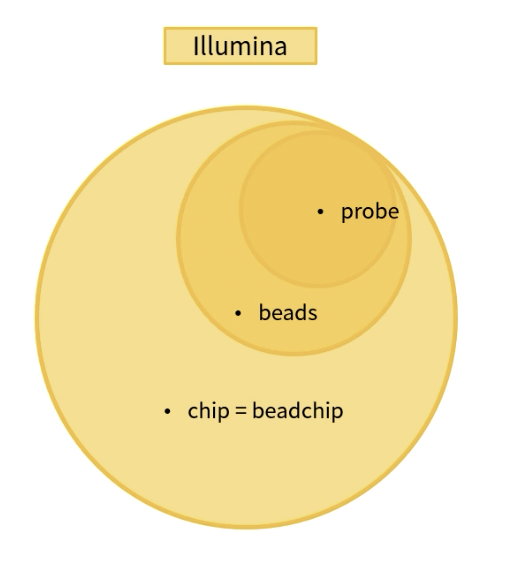
- Beadchip 主要由硅片Silicon Slides,也叫Silicon Wafer,和二氧化硅硅珠两部份组成,硅片是微珠的容器
- 一张成品Beadchip一般可以分为1-16个反应池,可以分别检测对应数量的样本,反应池内就是经过制作的硅片,通过一定的技术蚀刻出若干个小孔,每个小孔的尺寸都在微米级,正好容纳一颗微珠
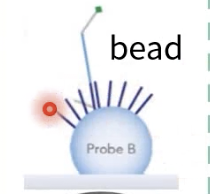
- 微珠是芯片最核心的地方,微珠的体积很小,也只有微米级,每个微珠的表面都各自耦联了70万个某一种相同序列的DNA探针,也就是形成了一个长满了DNA链的“毛珠”;一个反应池内,检测一个样本,大概有30个同样序列的微珠
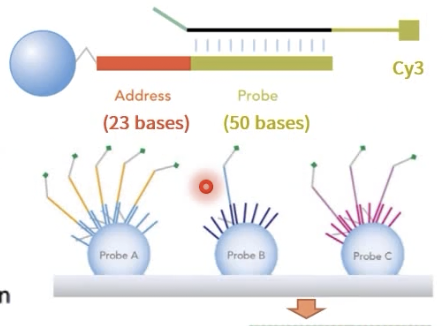
- “毛珠”上面的DNA探针的长度大概是73个nt,包括2个功能区域
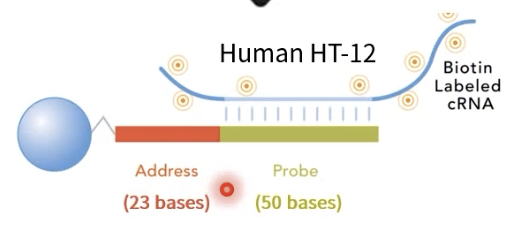
- Address序列:靠近珠子,是DNA的5’-端,识别微珠的标签序列,通过碱基排列,也就是4^23,可以得到微珠的“身份证号码”
- 探针序列:远离珠子,是DNA的3’-端,可以与待测cRNA形成互补杂交
- 这两者是一一对应的关系
- 在Illumina生产芯片的过程中,要把做芯片的上万种微珠,按照预定的比例进行混合,挂到硅片上;微珠会随机地落在小孔当中,然后通过检测每个小孔的address序列,就可以知道该微珠是负责检测哪个转录本的了
- 由于微珠是随机分布在每一个孔内的,世界上没有任何两片Illumina芯片是完全相同的,出厂时每片Illumina芯片都会带上一个地址文件,标注每一张芯片的每一个微孔当中分别是哪种微珠;完成实验,得到扫描数据后,要从Illumina下载这张芯片对应的地址文件,把探针序列转化为address序列,才能解读这张芯片
Beadchip 表达谱芯片的基本检测原理和步骤
- 检测样本的制备和Affymetrix是基本一致的,也是要生产带标记的cRNA,再和探针杂交
- 我们常用的标记有两种
- 生物素biotin,再加荧光结合的抗生物素结合蛋白
- 在cRNA合成所用的碱基上直接掺入荧光分子
- 标记了荧光的cRNA会被珠子“粘住”,而且表达量越多的cRNA被“粘住”的数量就越多,荧光的强度就越高