Affymetrix表达谱芯片的特点

- 应用最广泛的是表达谱芯片和SNP分型芯片,但目前SNP研究逐渐被测序取代,而且Illumina的芯片也很有优势
- 最具竞争力的可能还是品种齐全,应用广泛的表达谱芯片
- 表达谱芯片覆盖物种广泛
Affymetrix表达谱芯片种类
- 传统的3’-IVT芯片(in vitro transcription),使用oligo-DT引物和T7逆转录酶获得的cDNA,其得到的cDNA主要是靠近mRNA 3’-端的,探针主要针对每个基因的最后1-2个外显子来进行设计
- 新一代全转录组WT(whole transcriptome)芯片,使用随机引物和T7逆转录酶获得的cDNA,其得到的cDNA会覆盖转录本上更多的区域,相应的探针也是针对基因的整个转录本进行设计的
Affymetrix表达谱芯片制备的基本原理和步骤
- Affymetrix公司采用的技术称为光蚀刻技术,也可以称为光引导原位寡聚核苷酸合成技术
- 通过光的引导把碱基一个一个加到基质上,形成探针,表达谱分析(RNA)时是25nt,SNP分析时是30nt
Affymetrix表达谱芯片的组成
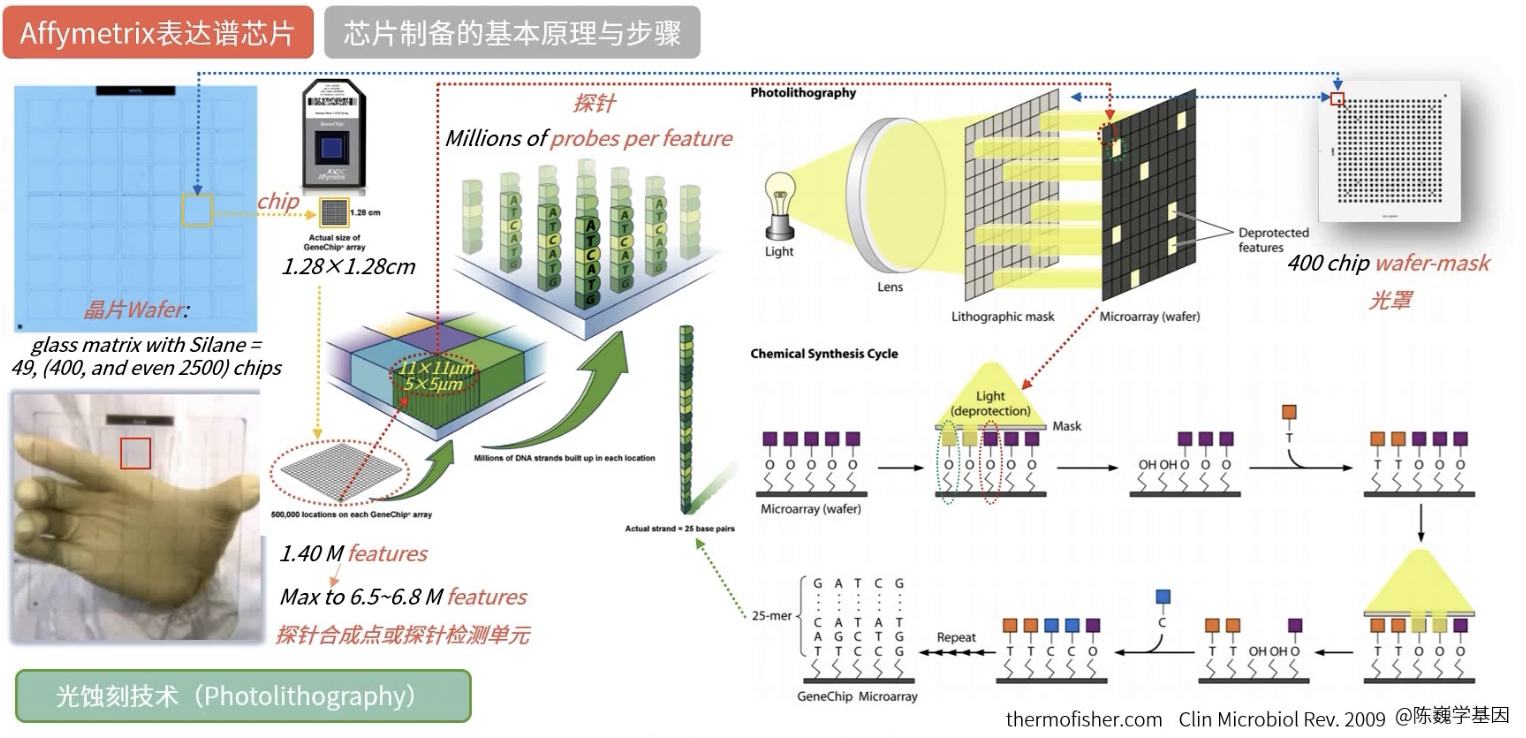
- 生物芯片的基片:一张大的玻璃片,表面结合硅烷分子而形成的玻璃硅片,专业名称Wafer,我们称之为晶片
- 上面的每一个格子都是未来要剪下来的芯片Chip,长宽都是1.28cm,最后会安装在比SD卡大一倍的外壳里,成为成品的卡式芯片
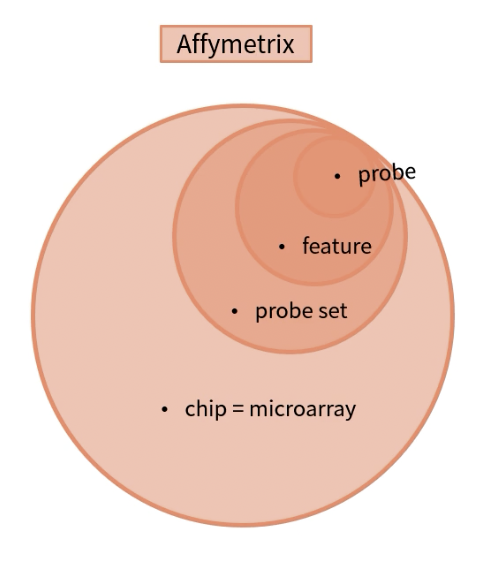
- 晶片根据大小不同,可以有49,400甚至2500个芯片,每个芯片上,又划分出140万,如今能达到650-680万个小格子,每个小格子被称为一个feature,称为一个探针合成点或者探针检测单元,这也就是未来芯片扫描图上的一个光点
- 每个feature的长宽,对于3’-IVT芯片一般是11µm,WT芯片是5µm,上面会通过高密度点阵技术和光蚀刻技术生长出几百万个具有相同序列的DNA链,也就是探针
- 制造DNA探针的思路是:一步一步,将单个核苷酸连在链上,构成一条25nt的长链;光蚀刻技术通过使用蚀刻光照和紫外光,可以实现精确的控制,从而将含有A/T/C/G四种碱基的核苷酸连接到芯片上的特定feature上
- 光照和晶片具有一一对应的关系:一个格子对应一张chip,每个格子又具有大量的小格子window,暂且称为通光孔,和每个chip上的feature一一对应,这些通光孔是否通过紫外光就决定了新的碱基是否可以连接到核苷酸链上
- 如图右上角,我们可以看到,一个通光孔对应一个feature,有少数通光孔是通光的,则那些对应的feature是亮起的,被称为deprotected features,即失去保护的features
- 失去保护的features(绿色框圈起的),会被添加上新的碱基,被保护的features则不会
Affymetrix表达谱芯片合成的过程
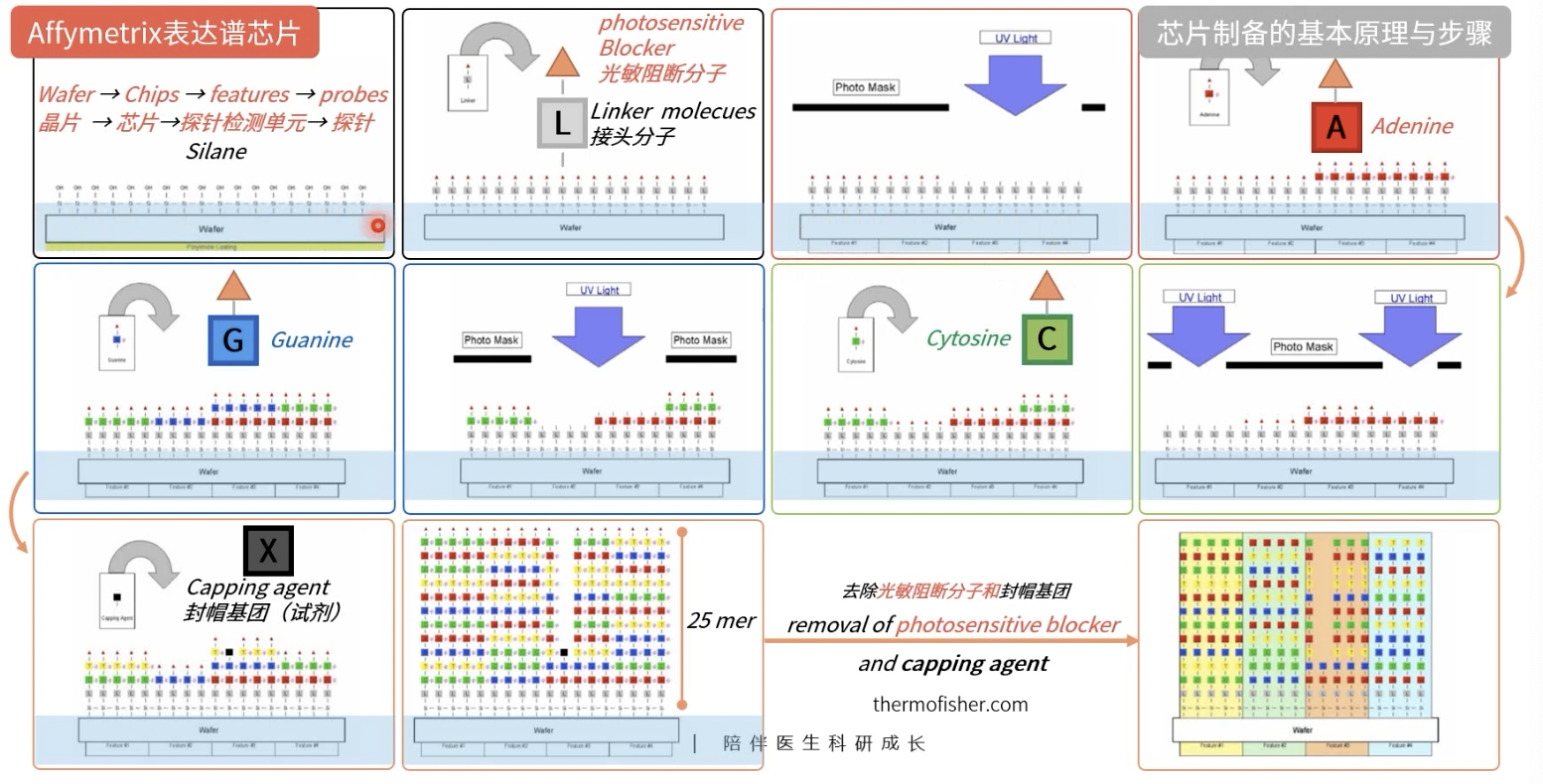
- 图一:晶片浸泡在含有硅烷的液体中,导致硅烷分子可以与玻璃相结合,这时每个和晶片结合的硅烷分子都将为构建新的DNA链,或者说探针提供起点,硅烷分子排列得越紧密,DNA探针组装得就越紧密
- 图二:我们会将接头分子添加到每个硅烷分子当中,接头分子以L表示,作为添加第一个核苷酸的起点;光敏阻断分子是一种阻止DNA链延长的保护剂分子,用红色小三角表示
- 当阻断分子作为一个保护基团出现在DNA链上,不能向链上添加新的核苷酸
- 保护基团是对光敏感的,也就是说光可以去除这些保护基团
- 每个新添加的核苷酸分子都添加了这种保护基团的修饰,一旦受到紫外光的照射,保护基团就会从所连接的核苷酸的羟基上掉下来,把羟基暴露出来供DNA链的延长
- 图三:在掩模版的作用下,芯片的右边两个feature被照射,失去保护基团
- 图四:添加第一个核苷酸,芯片上右边两个feature是刚刚被照射过的部份,去掉了保护基团的。它们就会与第一个核苷酸,图中是腺嘌呤脱氧核苷酸A结合;而刚刚没有被紫外光照射的左边两个feature是不会与新加入的A结合的
- 图五(注意第二横行的图是从右往左进行的):在另一种掩模版的作用下,此时芯片(从左到右)的第一和第四个feature受到紫外光的照射,失去保护基团
- 图六:添加第二个核苷酸,图中是胞嘧啶脱氧核苷酸
- 图七&八:图三&四、图五&六的step-by-step
- 图九(第三行左一):小插曲,偶尔会有某个被光照去掉了保护基团的DNA链在加入新的核苷酸后没有结合,这可能会导致DNA探针合成的过程出现某种“错位/缺失突变”,我们应对这样的方法是在每次连接反应和光照之间加入某种封帽基团(试剂),以黑色的X进行表示,其会结合没有保护基团的DNA,但是没有光敏感性,因此这种偶尔因“跟不上脚步”而出错的探针就会被废除
- 图十:在探针长度达到了设计长度(图中的mer是monomeric unit,单体单元的意思,在单链的核苷酸链中意思与nt相同,双链的核苷酸链中与意思与bp相同)后,添加核苷酸的反应就会停止
- 图十一:最终的反应是去除光敏阻断分子和封帽基团;再复习一次,表达谱分析(RNA)时是25nt,SNP分析时是30nt
- Affymetrix的表达谱芯片3’端与硅烷相连,DNA探针合成的顺序是3’-5’
- 我们是以晶片的形式去合成DNA探针的,在合成结束后,晶片会被切割成芯片,然后塞入对应的外壳中
NimbleGen表达谱芯片合成的过程与Affymetrix非常相似
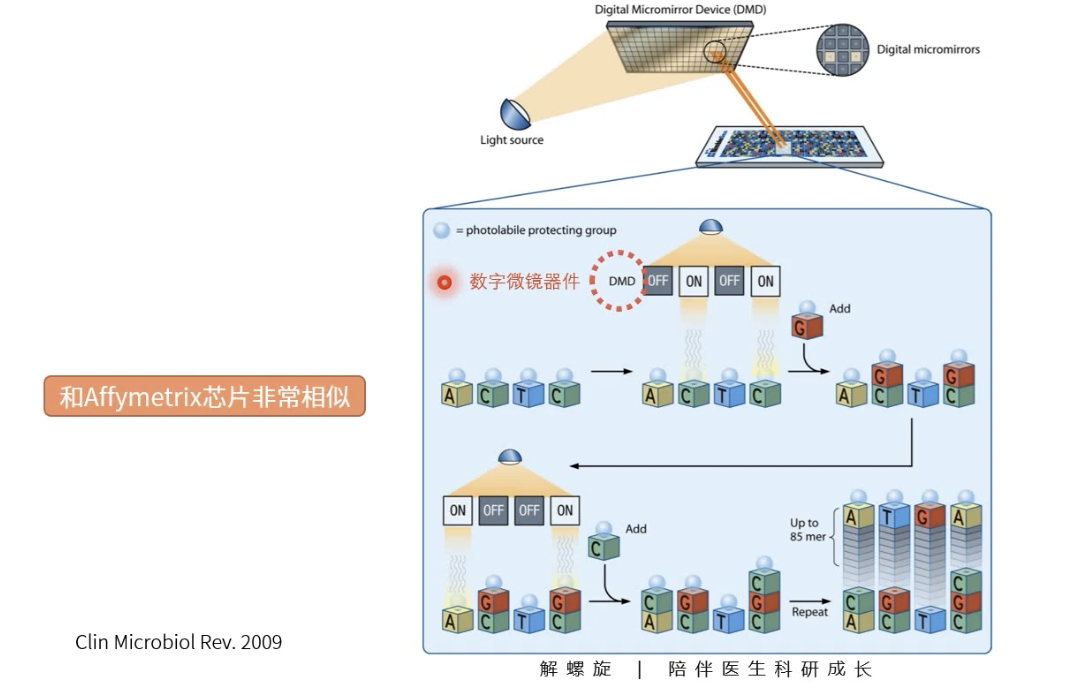
- 只不过Affymetrix在调整紫外光光照时使用的是掩模版,而NimbleGen使用的是数字微镜器件Digital Micromirror Device,简称DMD
Affymetrix表达谱芯片检测的基本原理和步骤
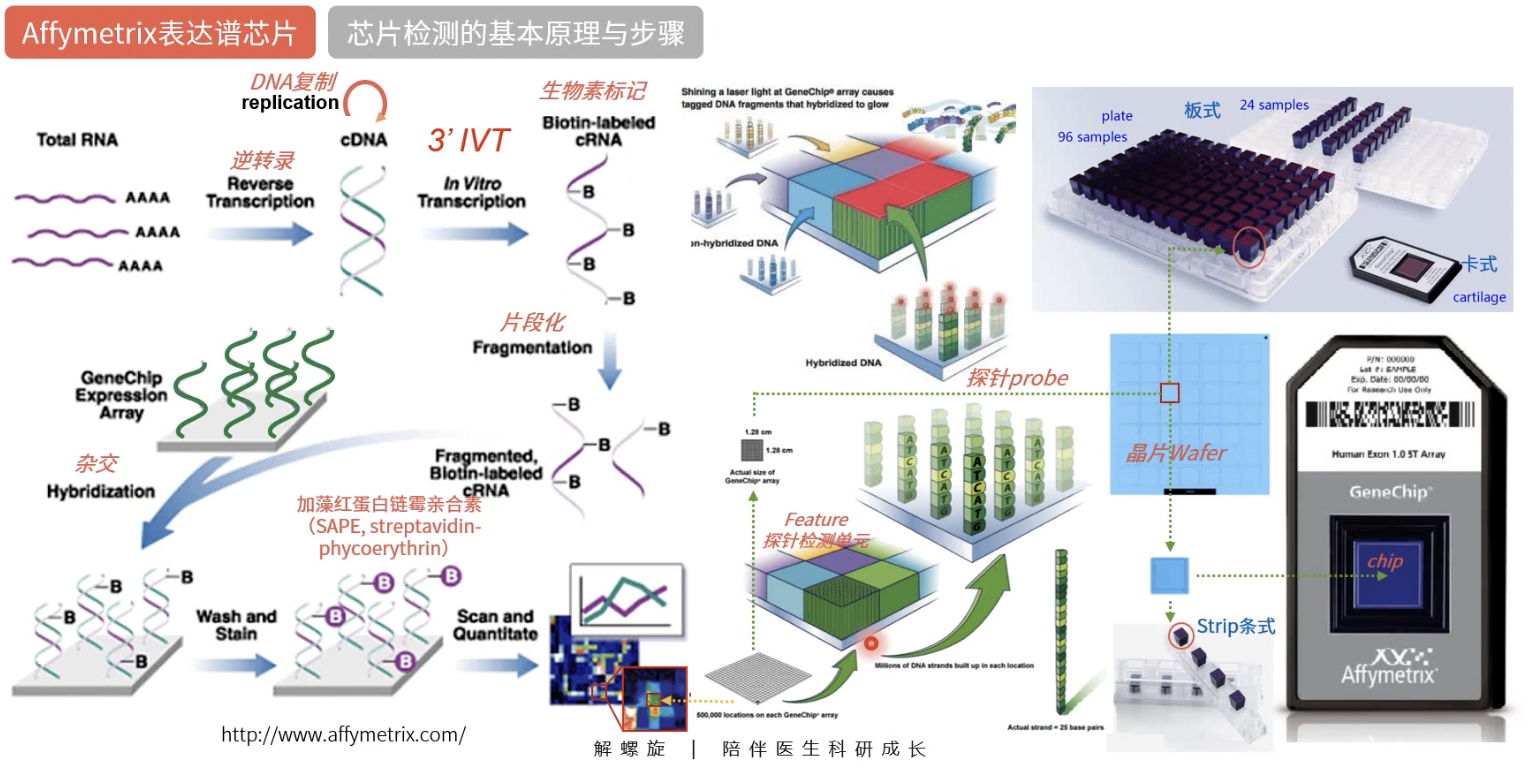
- 以3’-IVT芯片为例
- 实验开始时,加入提取的、待检测的转录组RNA,先通过逆转录得到第一链的cDNA,紧接着通过复制合成第二链的cDNA,变成双链cDNA,得到的双链cDNA会作为接下来转录的模版
- 接下来,我们会使用掺有生物素biotin标记的UTP在内的4种单核苷酸混合物进行体外转录,转录得到的cRNA因为转录的原料中含有被生物素标记的UTP,所以转录出的cRNA片段是带有cRNA标签的
- 将cRNA达成片段后,再依照碱基互补配对的原则,与芯片上的探针进行杂交,将未结合的cRNA片段洗脱后用标记了藻红蛋白的链霉亲和素streptavidin phycoerythrin,也就是SAPE对芯片进行染色,SAPE中的链霉亲和素会与生物素进行特异性结合,而藻红蛋白可以在激发光的照射下散发出红色荧光,这里可能还有一部荧光信号放大过程,之后进行激光扫描,得到一张有密密麻麻光点的照片
- 这张照片就是荧光信号的矩阵,光点的X-Y轴位置是探针的ID号,光点的光强度对应着被杂交的cRNA的量,而这个cRNA的量就反映了样本中mRNA的表达量
- 我们在上一步讲过,每个光点都是一个feature,探针合成点或者说探针检测单元;每个feature含有几百万个完全一样的25mer长的探针,每张芯片上有几百万个feature,每个芯片都来源于初试晶片的裁切,装入外壳,成为不同的芯片
- 芯片的种类有卡式、条式和版式,卡式是经典芯片类型,条式和版式是为了增加检测的通量,减少批次差异
- 版式芯片可以留意一下Affymetrix官网,其中U133A/GPL-96和U133B/GPL-97,这套芯片主要是版式的,其中U133A和U133A 2.0/GPL-571 有效探针是完全一样的,但U133A 2.0主要以卡式形式出售
- 另外,Affymetrix加入了独特的PM/MM探针设计,使得检测的特异性和灵敏性进一步提高,在PM/MM探针设计中,MM探针是有效的内参照,其与PM探针一样,可以与非特异性序列结合,进而可以将不同来源的样本中的背景信号有效扣除,这种独特的设计对于区分特异性和非特异性杂交是相当灵敏的
- 可以说,相比于单一的基因探针,PM/MM探针的高特异性和高灵敏度更适合用于检测低表达丰度的基因
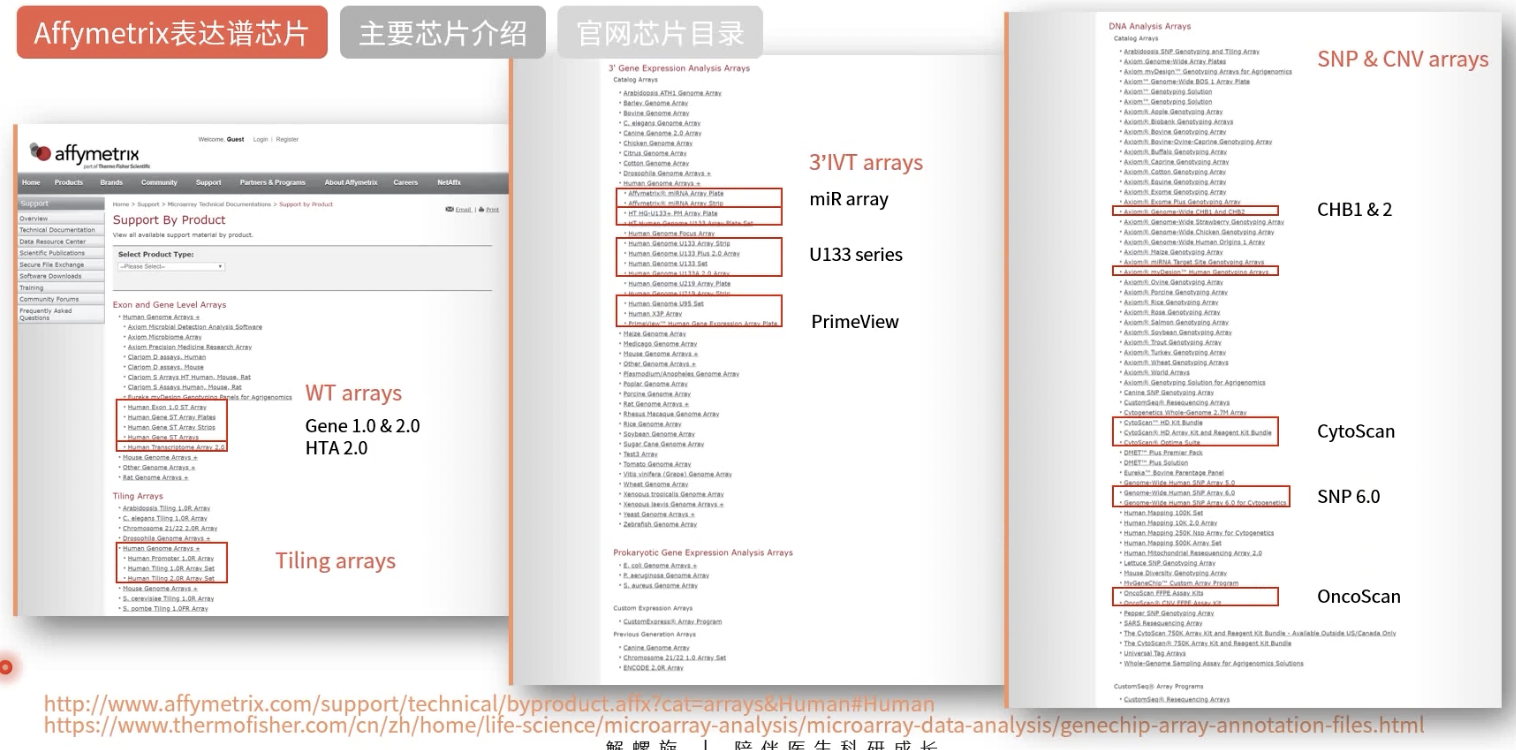
Affymetrix主要表达谱芯片介绍
3’-IVT
- 3’-in vitro transcription,3’-端表达谱芯片
- 3’-IVT芯片的探针主要针对每个基因的最后一、二个外显子来进行设计,(注意不是3’-UTR),以检测到的3’-端信号值推论到整个转录本而得到基因表达水平值;检测时采用Oligo dT引物和T7逆转录酶产生cDNA
- 这种设计已被广泛使用,并有大量发表的文献支持,其中包括经典的U133芯片和较为经济的PrimeView 芯片等;
*这种芯片的缺点:
- 这种检测方式基于的假设是每个基因3’-端是特异的,很多同源基因是区分不开的;
- 3’-IVT芯片对于只有一个转录本产物的表达检测是适当的,但对于3’-端序列相同的不同转录本则只能得到一个3’-端表达的概括值,而不能区分转录本,所以不能用于研究基因可变剪接(splicing)
- 价格上没有优势
全转录组芯片
- WT,Whole Transcriptome
- 为当前流行的芯片类型,比3’-IVT优势更多,性价比高,强烈推荐;分为Transcriptome Array(如HTA2.0)、Exon ST Array(如HuEx 1.0 ST)和Gene ST Array(如HuGene 2.0 ST) 等;检测时采用随机引物和T7逆转录酶产生cDNA;
- 这种芯片的优点
- 结果准确:对一个基因众多外显子设计多组探针,覆盖区域广,综合评价,避免由于3’-端序列特异性差造成的误差和混淆;
- 全转录本表达谱:以全基因组的每个转录本为单位设计探针,为转录本水平的表达研究,结果更加细致、准确可靠;
- 可变剪接的分析:在检测不同的转录本的表达量的变化的时候可以对可变剪接进行分析。特别是HTA2.0,在外显子连接部位还专门设计了叠瓦排列的探针,研究基因剪接非常优秀的芯片
- 可以检测长链非编码RNA(lncRNA)
- 在进行后续定量设计时,要考虑到引物设计的位置,否则验证结果可能会与芯片结果不一致
两种芯片的差别:用可变剪接来分析
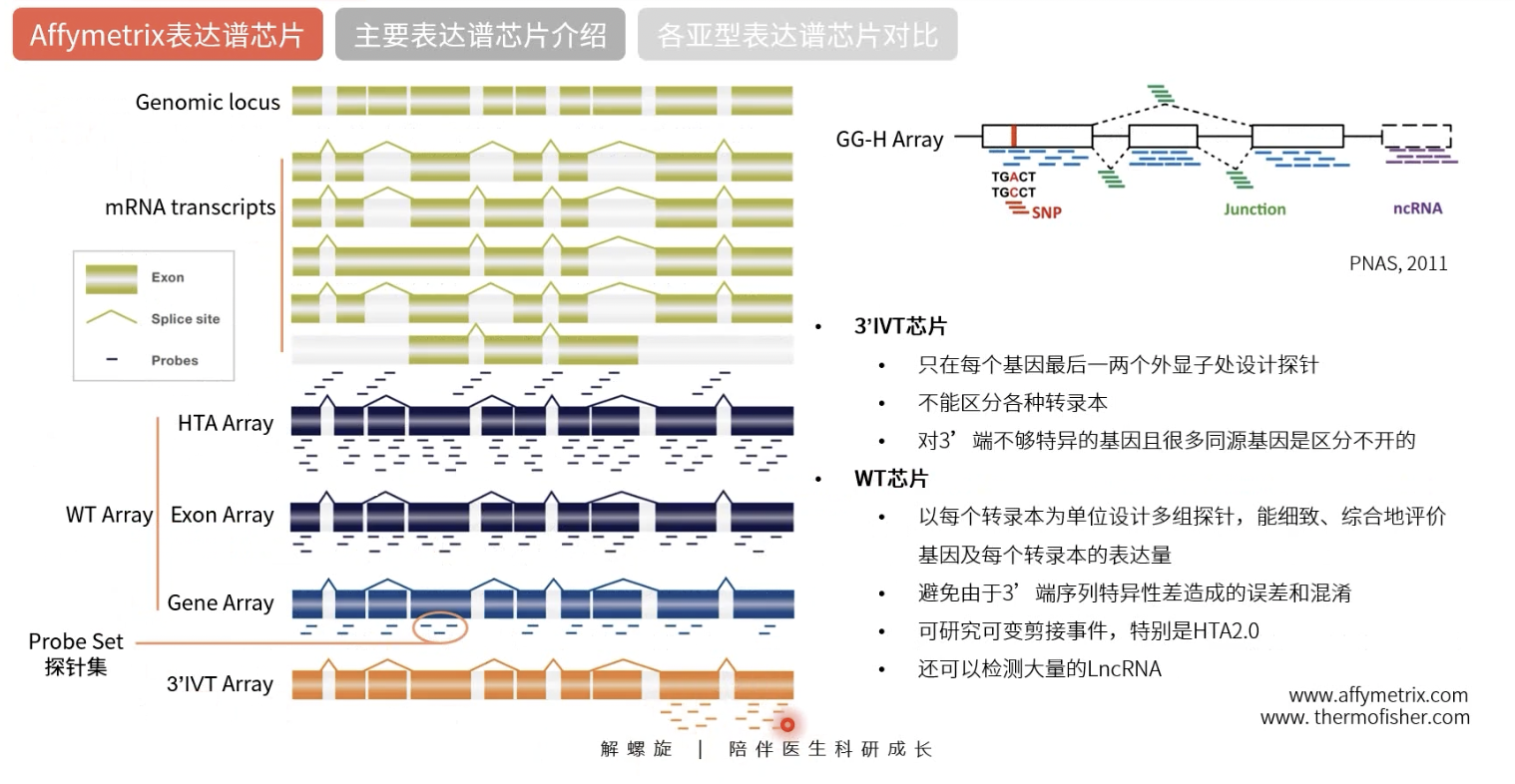
- WT芯片包括了转录组芯片transcriptome array,比如HTA 2.0(H- human),外显子芯片Exon ST Array(如HuEx 1.0 ST,ST-Sense Target,表示检测正义链核酸),基因芯片Gene ST Array(如HuGene 2.0 ST,用于全转录本表达谱和选择性剪切的分析)
- Exon ST芯片探针设计是基于完整的基因组信息,包括已有功能注释的外显子和生物信息学推测的外显子,用于全转录本水平分析和外显子剪接分析,其中探针选择区PSR,对于外显子芯片,每个外显子就是一个PSR,平均有4条探针,并且会组成一个Probe Set,目前Exon ST主要用作转录本水平的研究和可变剪接的分子机理研究,也可用于药靶基因的新型选择性剪接异构体的发现,将可变剪接方式与生物表型变化连接起来
- Gene ST是Exon ST的简化版本,可以同时进行基因水平和外显子水平的检测,每个探针选择区平均2条左右的探针组成一个ProbeSet,对基因表达可以进行更为精确的检测,着重于注释完善,功能明确,研究充分的基因区域;Gene ST在芯片设计时,会尽可能使用Exon ST芯片探针设计的子集,主要用于基因水平表达分析,一般不用于外显子剪接分析
- Gene系列比较基本,可以满足一般基因表达量检测的需求,Exon系列比较全面,检测表达的精确性更高,而转录组芯片,比如HTA 2.0,探针更全,每个探针选择区平均有10条探针组成一个Probeset;而且在外显子连接部位,也就是Gene Junction涉及可变剪切,还专门设计了4条叠瓦排列的探针,因此可以得到最全、最精确的表达量,是研究基因可变剪切的首选芯片
- 传说中的“多合一”芯片:Glue-Grant HTA,有690万探针,基因和转录本表达、选择性剪切、SNP、lncRNA、miRNA全都能分析,在多个序列上优于测序
不同芯片的数据量
- HTA 2.0的csv格式注释文件——1.35GB,100万左右的探针注释probeset,基本别想用笔记本+excel打开编辑,会需要用到R语言来处理
- Exon ST的csv格式注释文件——912MB,100万左右的探针注释probeset,基本别想用笔记本+excel打开编辑,会需要用到R语言来处理
- Gene 2.0——353000左右的探针注释probeset
- Gene 1.0——257000左右的探针注释probeset
- U133 plus 2.0——54000左右的探针注释probeset
- PrimeView——49000左右的探针注释probeset
- U95Av2——12000左右的探针注释probeset
- 推荐初学者试用U133系列及以下水平
Affymetrix非表达谱类型芯片
Tiling芯片
- 或称叠瓦式芯片、嵌合芯片
- 用于寻找和发现新转录本(transcript mapping) ,以及结合染色质免疫共沉淀实验研究蛋白和基因互作(ChIP mapping)
- 根据基因组序列设计探针,如Human Tiling 1.0R 和2.0R,每35bp设计一个25mer探针(间隔10bp),几乎涵盖了目标DNA的全部序列,是目前分辨率最高的基因芯片类型。
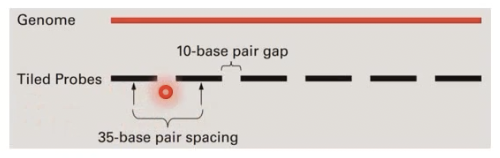
- 由于 tiling芯片是针对基因组序列设计的高密度芯片,因此可以用作全转录组分析,其密集的探针数能保证对整个基因组全转录本的扫描
- Tiling 芯片的另一个作用就是结合染色质免疫共沉淀实验(ChIP)研究转录因子或其他DNA结合蛋白的结合位点分析,以及染色体复制起始位置分析等检测目的。
- 目前Affymetrix公司已经开发了人、小鼠、拟南芥、线虫、酵母等模式生物的全基因组Tiling芯片
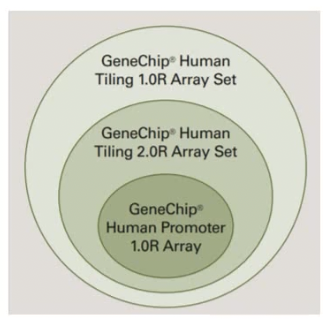
- Tiling芯片探针是最多的,基本上达到了密度的极限,每张芯片 650万探针全基因组tiling 芯片目前有Tiling 1.0R和 2.0R 芯片集Array Set,其中1.0R芯片集由14张芯片组成,是专门为transcript mapping设计的,2.0R芯片集是1.0R芯片集的精简版,共7张芯片组成。专门做 ChIP 的芯片又叫Promoter tiling array,如 Human Promoter 1.0R,它的设计和全基因组tiling芯片不同,专注于启动子部位
- Tiling 1.0R、2.0R和Human Promoter 1.0R三种芯片的关如下图所示
SNP芯片
- 目前Affymetrix最新的SNP芯片是Axiom芯片,基于DNA连接反应
- 在Axiom的芯片当中,CHB1和 CHB2是两款很常用的、针对中国汉族人(Han Chinese populations)的SNP分型芯片,联合起来共含 128万多个SNP位点和3万simple indel位点。
- 而老的卡式芯片,是基于目标DNA 片段与探针序列进行杂交,如经典的 SNP 6.0,有90多万个 SNP位点的探针和94万个拷贝数变异探针。SNP芯片常用来做GWAS研究。
- 如果想做CNV研究,我们除了可以使用上面的 SNP芯片,还可以使用OncoScan 和CytoScan系列
- 此外,Affymetrix接受定制芯片(Made-to-Order Array Program等) ,其中有一款 U133系列的专供石蜡标本表达谱的芯片(X3P Array) ,大家可能经常会遇到
Affymetrix各种芯片一览