GSEA基本介绍
- GSEA,全称是gene set enrichment analysis基因富集分析,是博劳德研究所研究团队开发的一个针对全基因组表达谱数据进行分析的工具,免费注册后即可进行下载
- GSEA分析所适用的主场景之一:它能帮助生物学家在两种不同的生物学状态中,判断某一组有特定意义的基因集合的表达模式更接近于其中哪一种。GSEA是一种非常常见且实用的分析方法,可以将数个基因组成的功能基因数据集与测序及芯片得到的全部数据做出简单而清晰的关联分析。
- GSEA富集分析的特点
- 无需差异基因
- 分析的是基因集合而非单个基因(GO)或少数基因(Pathway)
- 富集分析(见下)
- 将基因与预定义的基因集合(MSigDB)进行比较
- 使用GSEA分析结果,发表文章时,需要注意引用以下参考文献:
- Vamsi K Mootha, Cecilia M Lindgren, Karl-Fredrik Eriksson, et al. Nat Genet.03 Jul;34(3):267-73
- Subramanian A1, Tamayo P, Mootha VK,et al. Proc Natl Acad Sci U S A. 2005 Oct 25;102(43):15545-50. Epub 2005 Sep 30
如何理解富集分析
- 单基因分析:把实验组和对照组进行高通量测序或基因芯片检测获得的数据直接进行比对分析,发现基因表达发生了变化,到此为止就是单基因分析
- 单基因分析没有考虑基因间的相互作用,因此很难对基因的表达变化作出解释
- 将获得的两组数据进行一定处理后与按先验知识归类的基因集合比对分析,将某个干预和某个生物学功能变化联系起来,这个过程就叫富集分析
GSEA原理
GSEA分析的文件准备
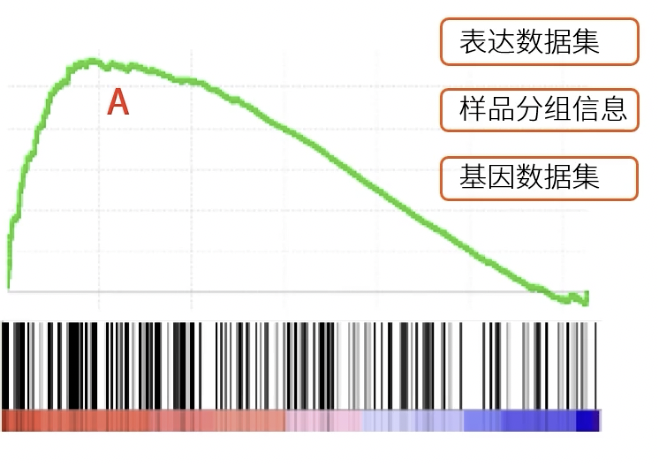
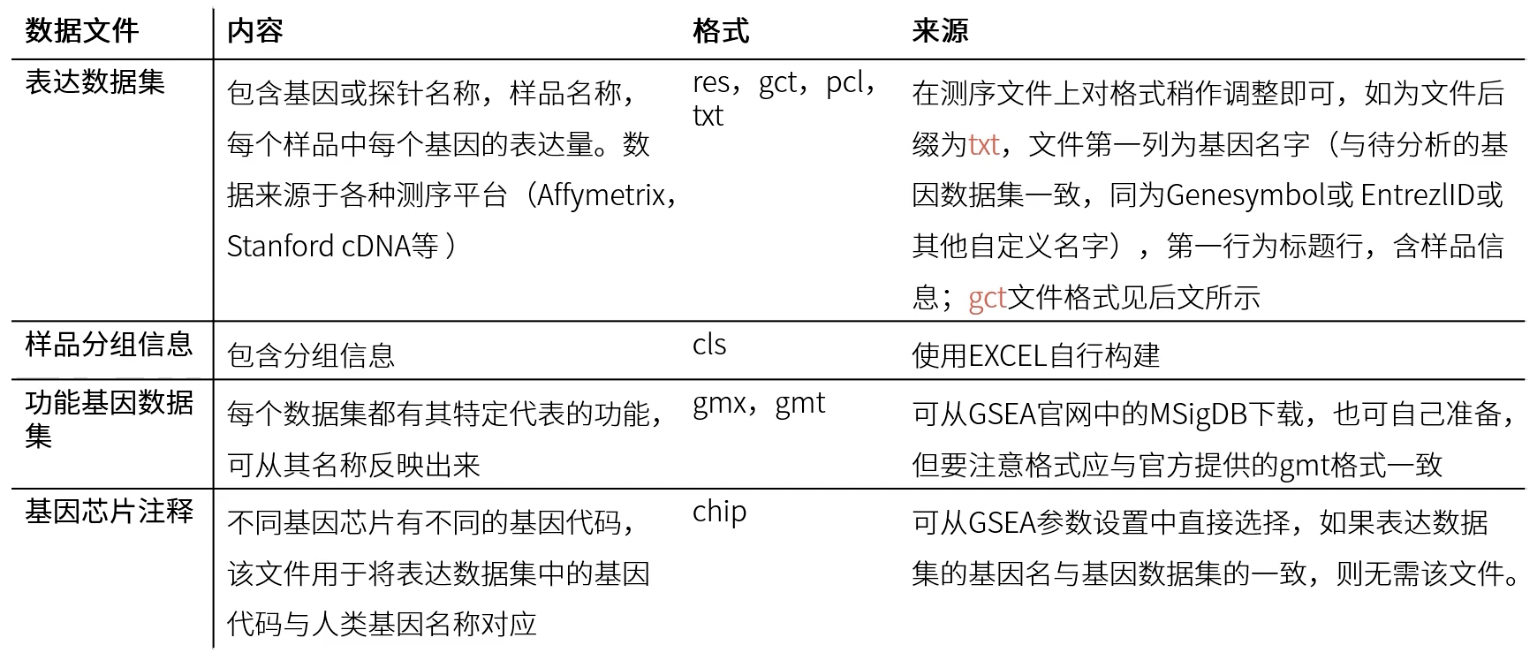
表达数据集
- 测序或芯片获得的表达谱信息,需要按照表达的丰度表示
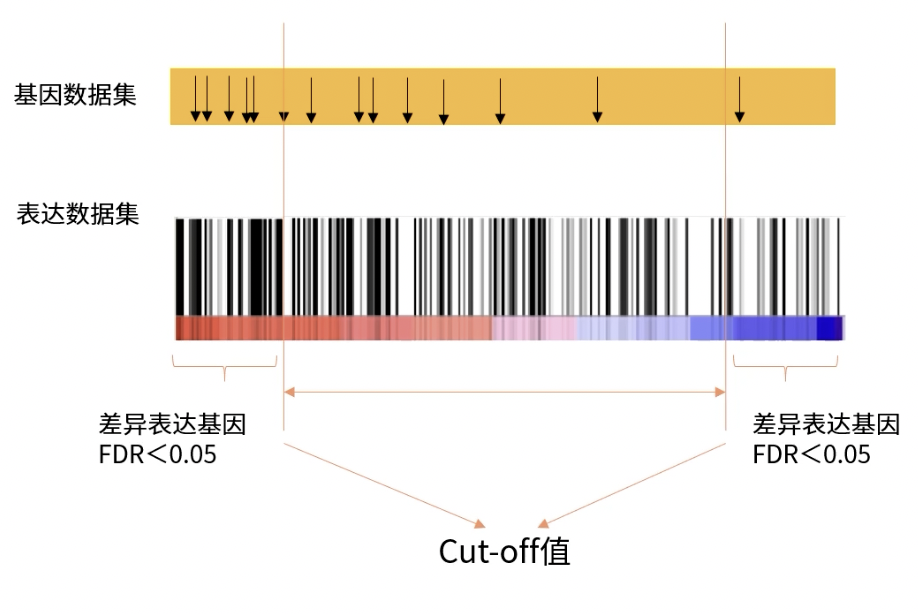
- 功能基因数据集中,出现在表达数据集当中的基因所处的位置,需要用黑色的竖线表示,如下图所示

样品分组文件
基因数据集
分析结果判读与关键概念
- 富集得分/富集分数ES:从排序后表达数据集的第一个基因开始,如果表达数据集出现在功能基因数据集中就加分,不在功能数据集中就减分;ES是一个动态变化的值,其峰值为最大富集分数
- 富集得分ES在计算过程中中不断加分或减分,是一个动态的数值,一般的数据图片中往往不呈现ES值,有的话也是最大ES值
- 最大富集分数的含义:
- 如果最大富集分数为正值,说明富集结果在顶部富集,功能基因数据集中的基因在表达数据集中高表达
- 如果最大富集分数为负值,说明富集结果在底部富集,功能基因数据集在表达数据集中低表达
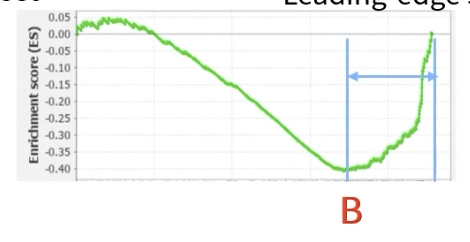
- 如果随机分配,说明表达数据集与功能基因数据集中对应的表型没有关系
- 图中的绿色曲线是ES的变化,绿色曲线的高度体现的是最大富集分数
- 核心基因Leading-edge Subset:最大ES前对应的基因,指的是对富集得分贡献最大的基因成员,
- 校正后的富集得分NES:常数,比较的是表达数据集在不同功能基因数据集中的富集程度
- NES的计算公式:某一功能基因集合的ES/所有基因集合随机组合得到的ES平均值
- 名义p值(Nominal p-value或NOM p-value)
- 这个值描述的是针对某一功能基因子集得到的,富集得分的统计显著性;p越小,富集性越好
- 因为没有进行功能基因子集大小和多重假设检验校正,所以是名义p值,一般需要p<0.05
- 错误发现率(False Discovery Rate或FDR q-value)
- 这个指标进行了功能基因子集大小和多重假设检验校正
- 这个指标描述了估算的可能性,也就是一个功能基因子集的NES中包含的错误的阳性发现率
- 举例:FDR=25%说明在特定的ES值下,4次中就有可能出现1次错误
- 一般要求FDR<25%,且要同时关注FDR和p值
- 总体错误率(Family-Wise Error Rate或FWER p-value)
- 是使用Bonferonni校正后的名义p值,在多重假设检验中广泛使用的错误控制指标
- 用于在检验出尽可能多的候选变量的同时,将错误发现概率控制在一个可以接受的范围内
GSEA与传统富集分析的区别
在原理上,GSEA与GO分析有很大的区别
- GO分析针对的是差异基因,也就是看重差异表达的基因在哪一些GO分类或者Pathway当中富集,从而说明这些差异基因影响了哪些基因或Pathway
- GO关注的是差异基因在生物学过程BP,分子功能MF和细胞组成CC中的富集定位,通过计算出差异基因每个GO terms的p-value、q-value和FDR值,从而对基因进行注释和分类
- KEGG通路分析的思路与GO富集分析相似,也是筛选出差异基因,通过统计学分析,判断差异基因可能与哪些信号通路相关
- GO分析和KEGG分析都需要人工设定cut-off值来筛选出差异表达的基因,也就是都会忽略对结果(可能有)贡献,但是没有落在差异显著范围内的基因
- GSEA不需要指定明确的差异基因阈值,算法利用的是测序或芯片获得的,全基因组表达谱,根据实际数据的整体趋势,在表达谱整体层次上,对数条基因进行分析;因为不需要指定差异基因的阈值,所以得出的结果会更加可靠。
GSEA操作流程简介
软件下载
数据准备
表达数据集:以gct格式为例

功能基因数据集
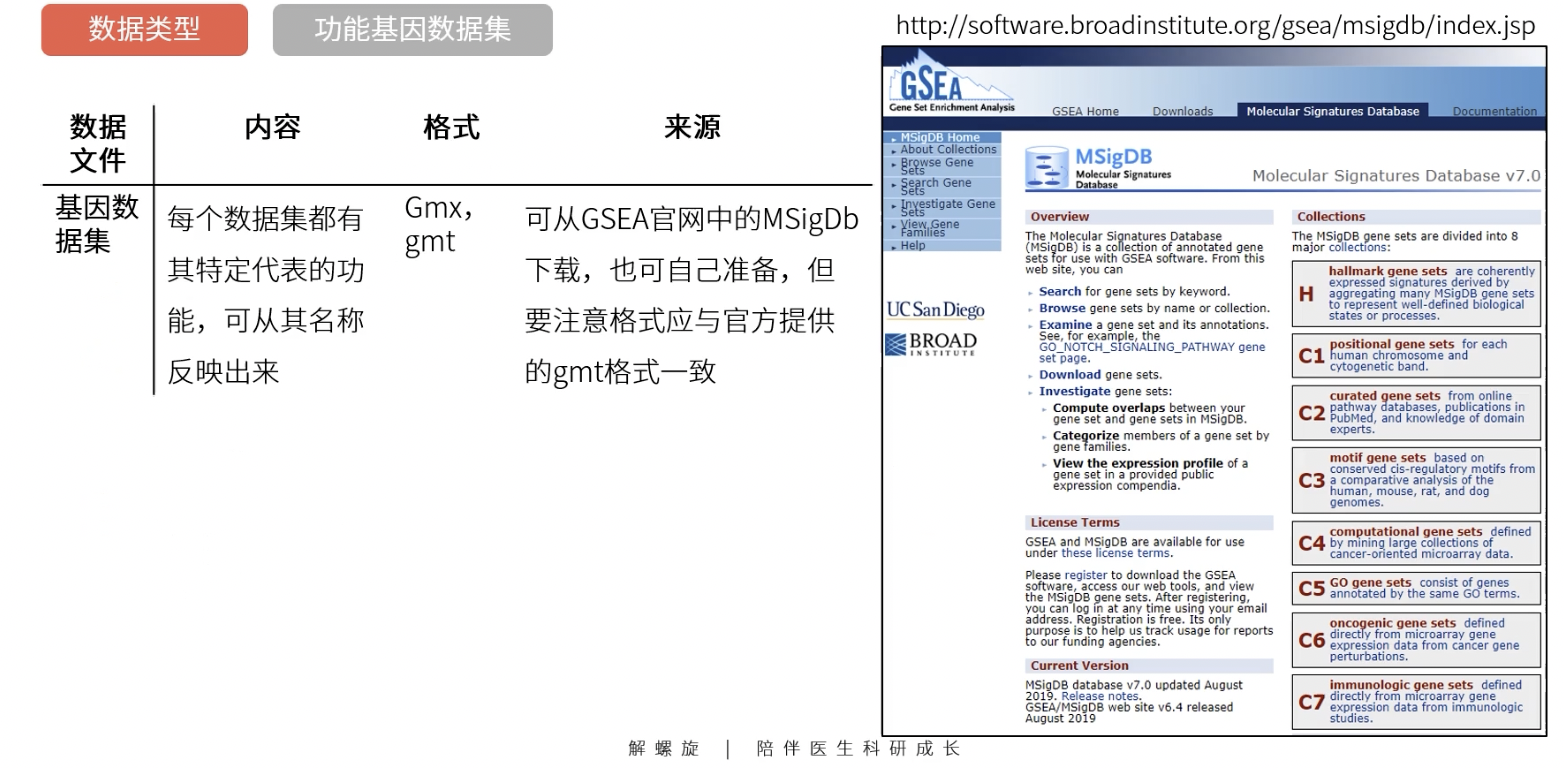
- gmt文件需要在GSEA网站中下载
- MSigDB将基因分成了各个功能表型群,但是只包含了人的基因序列。
基因芯片注释
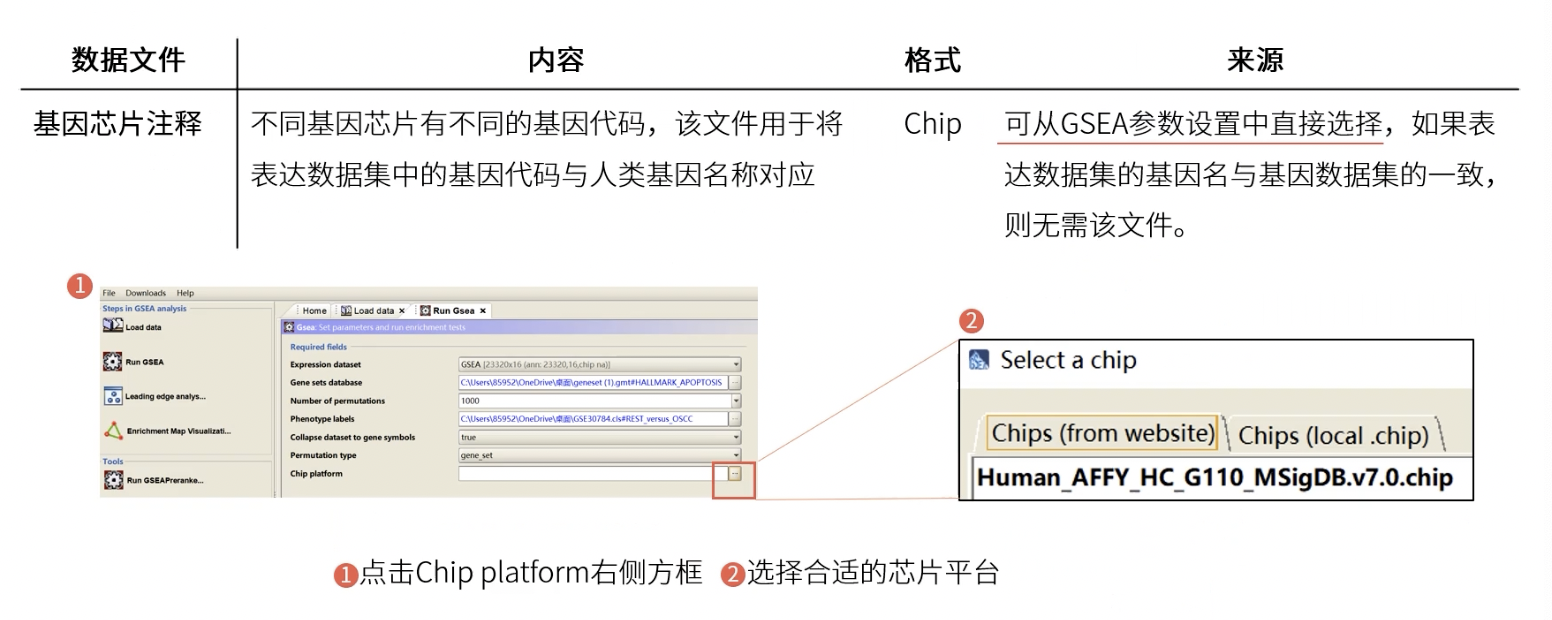
参数设置和软件运行
结果分析
结果分析概述
- 结果有意义的标准(同时满足)
- FDR<0.25
- p-value<0.05
- |NES|>1
- 名义p-value没有经过校正,而FDR值经过了功能基因子集大小和多重假设检验校正,因此这两者都要关注
- p值很小而FDR值很大提示富集不显著,有以下几种可能的原因
- 用于基因芯片分析的样本量很小
- 杂交信号弱
- 所选的基因子集没能很好地反应样本的生物学意义
表格型数据的分析
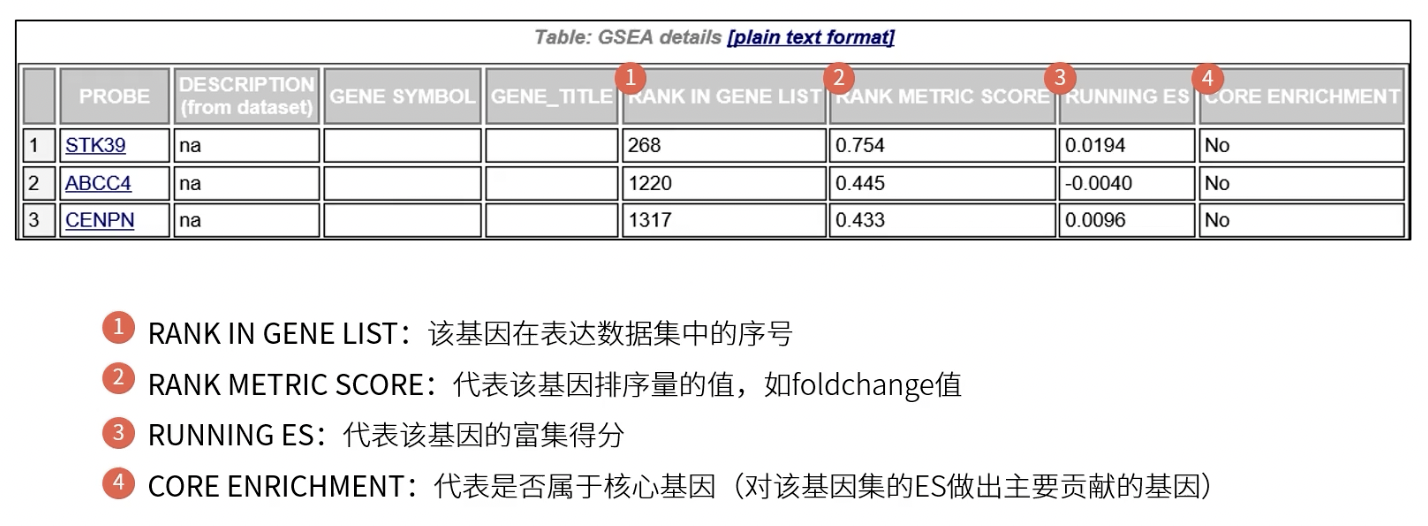
图片型数据的详细分析
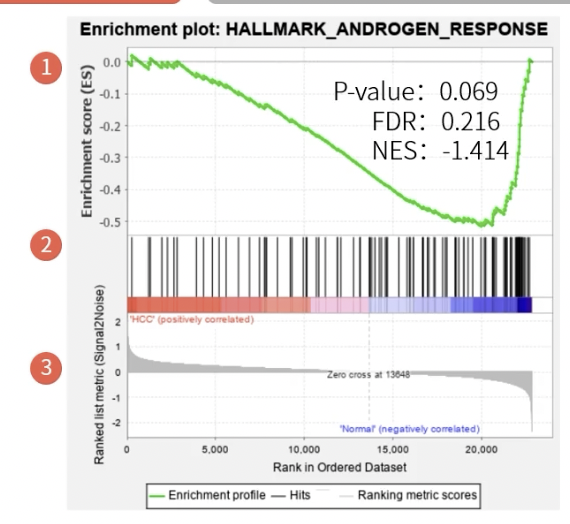
- 第一部份:ES折线图,横轴为表达数据集中的每一个基因,纵轴为对应的RunningES,峰值即为该基因集的最大ES值,当折线图为负向时,峰值右边基因为核心基因;当折线图为正向时,峰值左边基因为核心基因(如下图所示)
- 第二部分:基因位置图,表达数据集按照表达丰度(热图)排列,该功能基因数据集出现在表达数据集当中的,基因所处的位置会用黑色淑贤表示
- 第三部份:所有基因的Rank值分布图,采用的是Signal2Noise算法