DAVID数据库
DAVID数据库简介
- DAVID是一个生物信息数据库,也是一款免费的在线软件,可以为大规模的基因或者蛋白列表提供系统综合的生物功能注释信息,帮助用户从中提取生物学信息
- 目前,DAVID数据库主要用于差异基因的功能和通路富集分析,对于很多科研工作者来说,是非常好用的工具
- DAVID数据库官网:https://david.ncifcrf.gov/content.jsp?file=release.html
- 发表文章需要引用作者的文章,Huang DW, Sherman BT, Tan Q, et al.DAVD Bioinformatics Resources: expanded annotation database and novel algorithms to better extract biology from large gene lists. Nucleic Acids Res. 2007 Jul;35(Web Server issue):W169-75.
DAVID数据库功能介绍
- DAVID和其他的分析工具一样,都是将输入列表的基因关联到生物学注释term上,进而通过统计学方法找出最显著富集的生物学注释
- 复习
- GO的全称是Gene Ontology,即基因本体论
- GO分析是指蛋白质或者基因,可以通过ID对应或序列注释的方法找到与之对应的GO号,而GO号可以对应到相应的Term
- KEGG pathway分析与GO相似,主要分析基因产物在细胞中所在的通路
- DAVID数据库的功能
- 基因功能注释functional annotation:对输入的基因列表中的基因进行功能注释
- 基因功能分类gene functional classification,该工具采用全新的模糊聚类算法,能够将功能相关的基因聚为一个单元,结果的分支越高,说明该基因在基因列表中越重要
- 基因ID转换gene ID conversion:实现不同数据库的基因标识之间的转换,包括NCBI,PIR和Uniprot/SwissProt等。
- 基因ID对应基因名称gene name batch viewer:能够显示基因ID和对应的基因名称,相关基因以及所属物种,让研究者初步判断基因列表是否符合研究目的
GO基因功能注释分析
上传数据Upload
- Enter Gene List:
- 输入我们准备好的差异基因列表,注意输入的不能是单个基因,这样是富集不到有意义的通路或功能的
- gene list不能超过3000个基因
- 输入格式:每行一个基因名(推荐),或基因名用逗号隔开
- Select Identifier:选择”ENTREZ_GENE_ID”
- List Type:选择”Gene List”
- 点击”submit list”
List页面
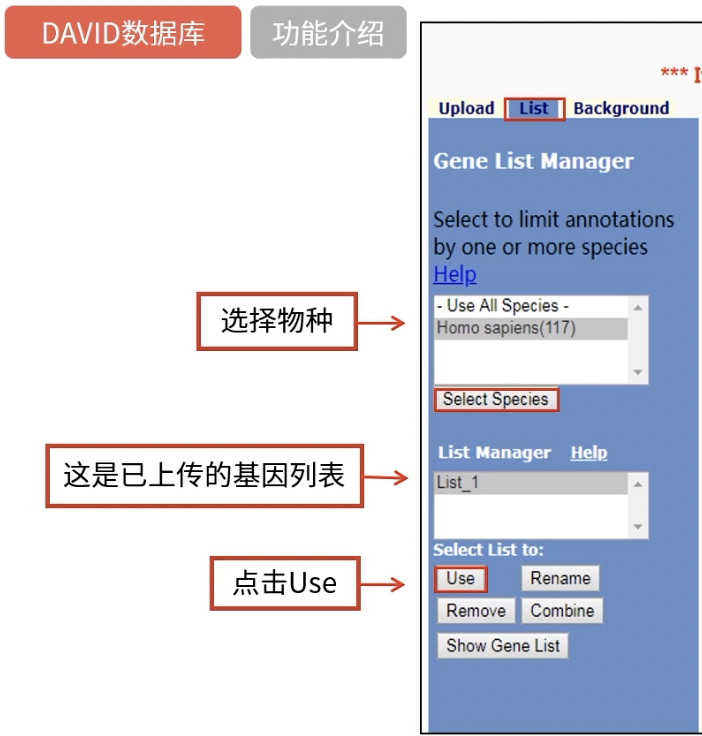
选择Background
- 原则是必须构建一个足够大的,研究者可能涉及的所有基因的集合,本节课选择默认的,人的全基因作为背景,也可以在之前的Upload环节中
开始分析
- GO富集分析可以粗略了解差异基因富集在哪些生物学功能、途径或者细胞定位。
- 对差异基因进行KEGG pathway分析,可以了解实验条件下显著改变的通路,在机制研究中显得尤为重要。
- 功能注释的三个工具
- Functional Annotation Clustering:使用模糊聚类方法,对被注释上的 Terms做聚类,即Terms被分成多组,并将给出聚类的分值。分值越高,代表该组内的基因在基因列表中越重要。
- Functional Annotation Chart:提供gene-term的富集分析,这是我们今天主要用到的工具。
- Functional Annotation Table:该工具实现了基因的功能注释,将输入列表中每个基因在选定数据库中的注释以表格形式呈现。
Functional Annotation Chart
- 点击”Download File”,把目标链接以txt文本格式下载下来,可以考虑把文件命名为GO,用excel可以打开
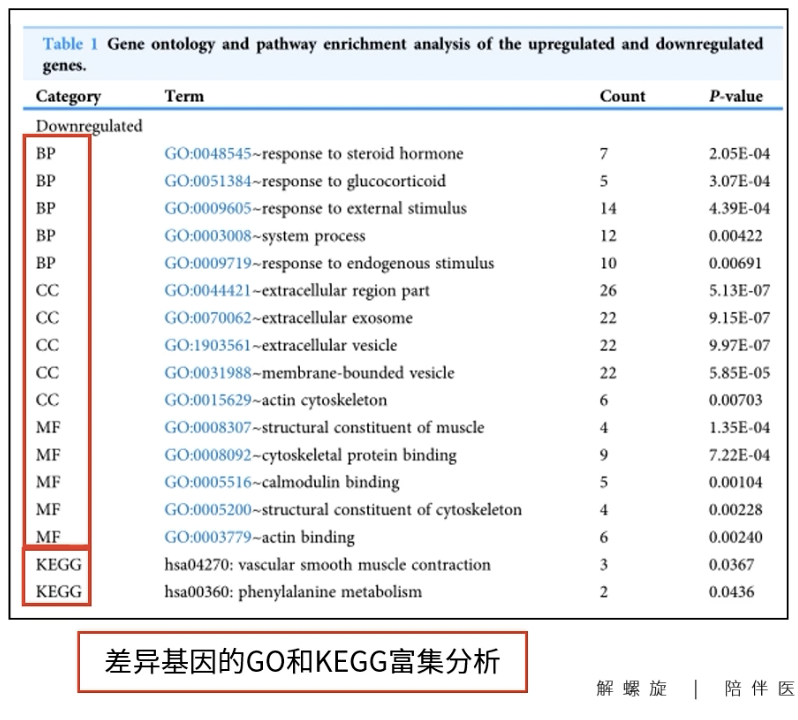
- 我们主要关注表格中的”Category””Term””Count””PValue””FDR”五项,其他列删除
- 对P值进行筛选,要求P<0.05(也可以对FDR值进行筛选,标准同样是p<0.05),然后对p值进行-log10转换,完成后需要将格式转换成文本
- 然后在term后新增一栏,命名为新的term,这一栏需要用MID函数(字符串,起始位置,保留字符的个数)将波浪号以及之前的内容删除。
- 保留字符的个数可以非常多,比如100,以完全保存我们想要的字符的个数
- 之后以新增的term和count做柱状图,为了让图比较好看,我们可以对count进行降序排序
- 也可以用term和-log10做图,方法同上
- 还可以用category,term,count和pvalue四项做表格,这个表格也可以放在文献中

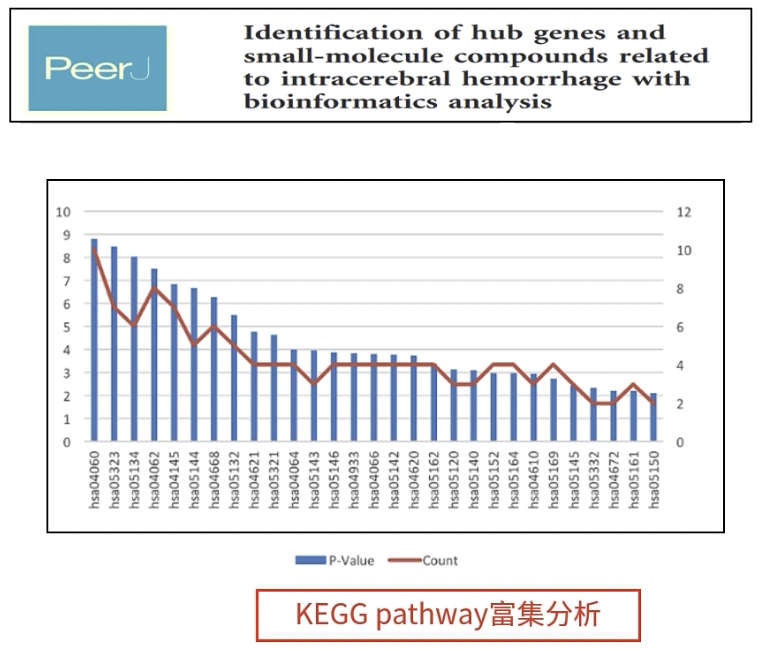
KEGG pathway分析
- 清除之前的选项后,点击KEGG_PATHWAY和functional annotation chare
- 出现的表格与前面的类似
- 点击Term的内容,可以查看具体的信号通路,其中红色标注的就是我们自己输入的差异基因
- 有时我们会得到几十条数据通路,这时需要我们根据收集到的p值和募集到的分子数量进行筛选,选择一条或几条通路
DAVID数据库应用实例
单基因列表分析流程
- 分析步骤
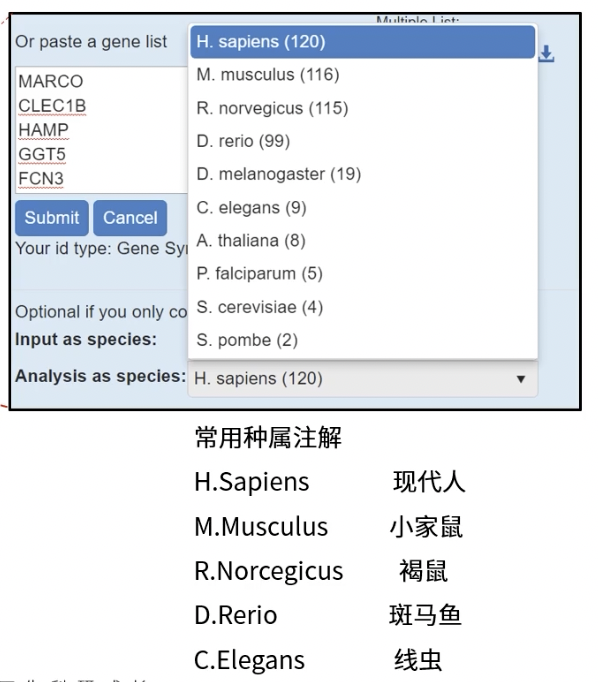
- 录入基因列表
- 选择种属信息
- 可选的
- 快速分析/定制化分析
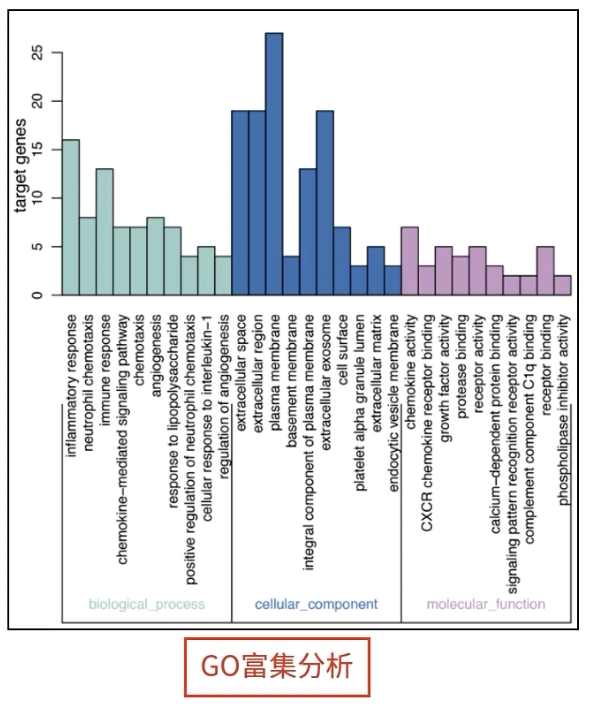
条形图
- 以-log10(Pvalue)进行填色,颜色越深,富集程度就越高
基因列表
基因注释
通路和富集分析
蛋白-蛋白互作分析
多基因列表分析流程(没用到,没学)
文献实例