选择的文献
- 如何利用泛读技巧,快速了解文章的论证套路
- 本文选择的例子都是IF<5的单变量研究,属于入门文章
补充:关于EMT的基本知识
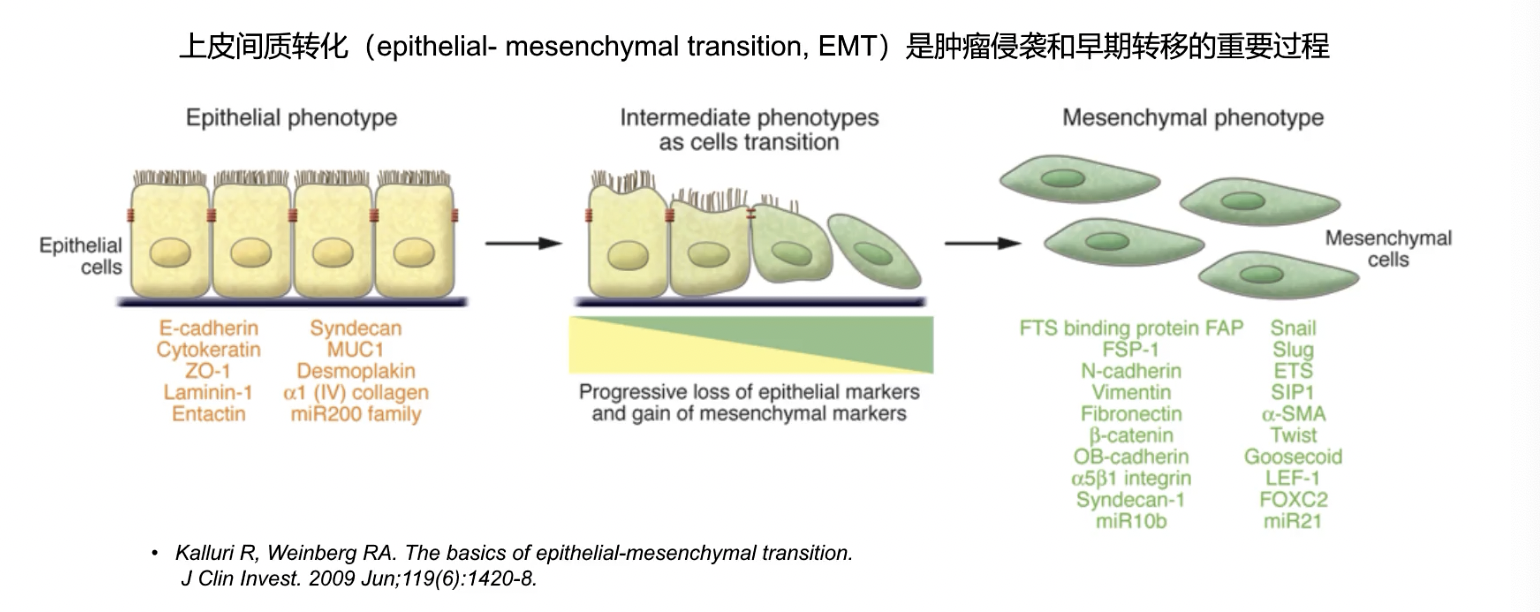
- EMT的重要标志物:E-cadherin(上皮),N-cadherin(间质),Vimentin(间质)
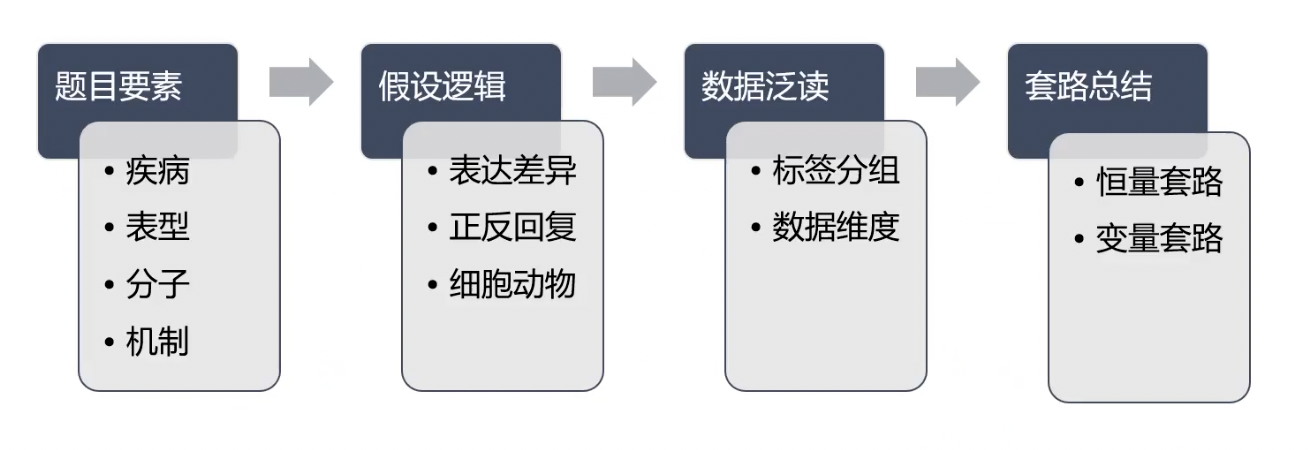
文章泛读的步骤
读题目
- 典型的题目应该具有疾病、表型、分子、机制四个要素,题目完全满足四要素时,不需要看abstract
- 当题目完全满足四要素时,不需要看abstract;否则,就需要阅读abstract来还原作者的科学假设
反推假设
- 根据变量论证的基本原则,我们在分析数据图表之前,要推测作者可能的行文逻辑
数据泛读
- “看图说话”的过程
- 直接跳过文章的Methods和Results部分,快速阅读,观察数据标签,横纵坐标分别代表什么
- 分组变化
- 区分数据的维度:数据的来源是组织、动物、细胞、分子
总结套路
- 恒量:表型用什么方法研究,检测的标志物有哪些
- 变量:单因素如何论证、多因素如何论证
文章1
- 科学假设:在「疾病」中,「分子」经由「机制」介导了「表型」
读题目和反推假设

- 没找到机制——乖乖去读abstract吧,但是这里面也没写,所以文章很可能没做机制,对于IF<3的文章,确实有很多没有机制研究
- 根据关键信息推测作者的科学假设(零机制的单变量研究)

- 根据单变量研究套路,反推假设,预测作者的行文逻辑
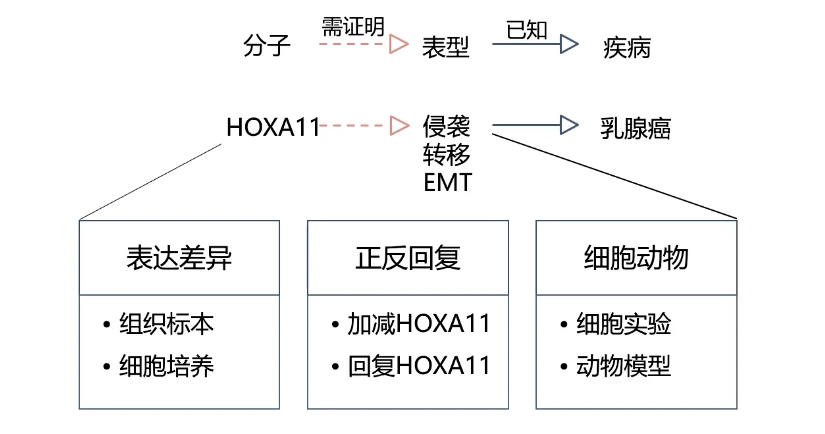
- 文章图片数量的要求:4-8个(大图),有些杂志社要求不能超过6个,通常前面的图是基础表达差异和细胞水平的图,动物和机制一般在下半场
读图片
- 看图片的时候要看图片的注释Figure Legends
Fig.1
- 看到expression就应该反应过来图片记录的是表达差异,数据维度是tissues和cells,还要关注实验方法
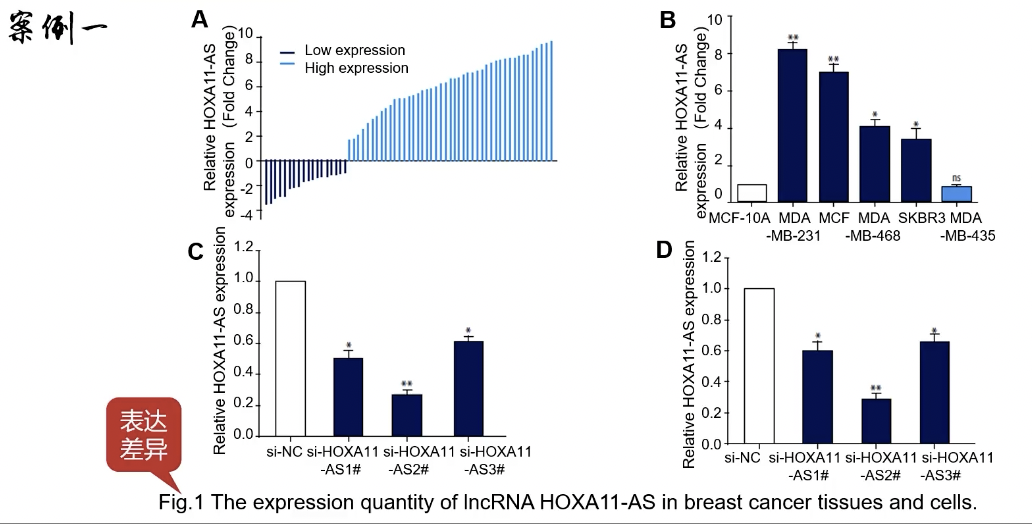
- Fig.1B可以看出是细胞维度上的实验,因为1)FigureLegends里面有cells;2)横坐标的名字是一堆乳腺癌研究的“经典细胞系”
- Fig.1C-1D:si-small interfering RNA,NC-negative control,不同的AS-不同靶点的siRNA,在设计siRNA的时候,为了避免脱靶,经常会设计针对多个靶点的多条siRNA,所以这两个图都是操作验证
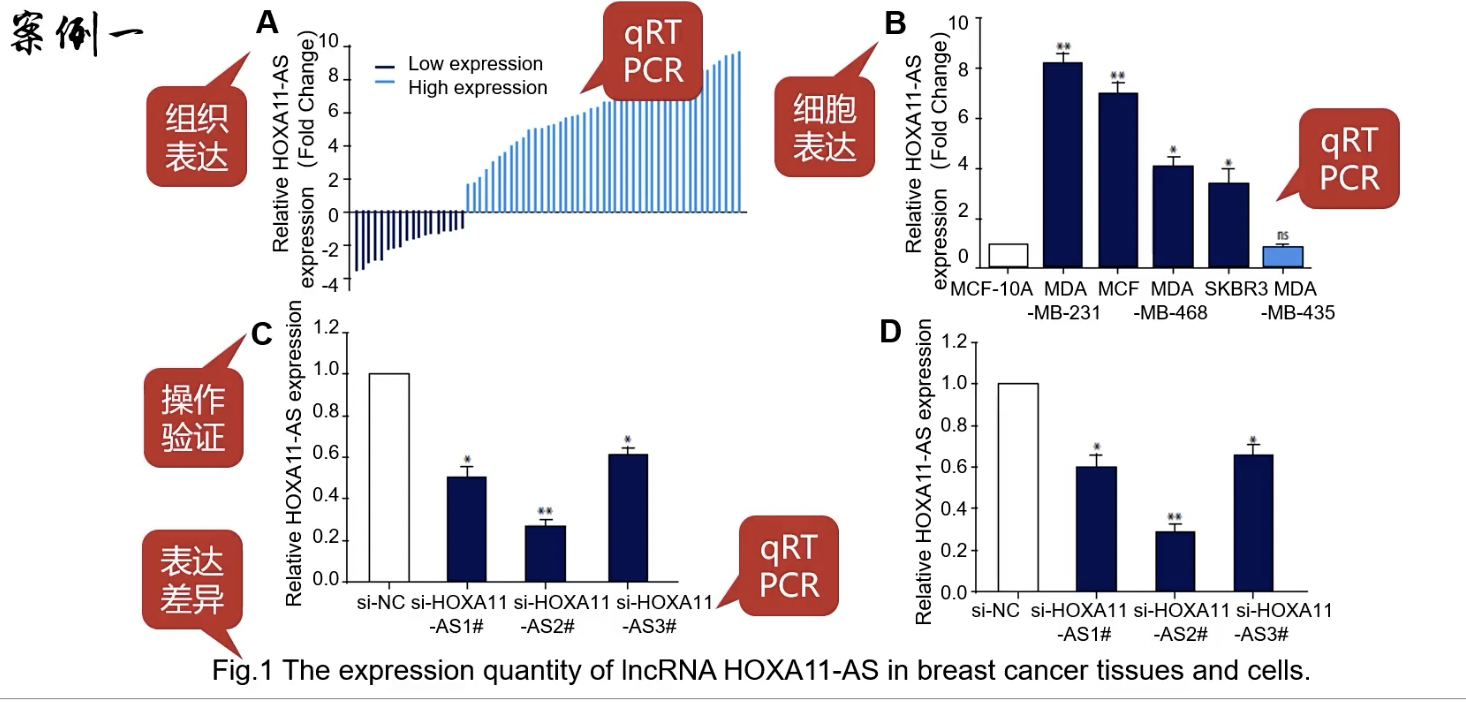
Fig.2
- 细胞表型的实验,涉及的细胞表型是增殖
- Fig.2A-B 分组是黑色的对照和蓝色的敲减(AS)
- Fig.2C 克隆形成的直观实验
- Fig.2D 克隆形成的统计结果,说明敲减了分子之后细胞增殖降低了
- 对于图形类结果的统计结果:一般是一个典型图+对应的统计图(统计似乎是半定量,只负责反映趋势),克隆形成是这样,western和IHC也可以是这样
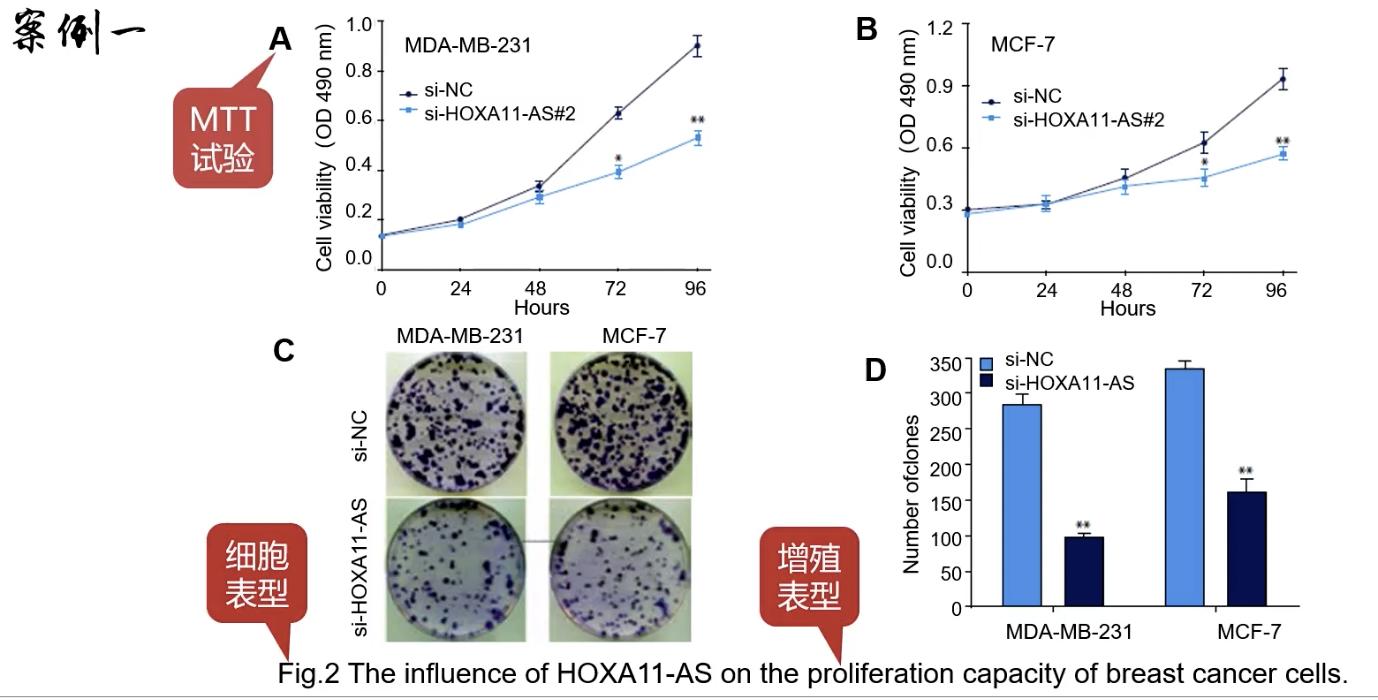
Fig.3
- biological function of breast cancer cells——细胞表型的结果
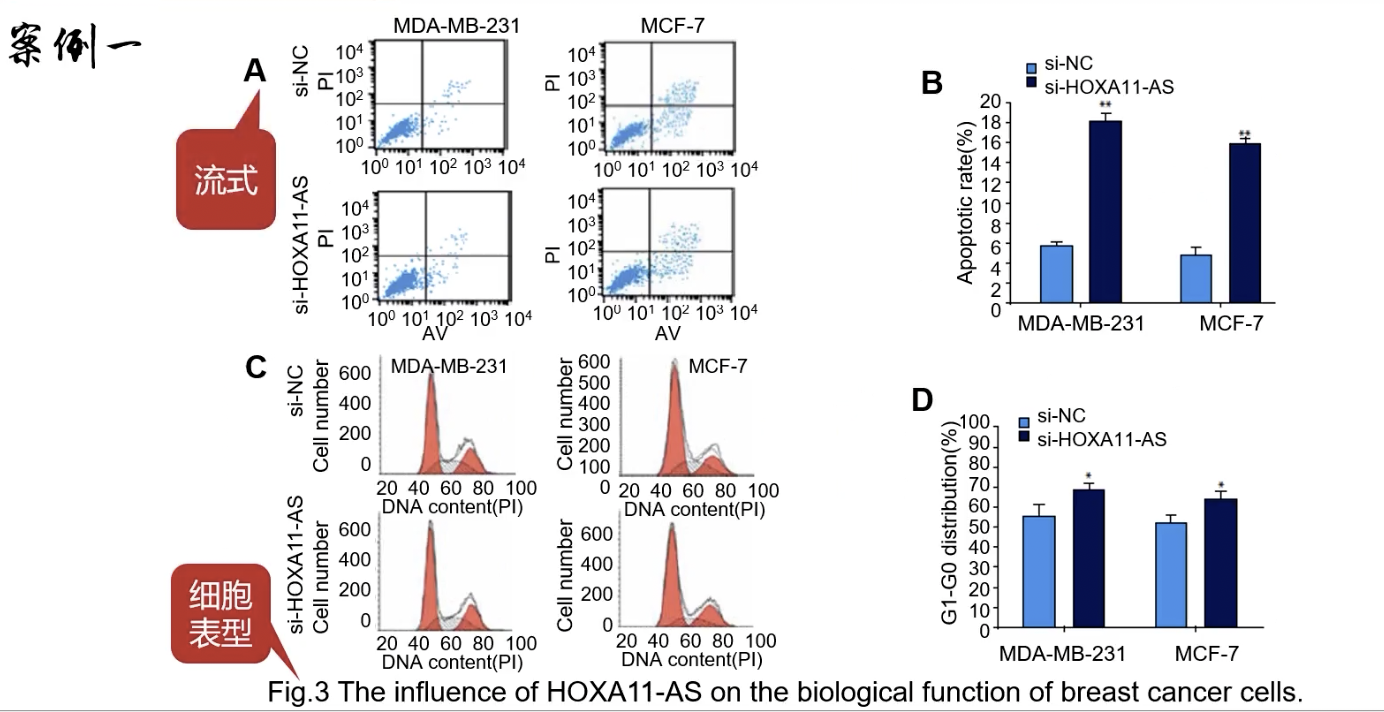
- Fig.3A-B:一个典型的流式结果,图B的纵坐标提示做的是凋亡相关的表型
- Fig.3C-D:另一个典型的流式结果,统计结果对应图D,纵坐标为G1-G0期的细胞分布-提示在做细胞周期相关的实验
Fig.4
- cell migration and invasion capacity-迁移和侵袭表型,仍然是两个经典的细胞系-细胞表型
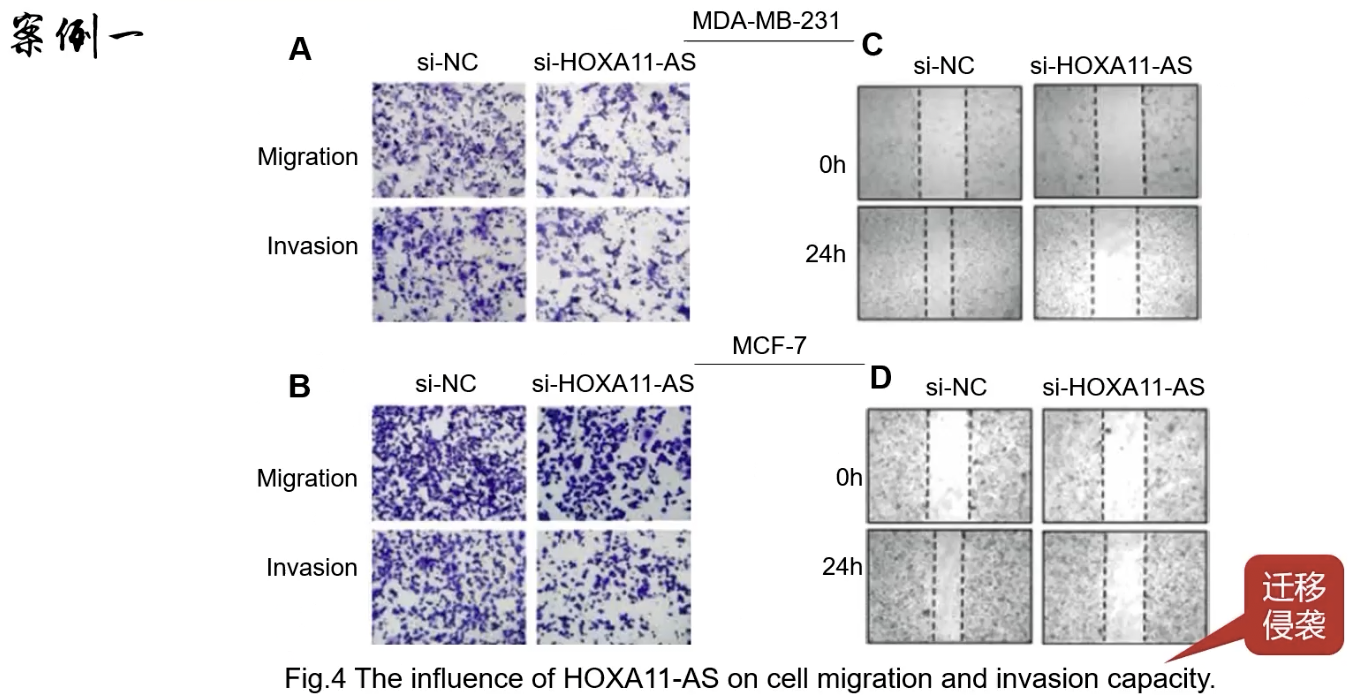
- 两个经典的,用来研究肿瘤和迁移表型的实验:Transwell assay和Wound healing assay(划痕实验)
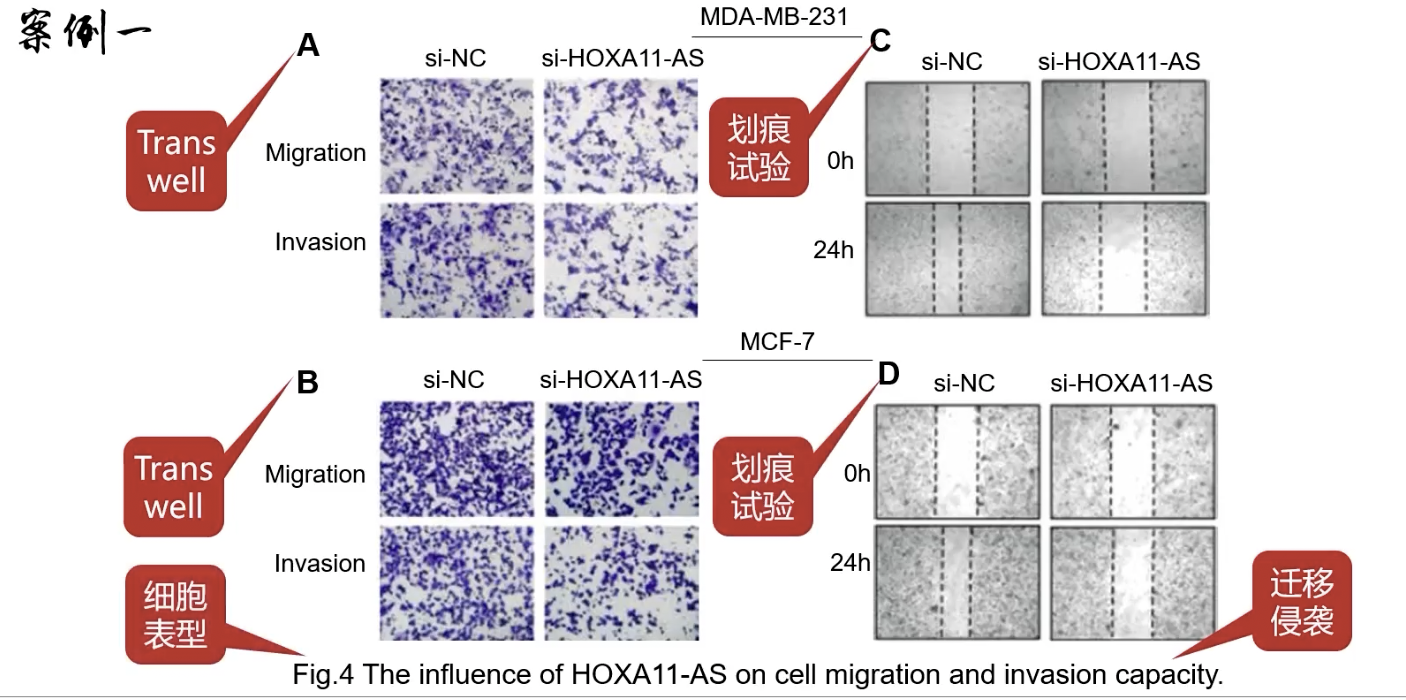
- 优点:另一组细胞水平的,关注别的表型的实验;缺点:缺少统计图
Fig.5
- 明显是动物水平的结果(Fig.5A是肉眼观察的组织标本/Legends里面的in vitro有提示
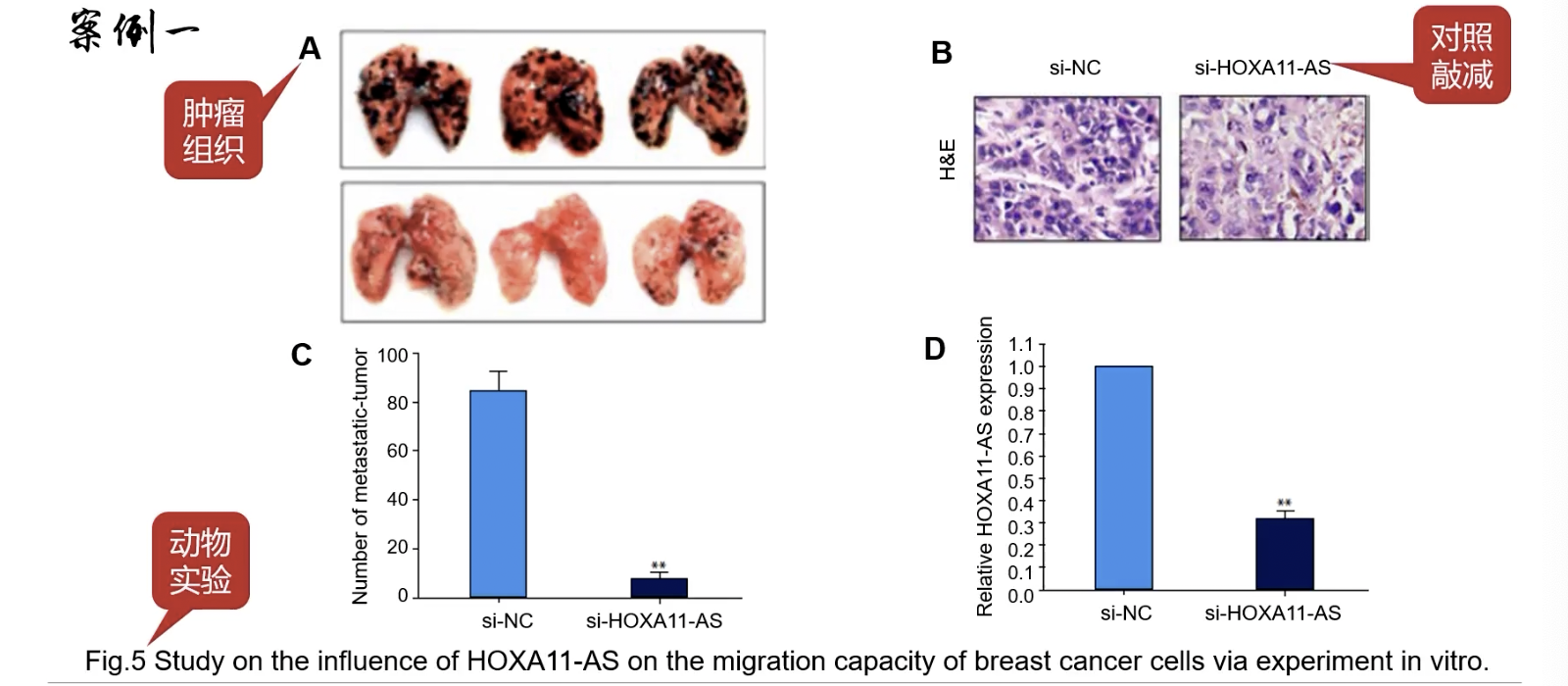
- Fig.5B是HE染色的代表图,是造模的验证过程,验证细胞移植到小鼠体内成功了
- Fig.5C是转移瘤的统计结果
- Fig.5D是通过qPCR验证的,敲减的结果,可以看到敲减后的HOXA11-AS的表达量显著降低了
Fig.6
- 细胞表型,研究的是HOXA11-AS和EMT之间的结果
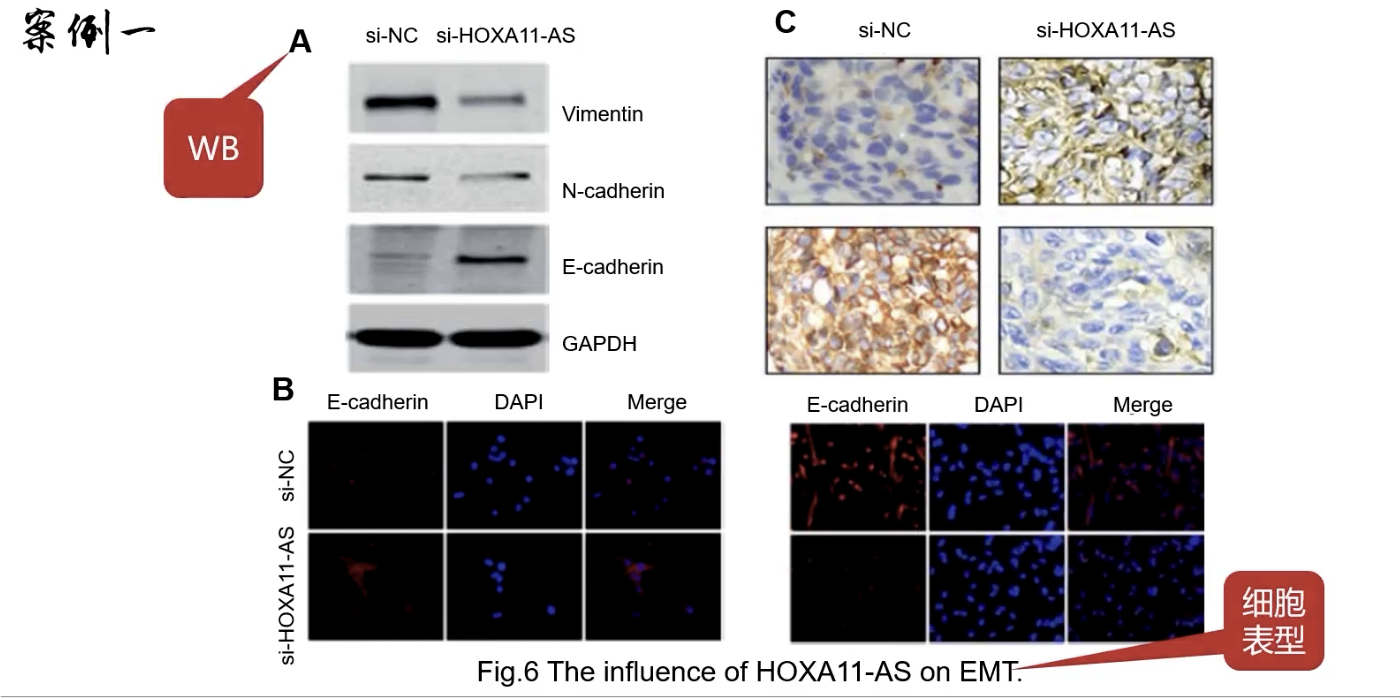
- Fig.6A:western的结果,上皮表型的E-cadherin增加了,间质表型的Vimentin和N-cadherin减少了,提示发生的上皮-间质转换过程减少了
- Fig.6B:免疫荧光的典型结果,A图的验证
- Fig.6C:IHC的代表结果,没有统计数据
总结
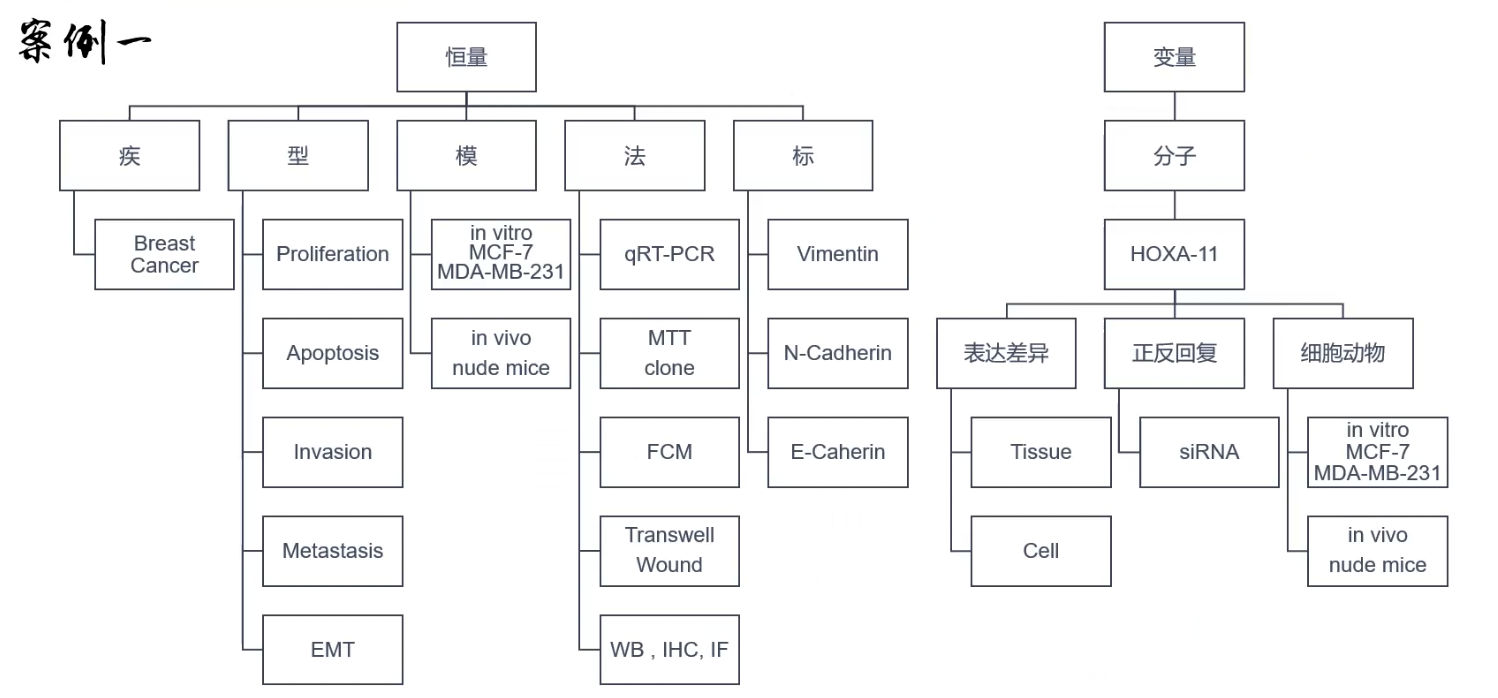
- 单变量的研究,论证套路上只有干扰,没有过表达和回复
- 数据维度:组织、细胞、动物、没有分子机制(有很多表型和对应的数据),走的是“加量不加价”的灌水路线
文章2

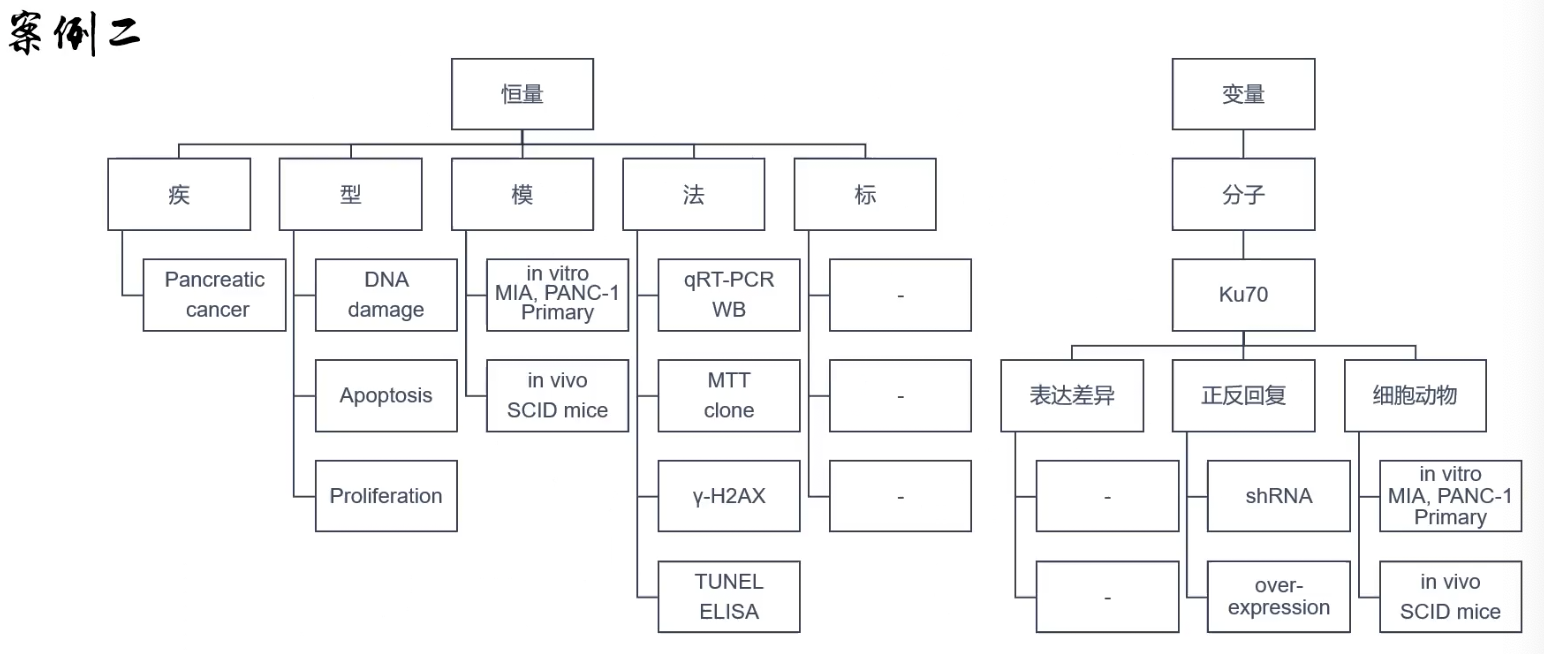
- 又是一个题目里没有机制的文章,可能是一个单变量零机制多表型的“灌水”研究,当然也可以把这篇文章视为二元变量的研究,因为题目中有gemcitabine,也就是吉西他滨这个药物,这个药物是用来诱导表型的;可以说这篇文章是二元变量,但是因为药物只用来诱导表型,所以是“伪二元”
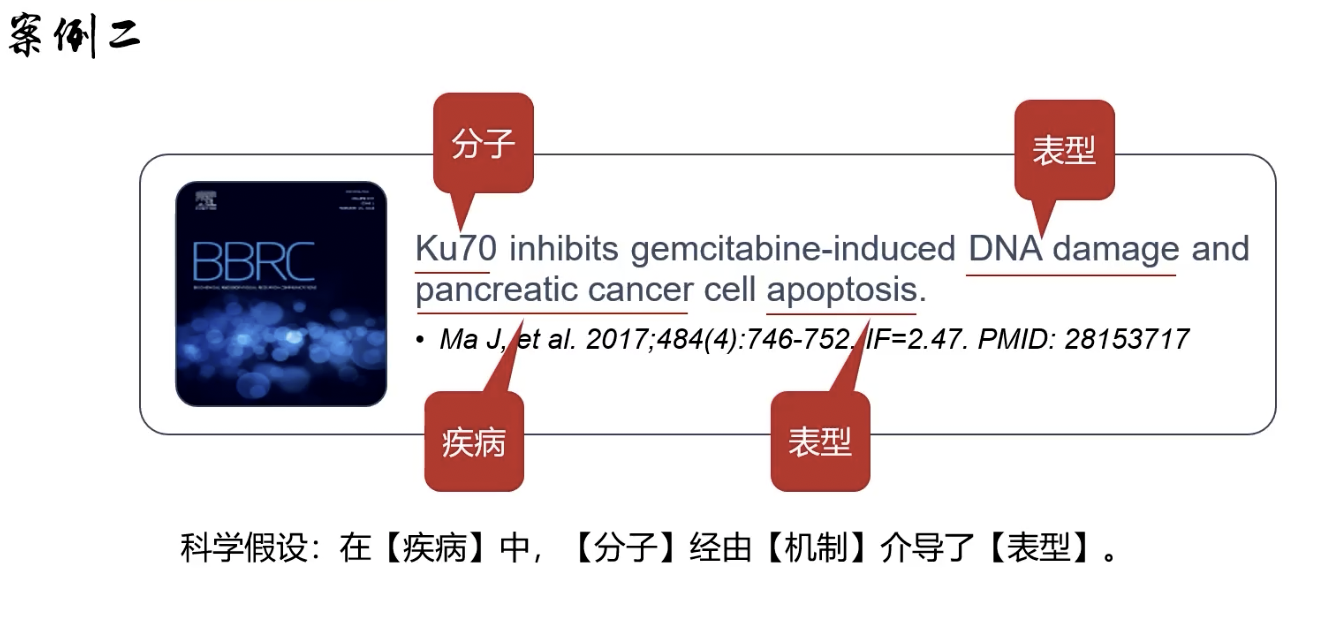

反推作者的假设逻辑
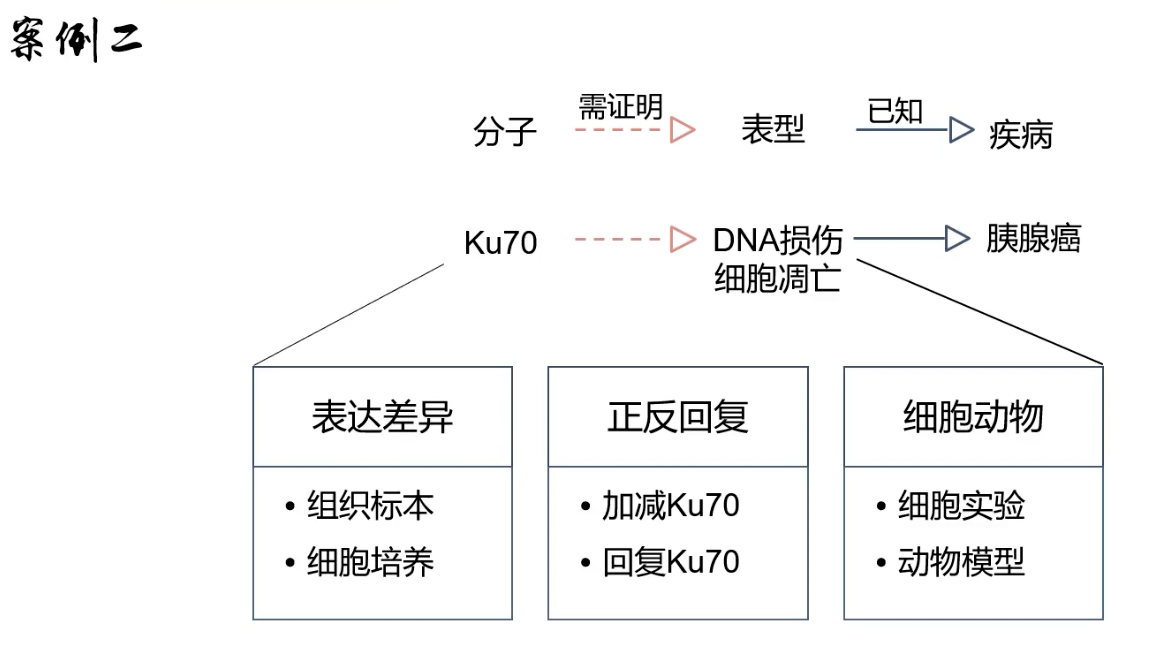
- 检测Ku70的基础表达差异
- 对Ku70用基因操作进行高表达和沉默,观察相应的表型变化
- 补充动物实验进行验证
读图片
Fig 1
- 细胞水平的敲减实验
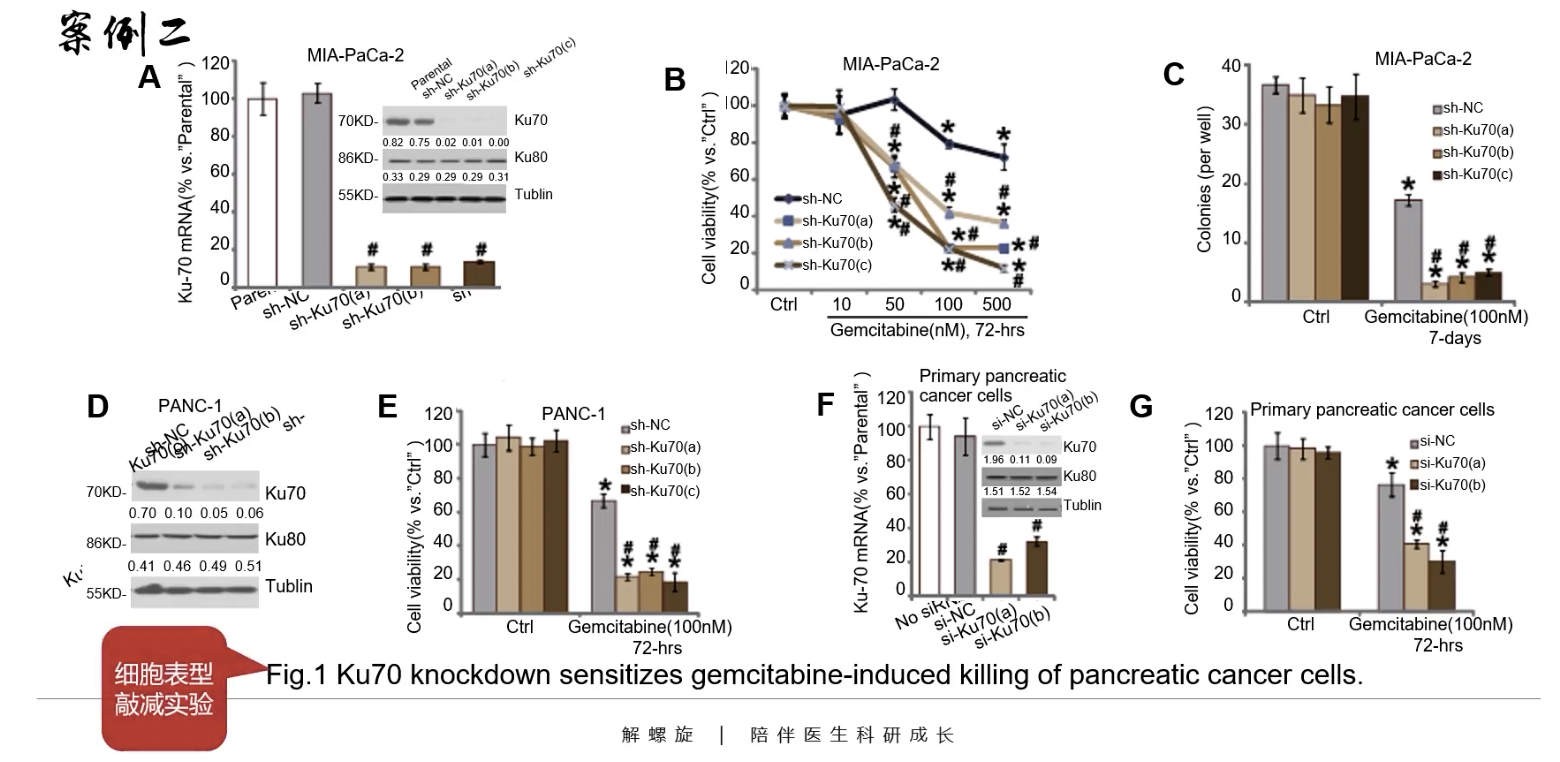
- Fig.1A大图和小图:敲减了Ku70之后的分子操作验证,对于敲减,作者做了a/b/c三个靶点相互验证,在蛋白水平还提供了western的操作结果
- Fig.1B的横坐标是吉西他滨的浓度,纵坐标是相对细胞活性,分组还是Ku70之后的分组验证,这是增殖表型的MTT比色法实验,在用MTT检测细胞增殖的同时摸索最佳的药物浓度
- Fig.1C:克隆形成实验,关注增殖表型,分组同样是Ku70敲减组
- Fig.1A-C作者使用的是胰腺癌的MIA-PaCa-2细胞系验证,后面的D-G作者换了其他的细胞系,方法是一样的,其中D-E是PANC-1细胞系,F-G是原代培养的胰腺癌细胞,D和F图是分子操作验证,E和G是用MTT比色法检测增殖表型
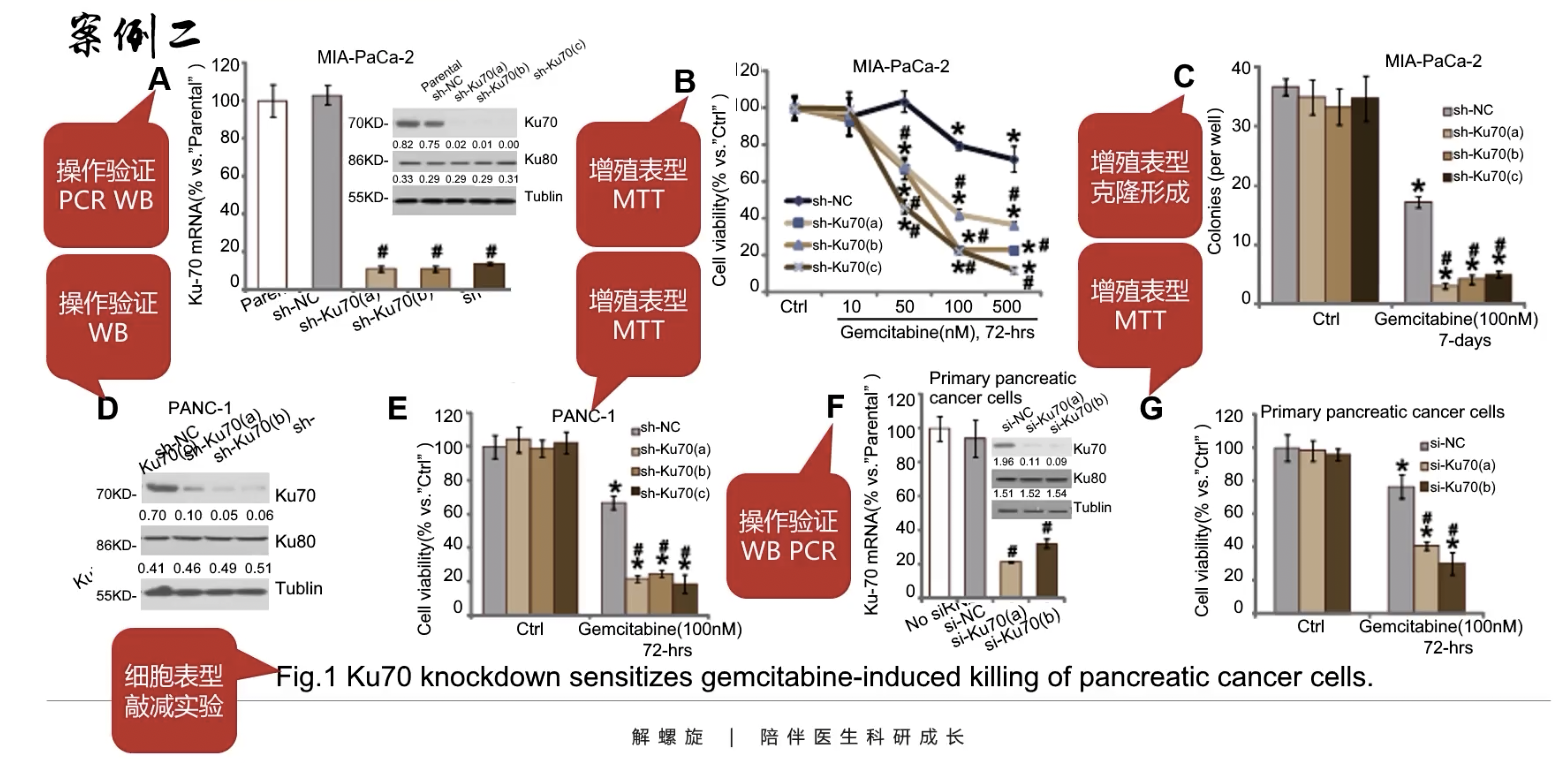
Fig.2
- 细胞水平的敲减实验,涉及的表型是DNA损伤和细胞凋亡
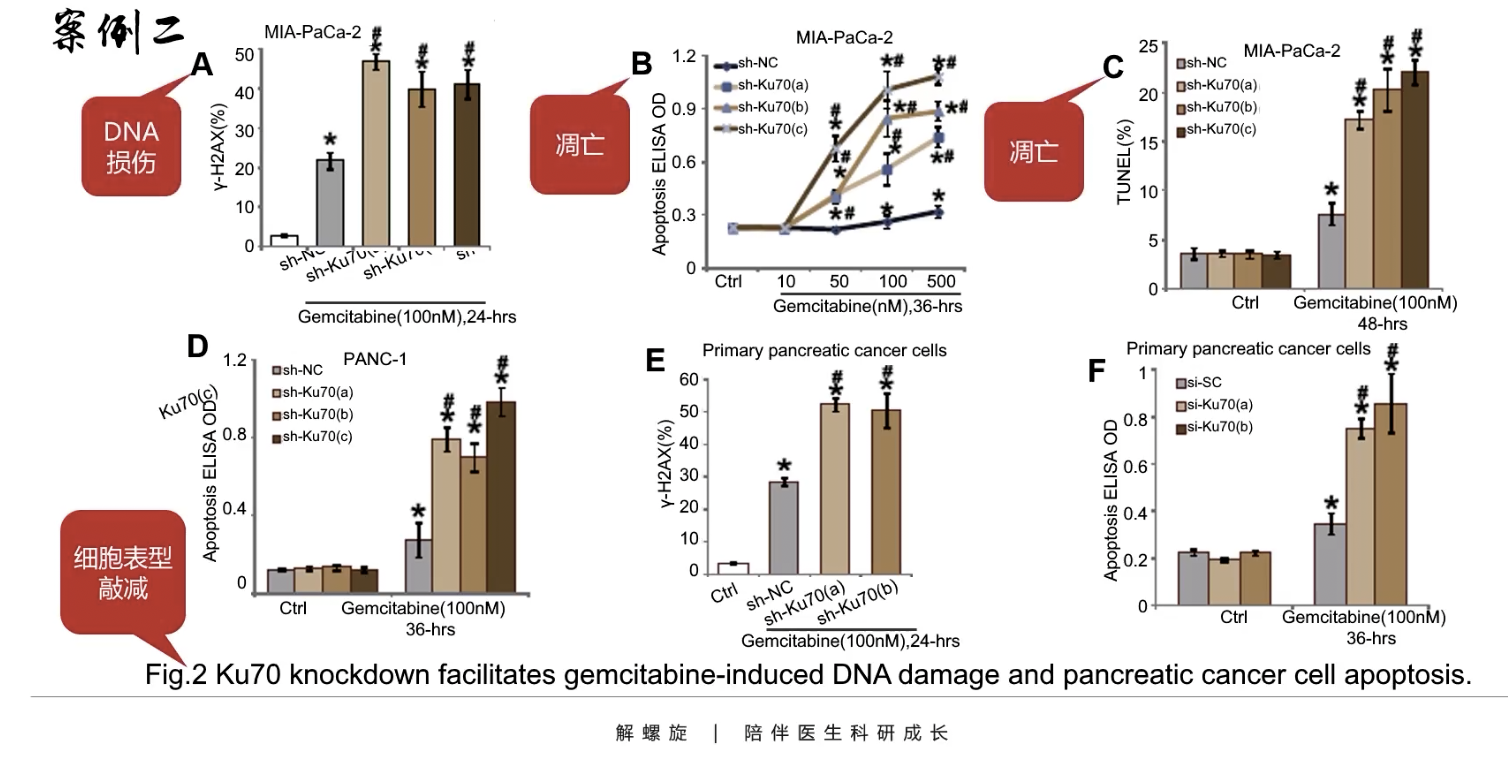
- Fig.2A-C是在MIA-PaCa-2细胞系内进行的敲减实验,分组还是阴性对照组和敲减组,其中检测DNA损伤的实验是γ-H2AX,检测凋亡的实验是TUNEL和ELISA
- Fig.2D-F是在PANC-1以及胰腺癌原代细胞细胞系内进行的敲减实验,其他同
Fig.3
- 过表达实验+细胞水平(over-expression+cancer cells)
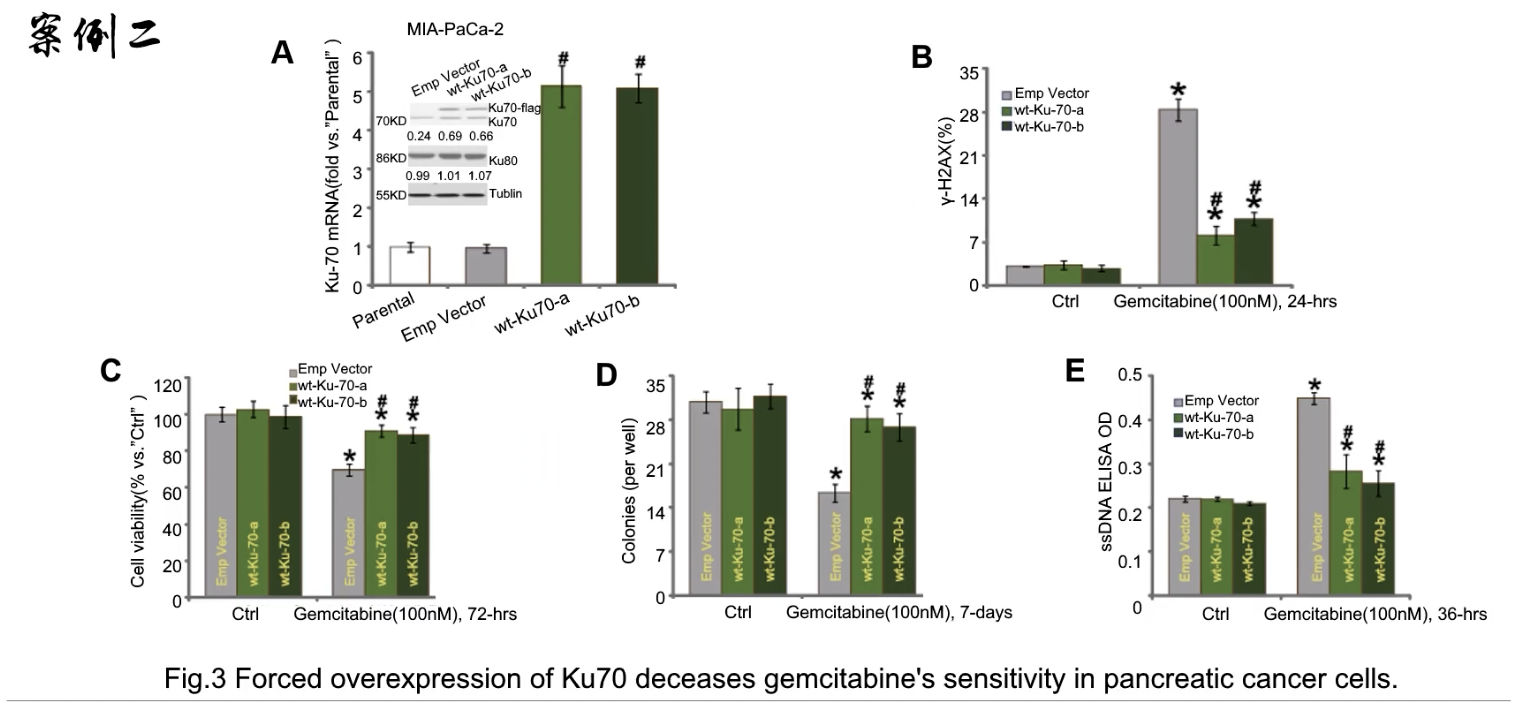
- Fig.3A:横坐标:wt-Ku70-a和wt-Ku70-b两个过表达的细胞,纵坐标Ku70的mRNA表达水平,里面的小图是用western检测带有FLAG标签的Ku70-分子操作验证
- Fig.3B:用γ-H2AX来检测DNA损伤表型,分组是对照和过表达
- Fig.3C-E:分组没变,做的实验分别是MTT比色法检测细胞活性(增殖表型),克隆形成(增殖表型)和ELISA检测(凋亡表型),这些方法和Fig.1-2是一样的,只是把敲减变成了过表达
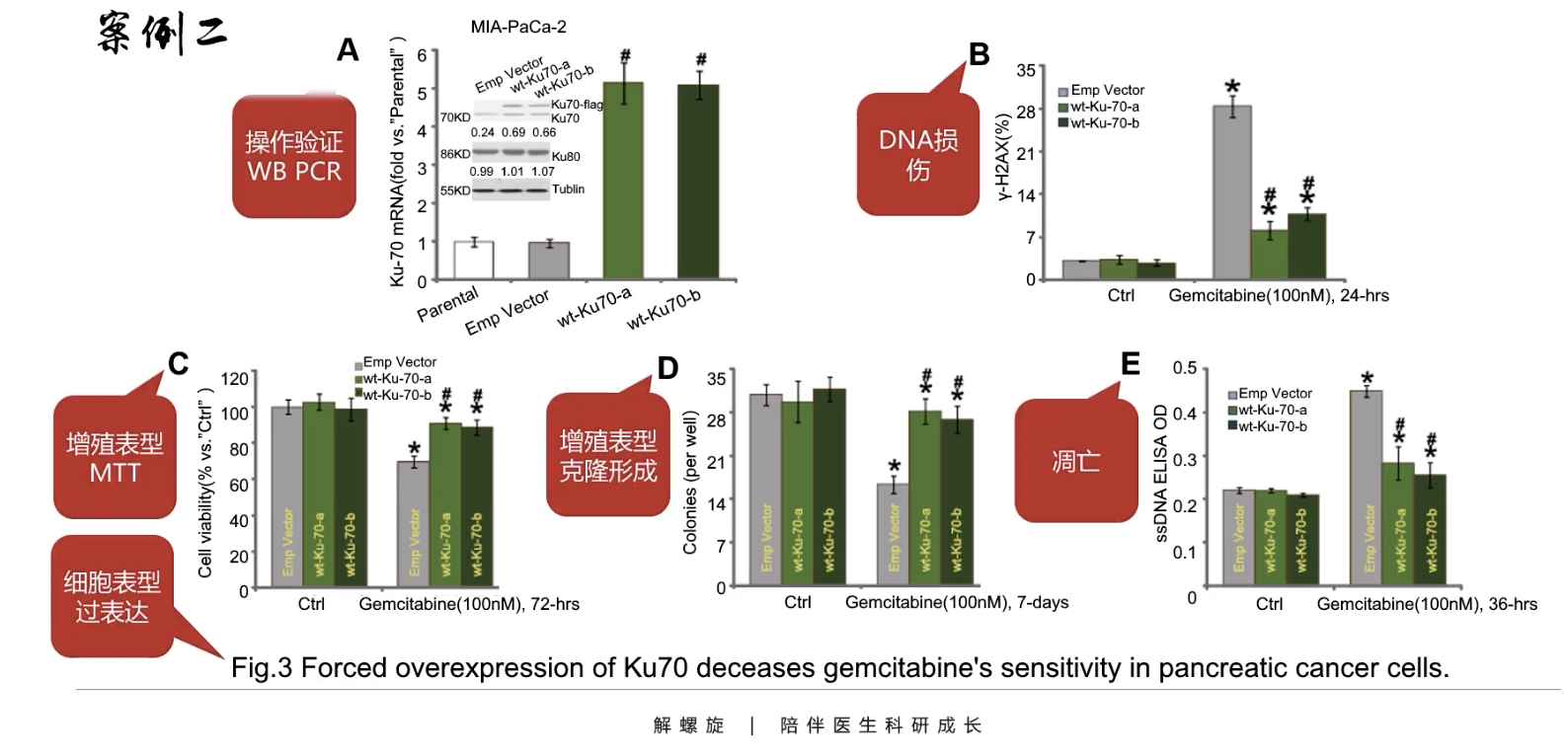
Fig.4
- 动物水平的(in vivo)敲减实验(knockdown)
- 分组:有没有药(gemcitabine)和有没有敲减(NC/a/b)
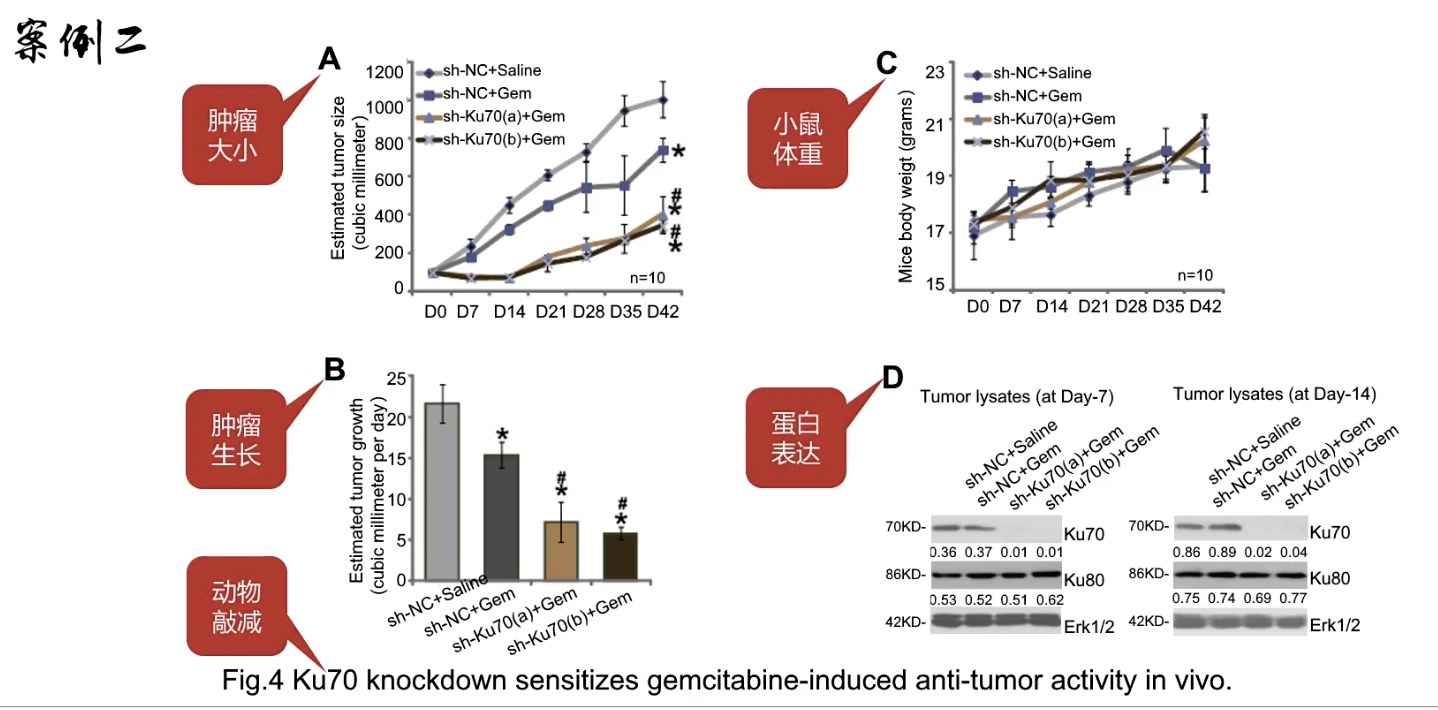
- Fig.4A和C:横坐标D0/7/14/21/28/35/42是天数
- Fig.4A:肿瘤大小,敲减+吉西他滨,肿瘤的大小显著变小了
- Fig.4B:肿瘤生长的情况,在抑制了Ku70后,加药可以显著抑制肿瘤的生长
- Fig.4C:小鼠体重
- Fig.4D:敲减后检测肿瘤的Ku70水平
总结
文章3
读题目和反推假设
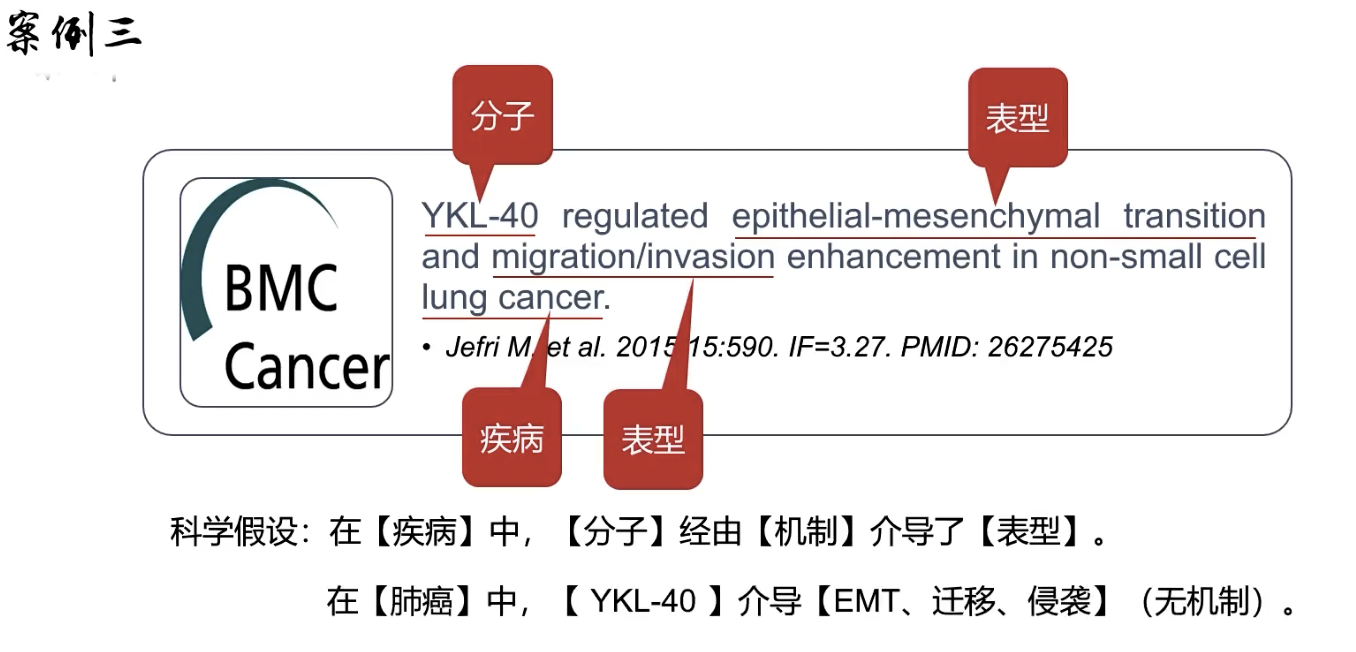
- 根据单变量研究套路,反推假设,预测作者的行文逻辑
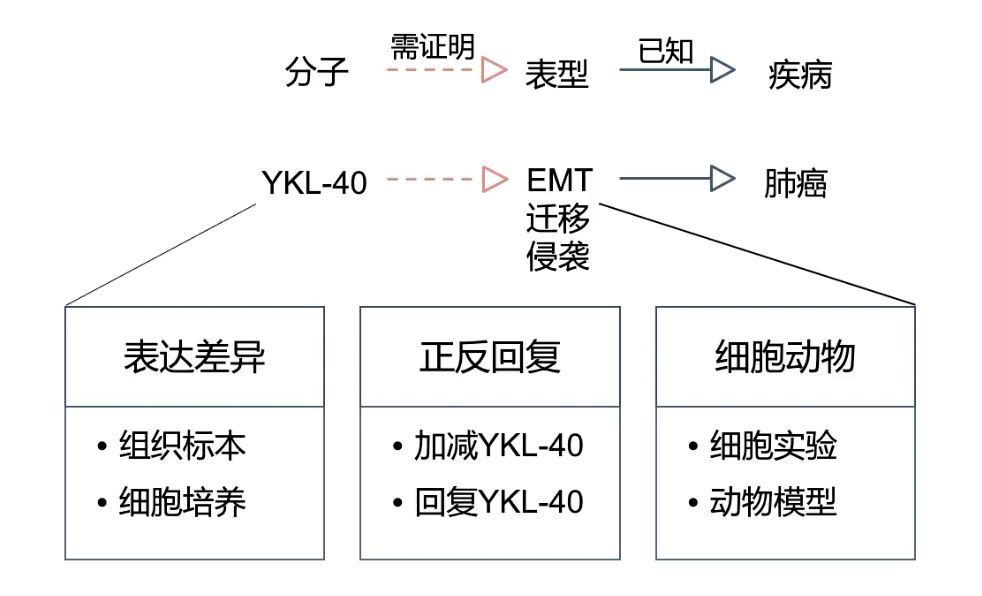
读图片
Fig.1
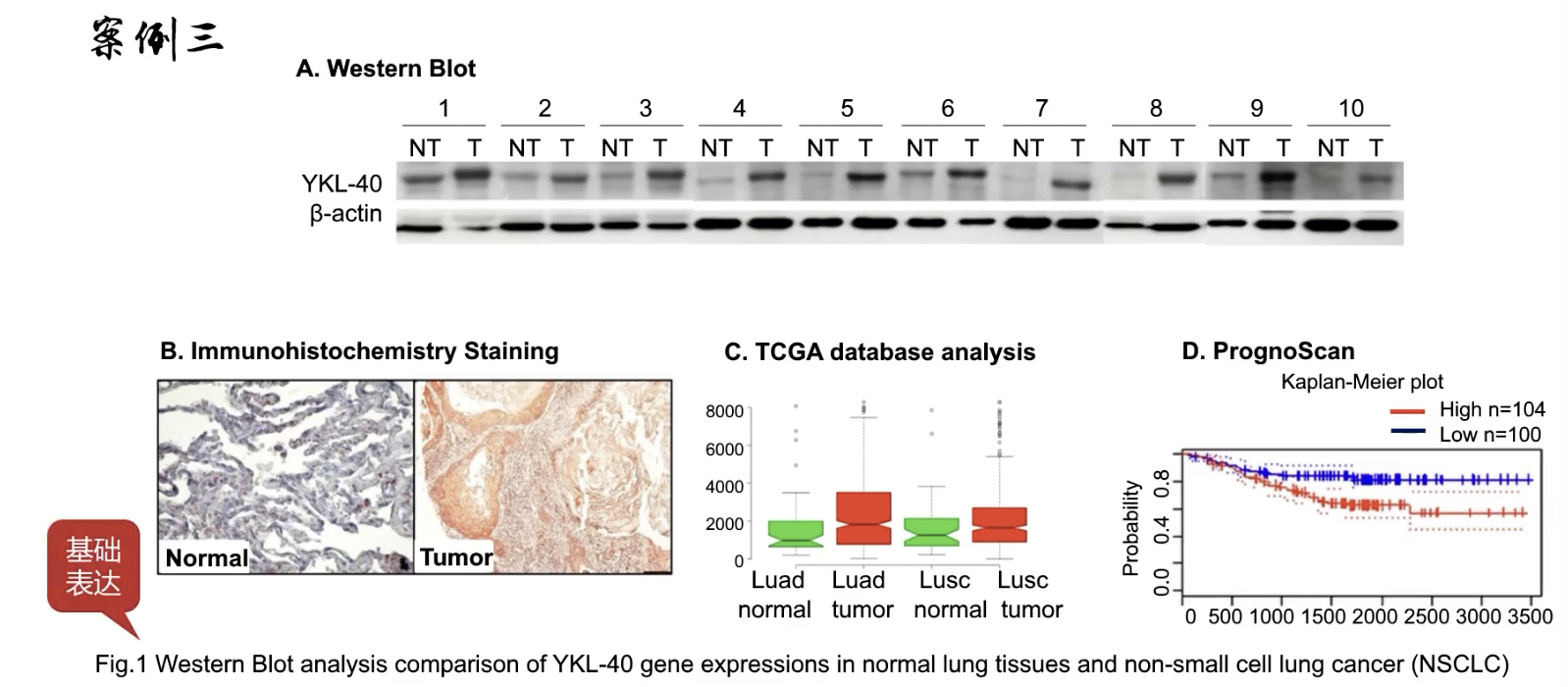
- FigureLegends关键词:western blot analysis ;tissues,说明是基础表达的结果;
- Fig.1A:NT-normal tissue;T-tumor,配对样本检测目标分子,提示目标分子在癌组织中高表达
- Fig.1B:IHC的结果,检测分子的常用方法
- Fig.1C:TCGA数据库的表达检测,通常在检测表达差异的时候,很多文章都是选了一些临床样本做分析,然后收集开源的数据库信息做佐证,可以用作间接证据
- Fig.1D:生存分析,高表达组和低表达组能分开(样本不是作者收集的,挖别人的数据发自己的文章的路子)
Fig.2
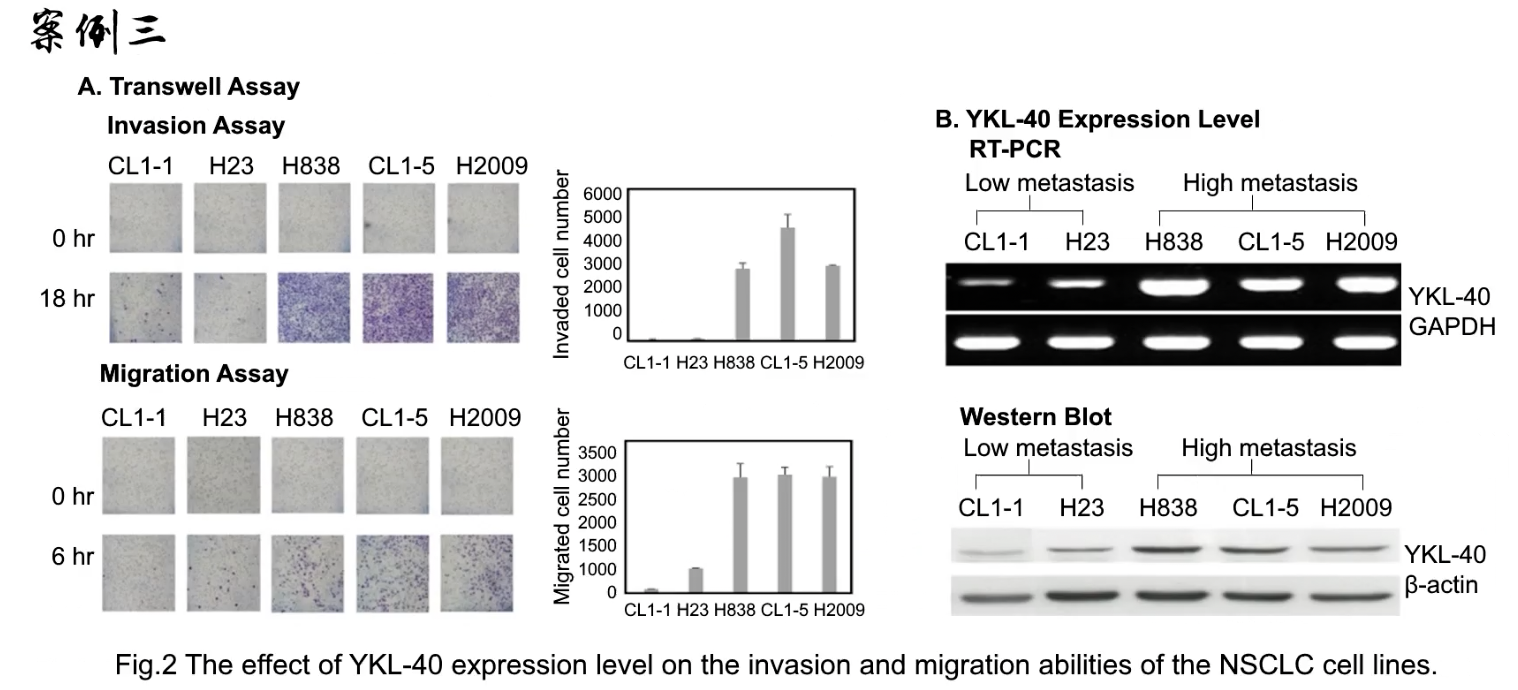
- 细胞水平+增殖/侵袭表型
- Fig.2A:transwell实验:最常见的检测迁移/侵袭能力的实验,根据有没有“铺胶”,来决定做的是invasion还是migration,从配的柱状图可以看出左侧两个细胞的迁移/侵袭能力比较弱,后三个比较强
- Fig.2B:细胞水平的表达检测,尝试证明目标基因YKL-40与迁移/侵袭表型之间的相关性,在mRNA和蛋白两个水平都得到了一致的结果
Fig.3
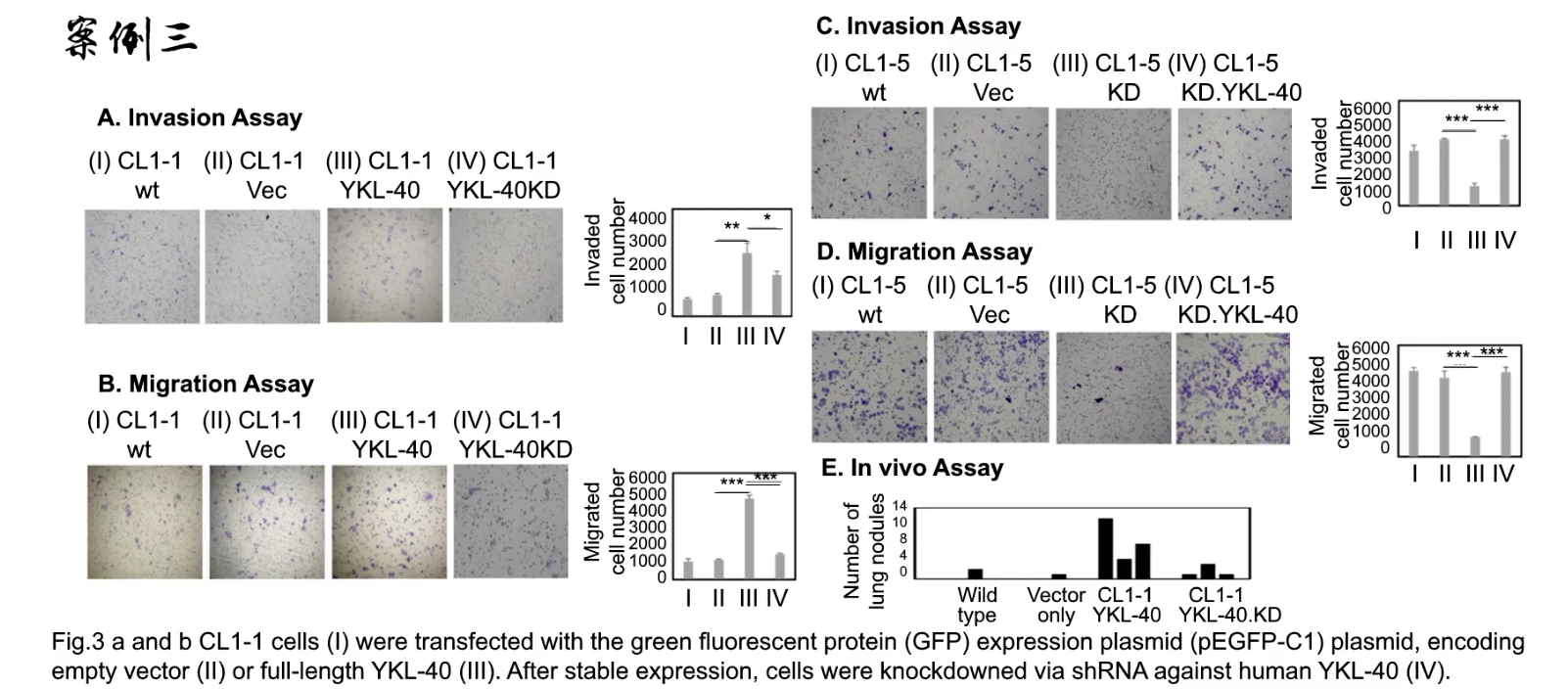
- rescue实验,Fig.3A-B是先过表达后干扰,Fig.3C-D反过来,先干扰后过表达
- Fig.3A-B过表达用的CL1-1细胞系在Fig.2中是目标分子YKL-40低表达的,Fig.3A图的侵袭实验、Fig.3B图的迁移实验分组都是一样的
- Fig.3E的in vivo字样提示了是动物实验,又是一个rescue
- Fig.3C-D过表达用的CL1-5细胞系在Fig.2中是目标分子YKL-40高表达的,Fig.3C图的侵袭实验、Fig.3D图的迁移实验分组都是一样的
Fig.4
- 细胞水平,关注EMT表型,使用的检测方法是qPCR和western
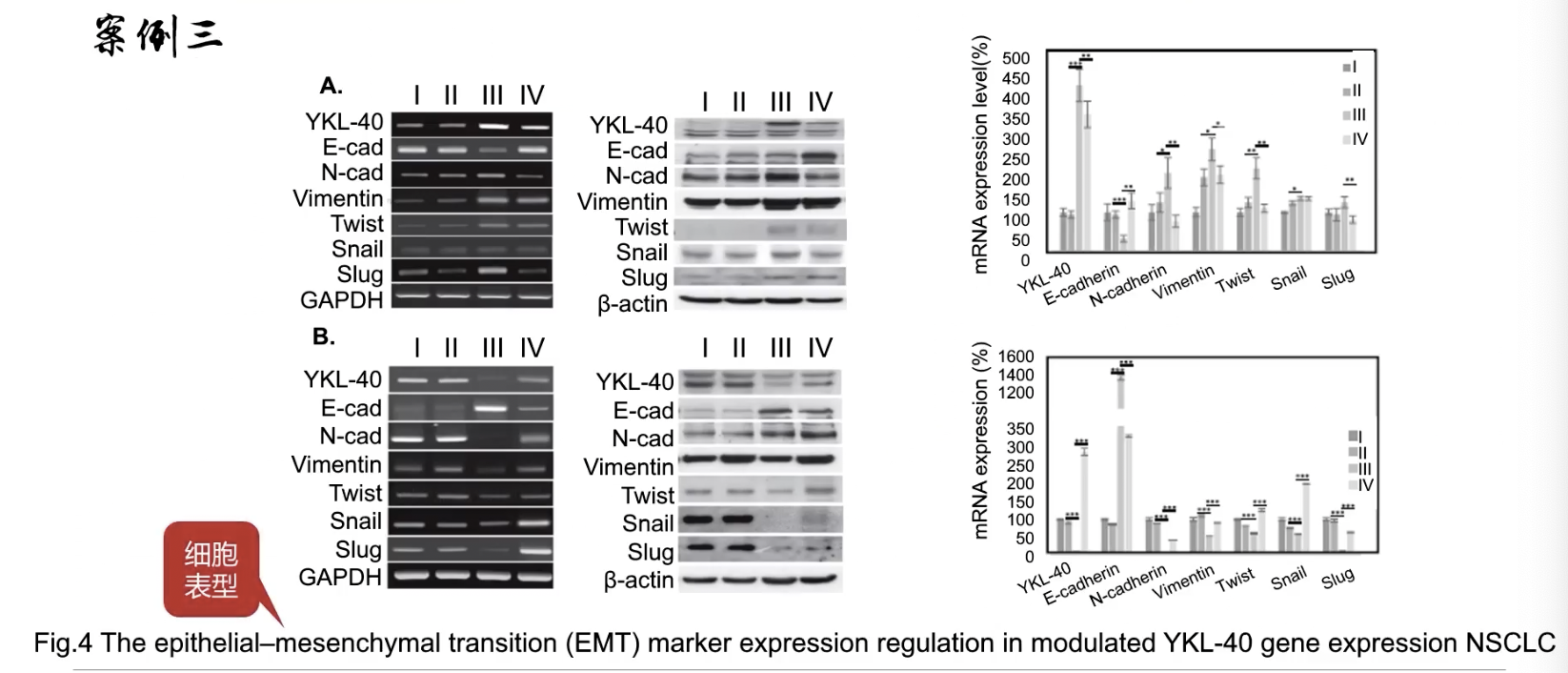
- 对两株不同的细胞进行了相关检测
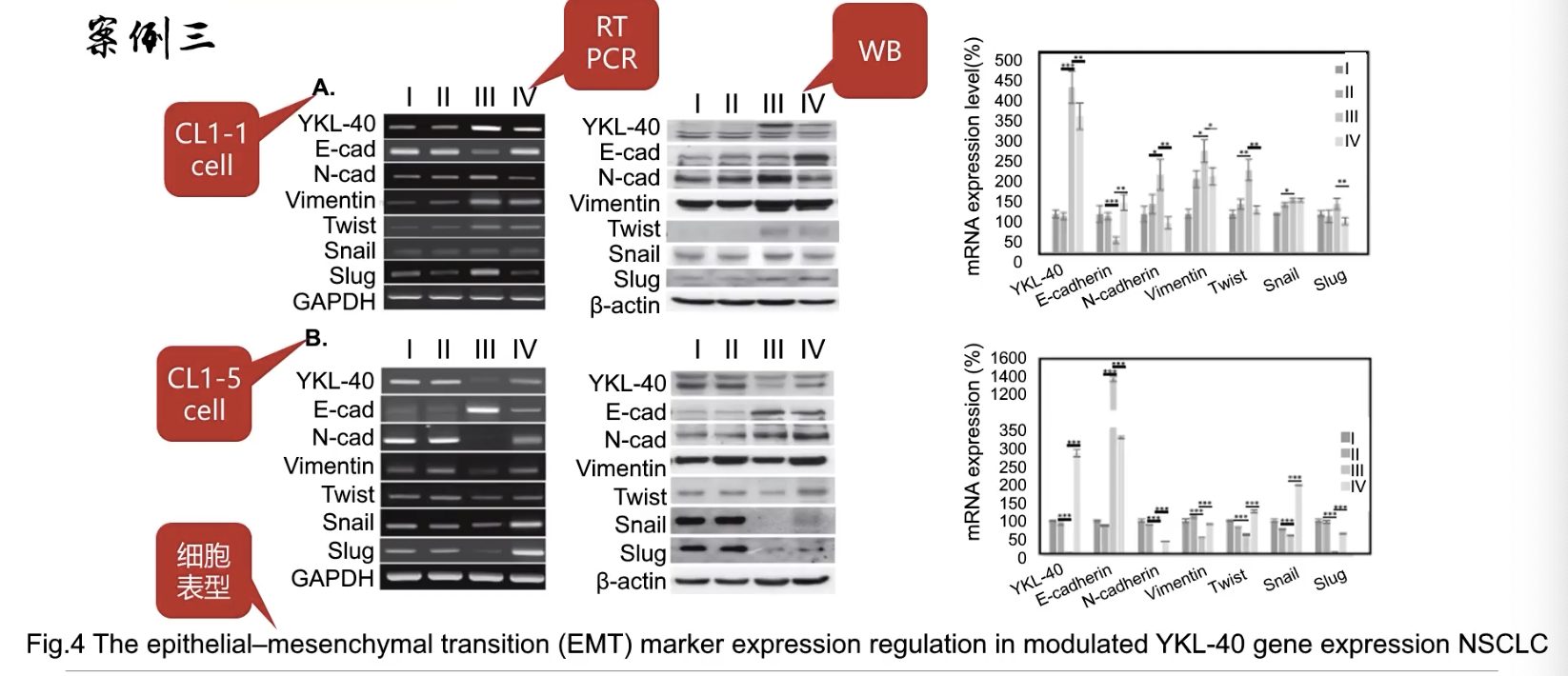
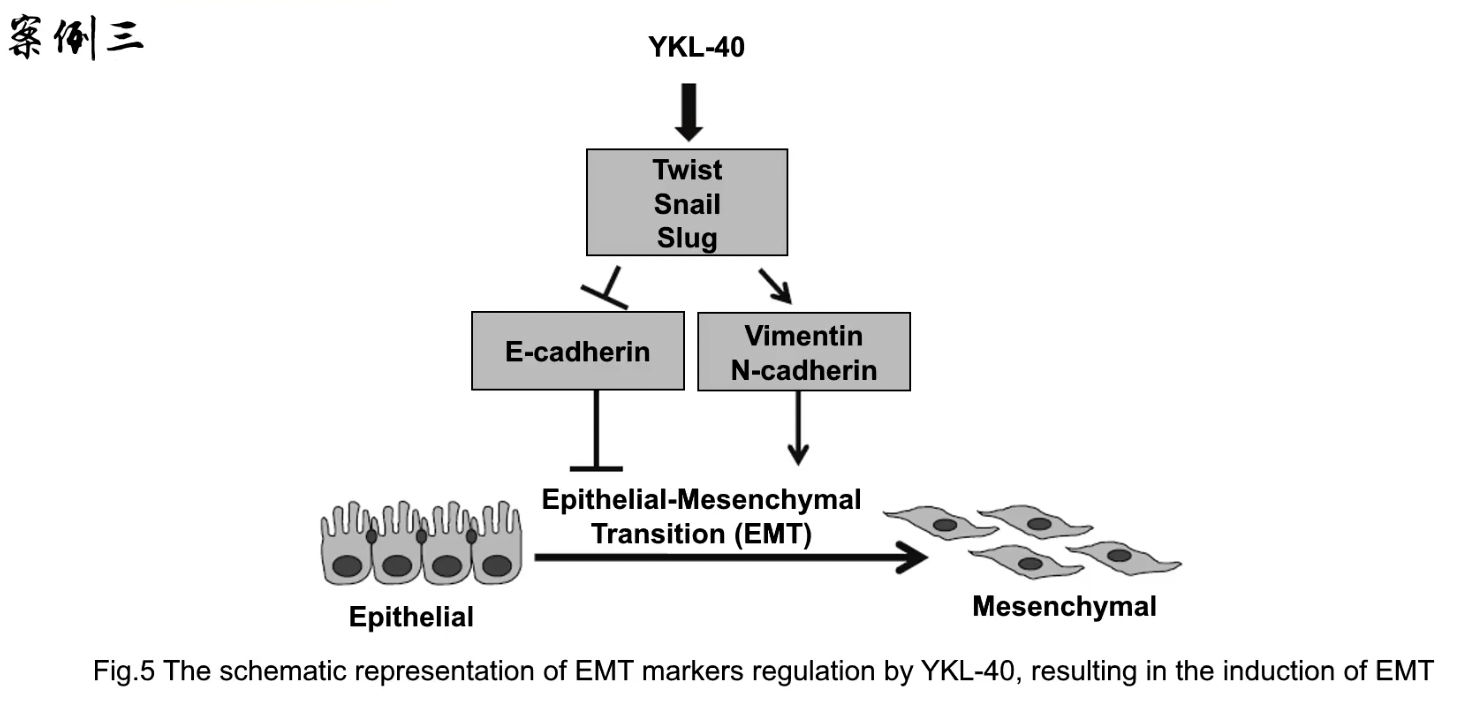
Fig.5
- 一个可能的机制图,作者只是根据自己的数据和前面已经发表的论文,“虚拟”了一张机制图出来晃人
总结
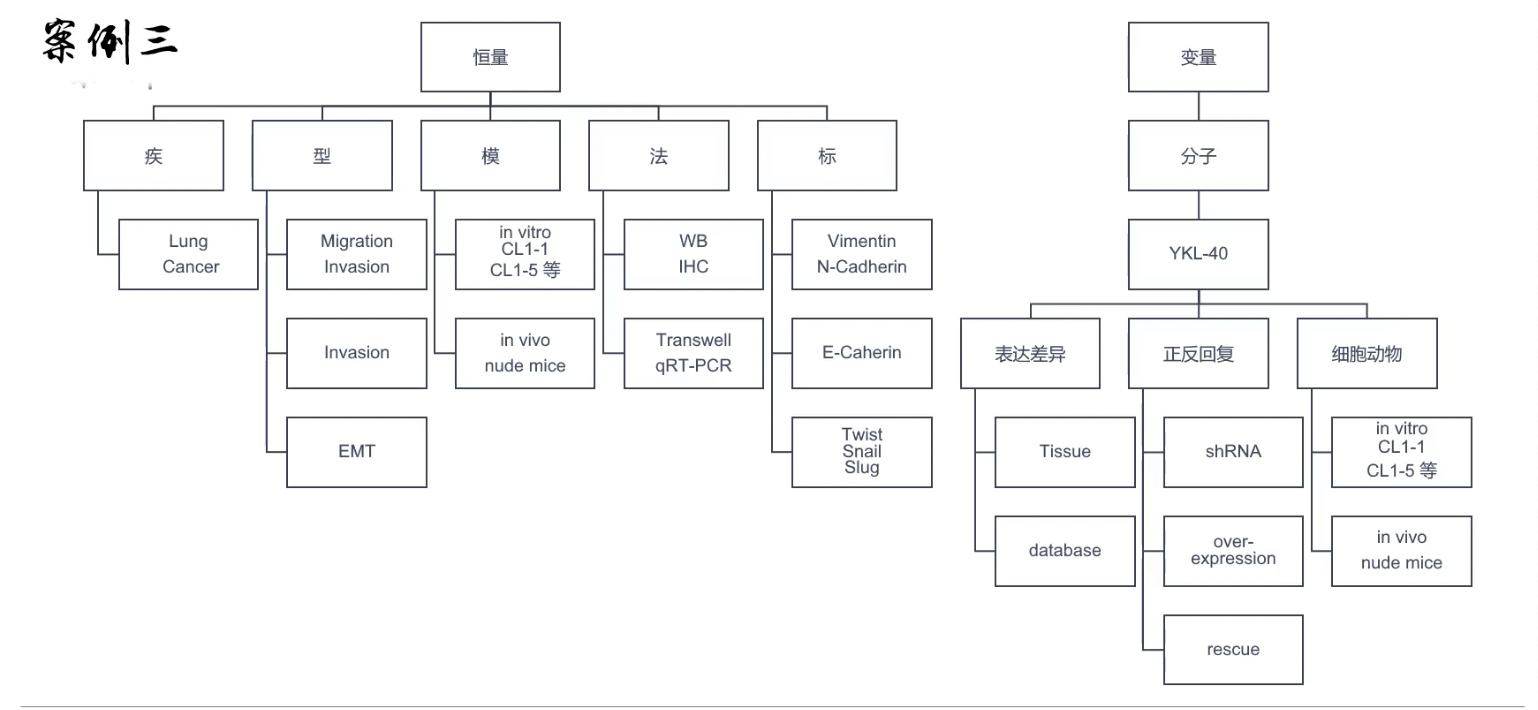
- 做了正反回复的全套实验,对于单变量研究来说相当严谨




