使用的文献:Thyroid transcription factor 1 enhances cellular statin sensitivity via perturbing cholesterol metabolism

- 本堂课要讲的第二个实验技术是ChIP。在示例文章中用到的这项技术ChIP英文全称是Chromatin Immunoprecipitation,中文名染色质免疫沉淀,是研究体内的蛋白质与DNA相互作用的有力的工具,通常用于研究转录因子与基因启动子区的结合,也能用于组蛋白特异性修饰位点的研究。研究的是体内的反应体系,跟EMSA就不太一样,因为EMSA是体外的实验。
- 体内研究是这项技术的优点,因为能够最大限度地反映体内反应的真实的情况,但这其实也算是最大的缺点,因为体内情况是很复杂的,所以如果我们用ChIP做出来的转录因子跟DNA有结合,但并不能说明转录因子蛋白与DNA是直接的结合,因为体内情况很复杂不能排除有其他的小三小四小五来帮助结合。
- 我们不可能去把所有的情况都一一排除,这也是科研的最大的问题。只能证实,无法证伪。所以做科研的一条重要思路就是体内和体外实验的结果来互相印证,通常ChIP技术也是要与其他技术联用的。
- 常见的联用技术是qPCR,这俩会被一起称为ChIP-qPCR,能够直接检测某个转录因子蛋白在既定的DNA序列上的富集。
- 如果将ChIP与二代测序技术相结合,也就是ChIP-Seq技术,将能够高效地在全基因组范围内检测与某个转录因子或组蛋白相互作用的DNA的区段。
实验原理
- 转录因子ChIP和组蛋白ChIP是有所差别的,我们讲的是转录因子ChIP。
- 细胞核存在一层膜结构,称核膜。核模把细胞质分隔成两个主要的空间,被核膜隔离在外面的是细胞质,被核膜圈在里的就是细胞核。
- 我们知道细胞核里是有基因组DNA的,转录因子蛋白能够识别并结合到基因组DNA上的一些特殊的序列,不是随便哪个序列都能结合。转录因子与DNA的结合其实不是非常的牢固,但是我们可以用一些手段把转录因子蛋白与DNA绑定到一起。
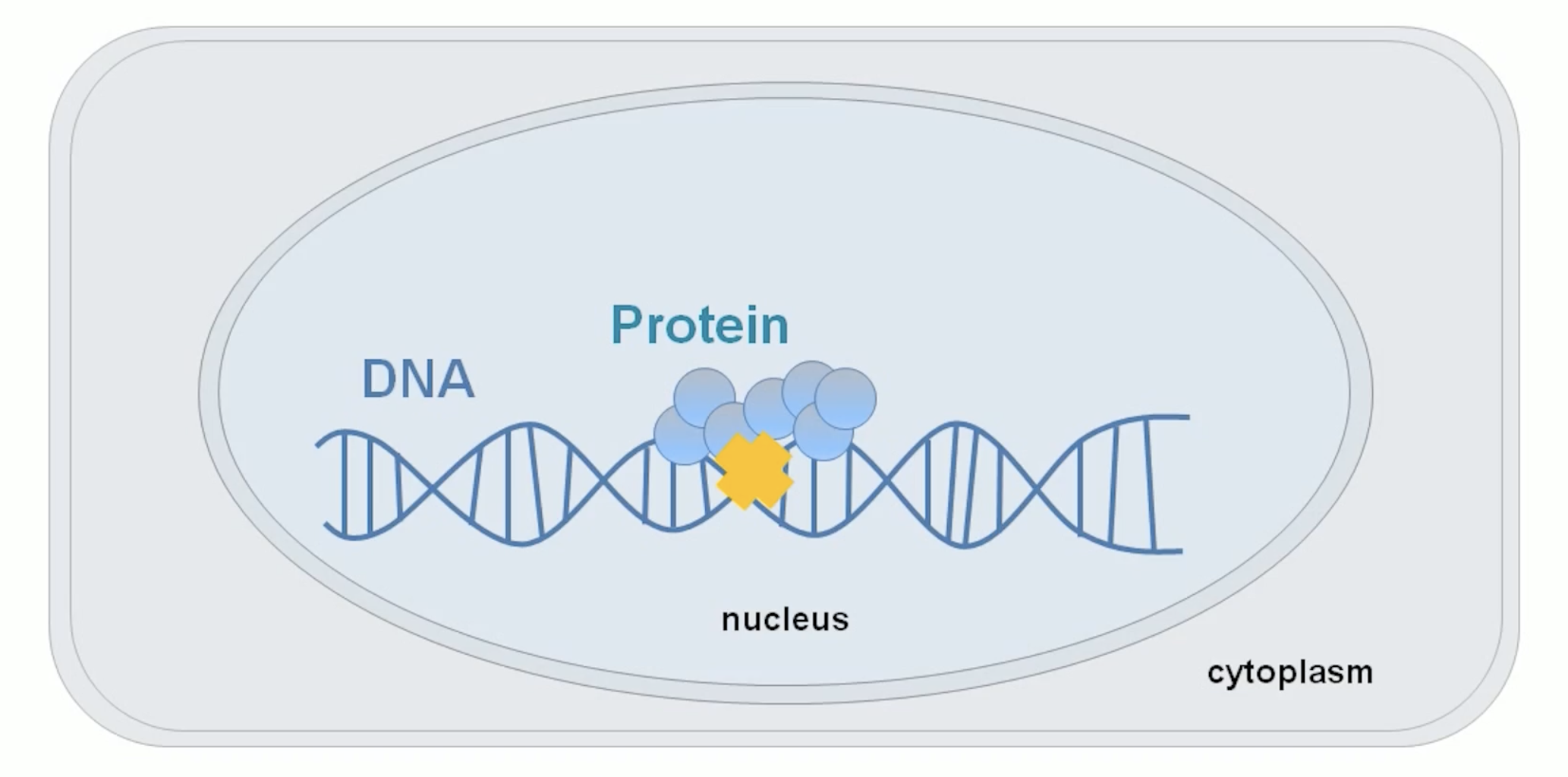
- 上图中的黄色叉就说明蛋白跟DNA就绑定好了。之后,我们就可以依次去掉细胞质,然后再把细胞核也裂解了,这样我们就能把转录因子跟DNA复合物暴露出来,然后用抗体去特异性的识别转录因子蛋白。这时就可以把抗体-转录因子-DNA复合物结合到一起。这时再加入Agrose beads,其中beads是可以与抗体结合的。我们进行离心,转录因子蛋白-DNA-抗体-Agrose beads一起离心下来。
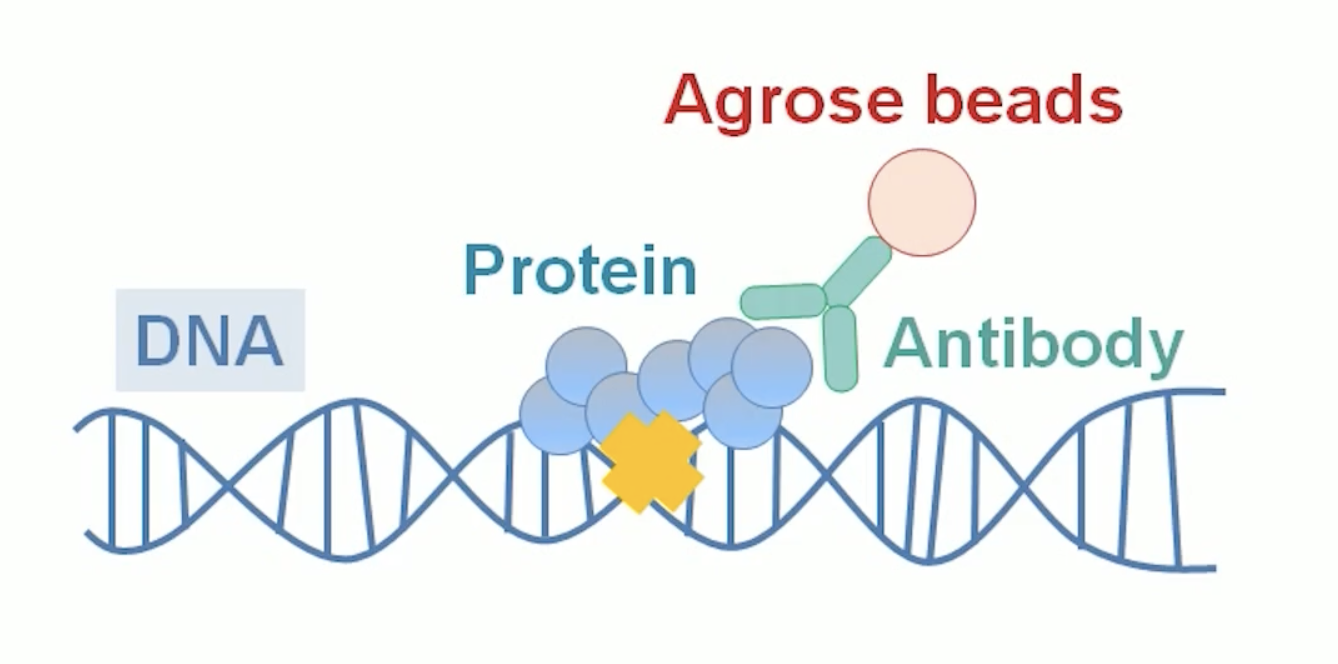
- 我们接下就可以把DNA纯化出来,用高通量测序的手段去测与转录因子蛋白结合的DNA序列是什么。也可以简单的用qPCR的方法去检测。
实验流程
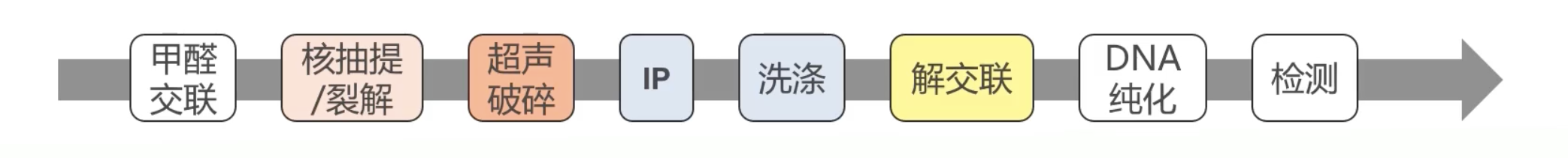
- 第一步:1%甲醛的室温交联,用1%的甲醛使转录因子蛋白与DNA绑定到一起。在这一步做组织和培养的细胞是稍微有些不同的
- 培养的细胞可以直接往培养上清中加入甲醛,使甲醛的最终浓度为1%。
- 如果直接把组织块丢进甲醛中,有两个问题,首先组织块内的甲醛浓度没办法去控制,其次是组织块有一定厚度,甲醛不能充分的渗透进去。
- 我们的做法是,我们需要先用小剪刀将组织块剪碎,然后再用玻璃匀浆器,将剪碎的组织就分离成单个的细胞。当然玻璃匀浆器是没办法非常充分的分离出单个细胞的,在玻璃匀浆器之后,我们还要再用滤网过滤一下保证得到的是单细胞,然后再加入甲醛进行交联。
- 交联完之就是核抽提和裂解,进行室温裂解就行。把细胞质去掉,只保留细胞核,然后用核裂解液将细胞核充分裂解。
- 第三步:Bioruptor超声破碎,主要是为了把基因组打断成一定大小的片段。基因组DNA是非常大的,抽过基因组DNA的同学应该知道,将细胞裂解之后用无水乙醇去沉淀基因组DNA,这时候是可以看到基因组DNA是肉眼可见的、长长的细丝状的,非常的长。这么长的基因组DNA,能够被某个特定的转录因子蛋白结合的位点肯定是非常有限的,会有非常多的DNA序列都是不能被转录因子结合。将基因组DNA打断后,我们就能够排除掉些不被结合的片段,而尽可能多的富集到被转录因子结合的片段。在这一步推荐用BioRobot公司的非接触式的超声破碎仪进行高功率的超声,具体的超声次数以组织及细胞类型而定。
- 第四步:进行免疫沉淀IP,在超声裂解物中加入Protein A/G Plusbeads和抗体以及相应的对照IgG进行免疫沉淀。这个实验中用的beads是特殊的是A/G Plus的beads,因为普通的Protein A或者是Protein G的beads表面是会有很多的孔洞,这样的活就会造成非常多的非特异性的吸附,而A/G Plus beads的表面是光滑的,所以就不会有这种额外的非特异性的吸附。
- 第五步:以不同离子强度的Buffer去清洗IP过后的beads,把非特异性的结合都清洗掉。
- 第六步:进行解交联,将蛋白-DNA复合物从beads上起脱下来进行解交联,也就是把蛋白和DNA解交联,我们在第一步中用甲醛交联了转录因子和DNA,在这里我们要把DNA和蛋白分开。
- 第七步:DNA的纯化。我们一般可以用酚/氯仿法去进行纯化
- 最后:用qPCR或者高通量测序的方法去检测IP到的DNA。

ChIP的数据分析
- 取决于检测IP到的DNA的方法是qPCR还是用高通量测序。
- 示例的文章中的数据分析是用的qPCR检测方法:
- 这样得到的数据一般会处理成5%input或者是10%input的倍数得到的富集度。input就是没有经过IP的样品里的基因组DNA,通常我们在实验的时候,会在细胞核裂解之后超声破碎前把样品平均的分成几等份,一份作为input,一份超声后用IgGIP,一份超声后用特异的抗体IP
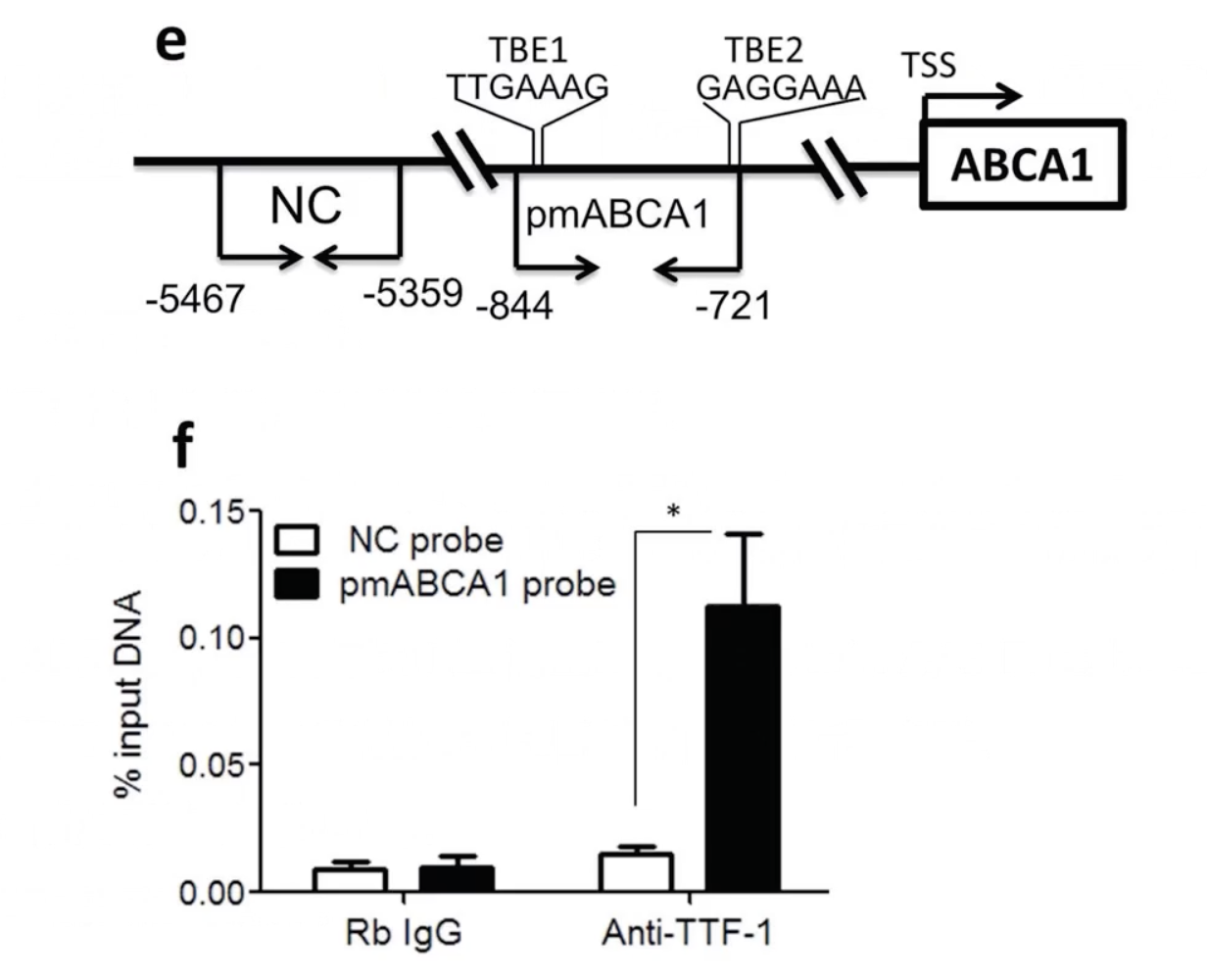
- 我们可以看一下示例文章的结果图,作者检测了ABCA1基因启动子区上的转录因子TTF-1的富集度。TSS就是转录起始位点,Transcription Start Site。一般我们会把TSS标为0,这也是这个图里的缺陷,应该在TSS的位置标个0的。
- 我们一般认为TSS上游是基因的启动子区,’pmABCA1’是基因的启动子区,’NC’就太远了,有可能是内含子之类的区域。
- 在基因的启动子区标记碱基的位置是用负数表示。作者发现在TSS上游的-721到-844有两个TTF-1的结合位点,并命名为TBE1和TBE2,也就是TTF-1 binding element 1和TTF-1 binding element 2。观察f的结果,可以看到抗TTF-1的抗体能拉下来核酸片段,这些核酸片段能够被针对ABCA1启动子-721到-844的引物P出来,而对于对照区域,作者是把’NC’这块非常非常远的这块区域作为对照。在实验设计上说,这块区域是TTF-1不能与之结合的。实验结果也证明,对照区域并没有检测到转录因子,所以TTF-1跟对照是不能结合。IgG这一组就很好理解了,因为IgG是不能特异的识别我们的目的转录因子蛋白的所以,无论是目标DNA序列还是对照的DNA序列都不能产生富集结果。
- 作者把数据处理成了%input DNA。虽然这种方法不是特别常用,但是也是可以的,只要能说明问题就行。还有一种不算常用的处理方法:把IgG这一组作为1,然后再看实验组的富集度是IgG组的多少倍。
- ChIP-Seq技术会涉及到高通量数据分析,他最后呈现出来图可以有两种,一种是比较直高的峰图,富集度越高的位置峰就越高;另外一种是Motif图,因为转录因子与不同的基因结合在结合位置的序列,也就是Binding element并不是一模一样的;通过对全基因组范围内Binding element扫描后进行分析总结,这些Binding element的序列特点,就能够得到Motif图,知道哪些位点的碱基是保守的,在这些Binding element里这些碱基就会高频的出现,而哪些位点的碱基是相对来说不保守的。
常见问题

- 问题2补充:ChIP和western不一样,western推荐使用单抗,但ChIP可以尝试使用多抗









