使用的文献:Thyroid transcription factor 1 enhances cellular statin sensitivity via perturbing cholesterol metabolism

- 在本节内容中,我们会继续介绍两项应用:第一项应用是如何通过Jasper网站获得转录因子结合位点的相关信息,第二项应用是如何通过UCSC联合Jasper预测靶基因启动子区域内的候选转录因子。
- 首先我们介绍如何查询一个转录因子在DNA序列上的转录因子结合位点。一种手段当然是查阅文献,有些文献中会把转录因子的结合位点在文章中直接点名出来,比如在我们的模板文章中作者就写出了TTF-1的结合位点,另一种方式就是通过生物信息学相关的网站进行查询,我们主要通过Jasper网站向大家进行演示。
演示:获得转录因子结合位点的相关信息
- Jasper是一个收录了众多转录因子偏好结合序列以及结合模式的数据库,它是一个开源的公共数据库,数据使用没有任何的限制。
- 为了通过Jasper数据库获得特定转录因子的结合位点信息,首先登录Jasper数据库。在整个页面的左侧是导航栏,Jasper数据库包含了九个子数据集,其中数据量最为庞大的子数据集是Jasper Core。如果需要更换其他子数据集可以点击左侧导航栏的Browse Collections,然后在下拉菜单中找到需要使用的子数据集即可。
- 在这里特别需要说明的是,当我们使用了Jasper数据库,并且发表我们自己研究成果时,请大家一定要在文章中引用Jasper数据库的相关文献,文献信息就在主页上的”Citing”区域。随后我们可以直接在主页的检索栏中输入我们要查询的转录因子名称,也可以点击左侧导航栏的Search按钮,我们在检索栏中输入NKX2-1。需要说明的是,在检索栏下方有很多的筛选条件选框,可以选择不同的子数据集,通常我们都选All,其他还可以选择物种Species,转录因子所属的转录因子的分类Class,转录因子分类相关内容请复习之前的内容。还可以选择转录因子所属的转录因子家族Family和不同的数据库版本等等。点击Search进行检索。
- 在新的页面中,我们可以看到转录因子的ID是PH开头的编号。Jasper数据库总共含有9个子数据集,每个子数据集它的ID都有不同的编码方式。PH表示的子数据集是Jasper PBM Homeo,这个子数据集当中的PBM是Protein Binding Microarray的缩写,也就是蛋白结合芯片,因为在这个子数据集当中通过蛋白结合芯片PBM的数据,收集了多个属的转录因子同源结构域数据。很遗憾的是,Jasper数据库当中还没有收录人的NKX2-1的信息。另外,在这张表中还列出了NKX2-1属于Helix-Turn-Helix这一个分类的转录因子,隶属于Homeo蛋白家族。
- 直接点击ID号进入详细的网页信息,网页的左侧是转录因子的简要信息,其中的Validation这一项专门列出了相关数据的文献出处,如果有需要可以点击链接下载相关文献。右侧的上方是转录因子结合位点的序列标识图,下方是Position Frequency Matrix位置频率矩阵PFM信息。无论是PFM还是序列标识图,都是可以下载的。
- 在模板文章中提到,TTF-1的结合位点序列是TTGAAAG及GAGGAAA,通过简单的比对我们就可以知道文章中提到的TTGAAAG及对应TTF-1结合位点中的第9位到第15位。

- 借着这张序列标识图,我们也说明一下序列标识图怎么看。
- 首先:第10位所有字母的总高度都比不上第9位的T的高度,说明第10位的碱基一致性和保守性相对第9位更差。其次,在第10位内部有三个字母T、C和A,其中高度最高也是排在最上面的是T,说明在第10位的内部出现概率最高的是T,然后是C,出现概率最低的是A,当然A也就排在第10位的最下方。但是模板文章给出的另外一段结合位点GAGGAAA并没有在预测结果中找到对应的序列,这应该就是实验验证序列和算法预测序列之间的差异。所以我们千万不要过于迷信生物信息学的预测结果。
演示:靶基因启动子区域的转录因子预测
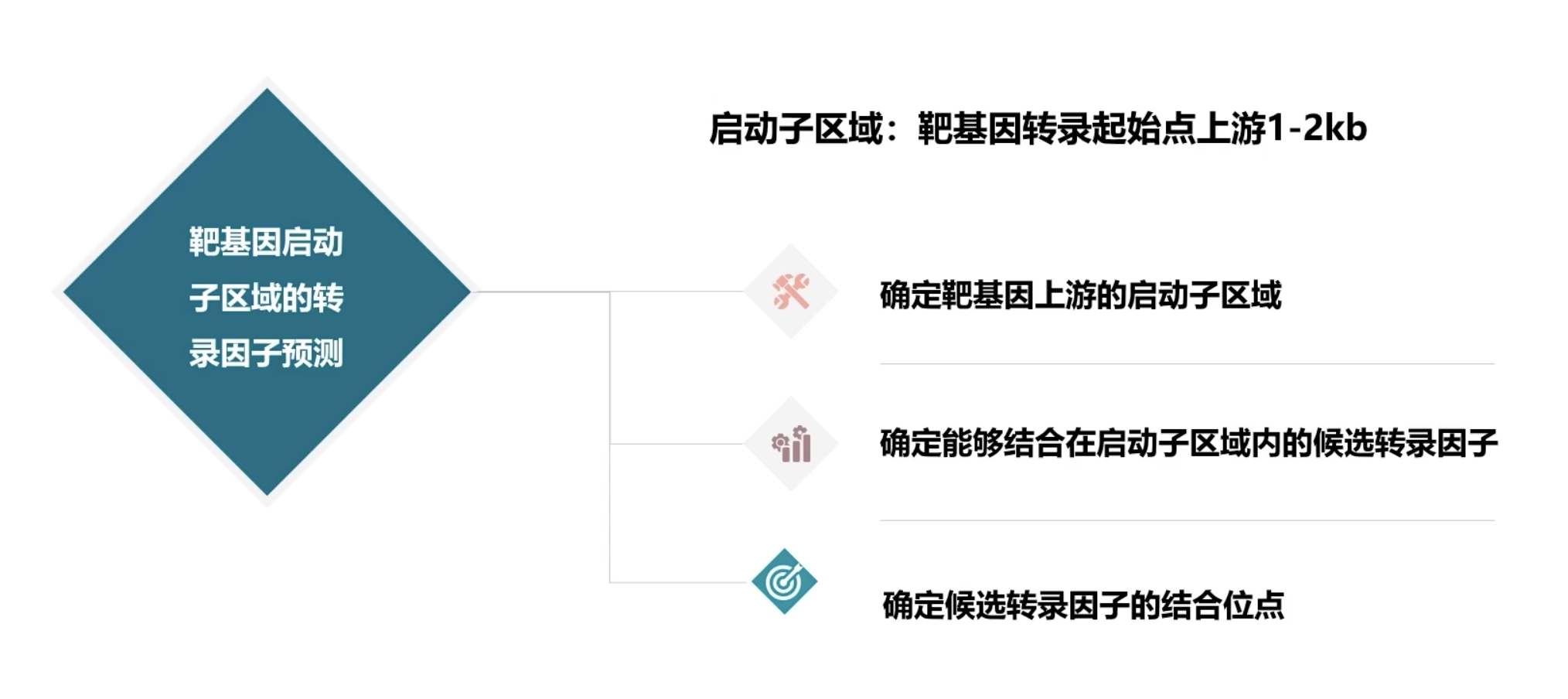
- 最后我们演示的内容是:对于特定的基因如何预测该基因启动子区域内可能结合的转录因子。
- 有些学员会问:为什么我们介绍的是通过靶基因倒推转录因子,而不是通过转录因子推测它的下游基因呢?第一,由于转录因子在DNA序列上的结合位点一般都很短,不超过20个bp,有时甚至更短;第二,由于转录因子结合位点有一定的序列冗余性,因此如果通过转录因子的结合位点,去基因组中通过BLAST找潜在的结合位点,那获得的候选基因实在太多了,后期验证会非常的困难。因此,更好的方法是通过靶基因,反推出可以结合在靶基因启动子区域内的转录因子,这样更具有可操作性。
- 到目前为止,对于基因的启动子区域还没有非常严格的区域界定。研究人员通常认为转录起始位点上游的1kb-2kb的区域范围内都是启动子区域。例如在本篇模板文章当中,研究者就是选用转录起始位点上游1kb的区域作为ABCA1基因的启动子区域。如果想把启动子区域放得宽一些,就可以选用转录起始位点上游2kb的区域。
- 我们以模板文章为例,选用ABCA1基因转录起始点上游1kb作为启动子区域。在整个检索过程中,可以把预测靶基因启动子区域的转录因子的全过程分为三个步骤:第一步是首先确定靶基因上游的启动子区域;第二步是确定能够结合在启动子区域内的候选转录因子;第三步是确定候选转录因子可以结合的TFBS,也就是转录因子结合位点。
- 既然要通过靶基因的启动子区域反推出转录因子,首先需要做的就是找到靶基因的启动子区域。我们以模板文章ABCA1为例,首先登录UCSC网站,UCSC网站是2001年开发的,至今每年都在更新,这个网站的维护机构是University of California Santa Cruz,缩写就是UCSC。这个网站的特点是:它是一个在线查看并提供基因组序列的交互式网站,涵盖大多数物种,包括脊椎动物、无脊椎动物以及主要的模式动物。
- UCSC网站在显示基因组信息时,网站同时会展示不同数据集的注释和数据源。数据源就是Tracks。而且UCSC的一个更实用的功能是,用户还可以根据自身的需求,自行在网站上添加公共的Tracks或者本地的Tracks。这个功能使得UCSC网站具有很好的扩展性。
- 我们首先点击Genomes,选择人的HG38基因组信息。如果我们要分析不是人的基因,而是小鼠的基因,就应该选择相应的小鼠基因组信息。先介绍一下UCSC页面信息,最上方是检索栏,下方是图形化的基因组信息。这些图形化的信息是和下方的功能选框以及数据源密切相关的。只要下方的功能选框和数据源改变,这些图形化的基因组信息就会跟着一起改变。网页的下方是功能选框,网站展示了九大功能分别是Mapping and Sequencing,Genes and Gene Predictions,Phenotype and Literature等等。在每一个功能选框的下方,都是不同的数据源,也就是不同的Tracks;比如在Mapping and Sequencing功能选框下方默认只展示Base Position一个数据源;在Base Position下方有下拉菜单,菜单内是数据展示的方式,Hide就是隐藏不展示数据源,Dense是数据展示较为紧密,Full就是展示全部的数据;有些选框内还有Pack, Squished这些都是数据展示的形式,可以根据个人的喜好进行选择。
- 现在在Mapping and Sequencing功能选框之下只默认展示Base Position,如果我需要增加数据源的显示,例如我要添加Assembly,直接在下拉菜单中选择展示的方式,比如我选Pack,确定之后点击Refresh刷新即可,图形化的基因组信息会自动更新。那怎么看我添加的数据源Tracks是否显示在上方的图形化界面当中呢,鼠标直接悬停在最左侧的灰色的柱子上,只要鼠标直接悬停在灰色的柱子上,网站就会自动显示信息,比如图形的第一行显示Base Position,第二行显示的就是刚才我添加的数据源Tracks Assembly,第三行显示的是GenCode V24,这就对应Genes and Gene Predictions功能选框下面的相应数据源Tracks了。对于查找靶基因的启动子区域我们不需要那么多的信息,所以我们就把无关的数据源Tracks全部都隐藏掉,只保留需要的数据源就可以。比如保留Base Position,隐藏Assembly,保留Gene Code,保留NCBI RefSeq等等,最后点击Refresh即可刷新当前界面,刷新后图形上展示的信息更少,界面更加直观。
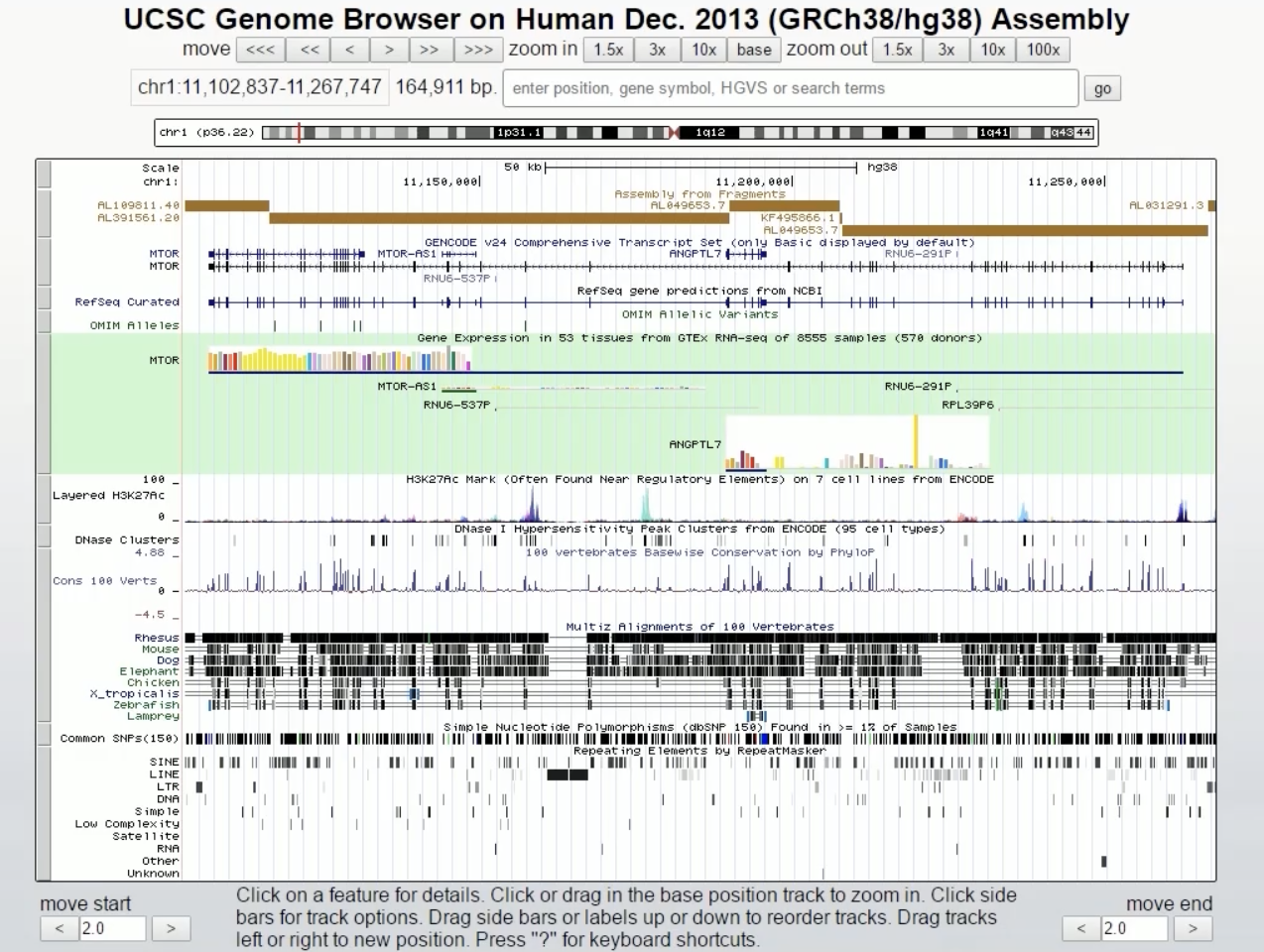

- 然后我们开始查找ABCA1基因的启动子区域,直接在检索栏内输入ABCA1,点击Go。我们稍微放大一些图形,点击Zoom Out 1.5,图形上显示ABCA1有多个ISOFORM异构体,而且异构体上有向左的箭头,这就表示ABCA1基因是在DNA的负链上编码的,而在ABCA1基因的旁边另外一个基因NipsNap3B,它的箭头向右,表示NipsNap基因是在DNA正链上编码的。
- 虽然ABCA1有多个ISOFORM,但是这些ISOFORM的转录起始点是一致的,因此转录起始点上游1kb的区域就是启动子区域,点击ABCA1进入新的页面。
- 在新的页面内下拉找到RefSeq Accession,点击进入,找到mRNA Genomic Alignment,ABCA1的简要信息,其中提到ABCA1是被DNA负链编码的,Strand为负。然后点击Genomic Sequence,在Sequence Retrieval Region Options当中其他选项都不要,只选Promoter Upstream by 1000Bases,这个选项表示当前选择的序列是转录起始点上游1000 bp的序列,也就是启动子区域。在Sequence Formatting Options当中选择All lower case,全部是小写字母显示,点击Submit。
- 刷新的页面当中显示的就是ABCA1转录起始点上游1kb的启动子区域。在模板文章当中研究者提到TTF-1有两段转录因子结合位点,位于ABCA1的启动子区域内,一段是TTGAAAG另一段是GAGGAAA,我们通过查找功能来验证一下这两段转录因子结合位点是不是在ABCA1启动子区域内。首先,我们输入TTGAAAG,这段序列在启动子区域内;然后我们再输入GAGGAAA,这段序列也在启动子区域内。至此我们已经通过UCSC网站获得了ABCA1基因的启动子区域。
- 现在开始演示第二步预测ABCA1基因启动子区域内候选的转录因子。
- 之前我们提到,UCSC有一个非常好的功能是用户可以根据自身的需求自行在网站上添加公共的数据源tracks这,是UCSC网站具有良好扩展性的体现。所以,为了分析ABCA1启动子区域的转录因子,我们首先需要通过UCSC网站调用Jasper的数据源tracks,然后才能进行分析。
- 要调用Jasper的数据源有两种方式:一种是直接从数据界面进行调用,点击tracks hubs,在检索栏输入Jasper,检索结果Jasper 2018 TFBS,点击connect连接成功;第二种方法是直接在主界面点击my data-track hubs,后面同样检索Jasper再连接。在加入了Jasper数据源之后,我们就需要:第一确定基因组的数据库,首先我们选择人的最新的基因组数据库HG38其次选择ABCA1启动子的序列位置,从刚才我们获得的ABCA1启动子的序列中,将序列的位置信息拷贝到检索栏,点击go按钮。图形数据中信息太多太复杂,我们只保留Jasper数据源Base position、gene code和NCBI refseq数据源即可,其他数据源全部隐藏掉。
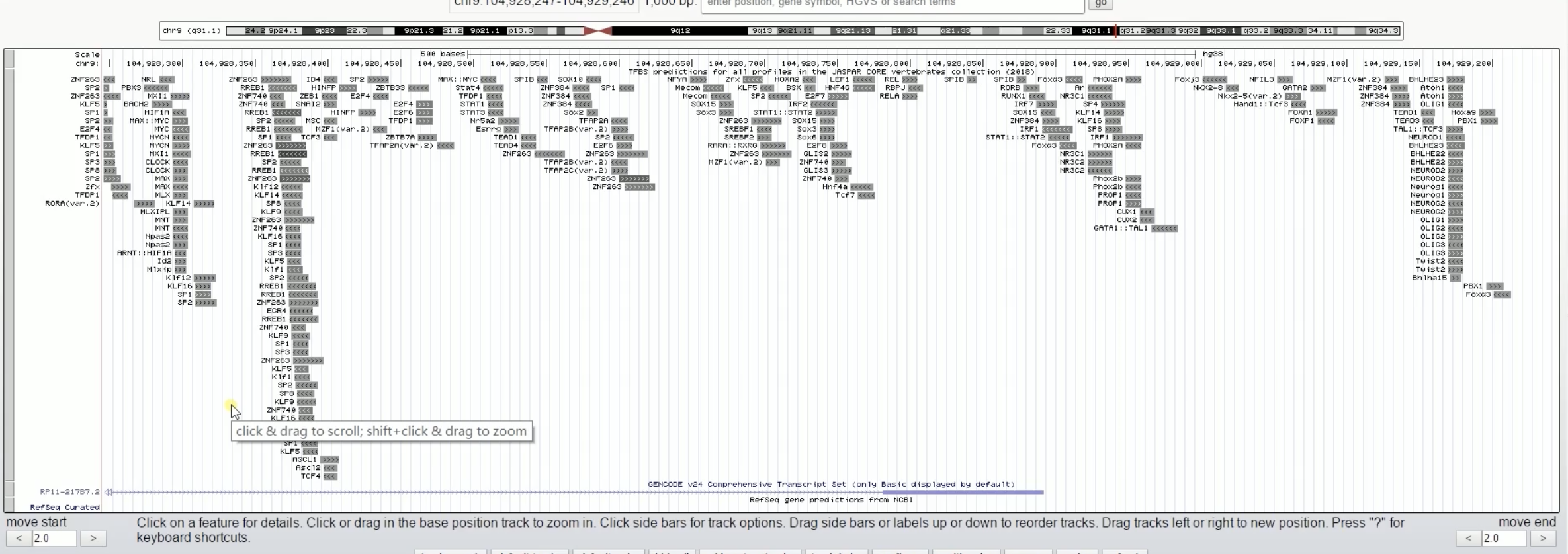
- 在图形数据的第二栏内显示的是通过Jasper转录因子数据源进行分析后获得的转录因子,这些转录因子都可以结合在ABCA1的启动子区域。有时,在分析时显示的转录因子太多或者太少,这时候需要我们对于筛选的条件进行调整。在Jasper的功能选框内点击Jasper 2018 TFBS,第一行Display Mode显示格式是Pack模式,第二行是阈值,现在默认的阈值是400,可以在0到1000的阈值之内进行调整。数字越大,阈值越高,要求越严格;反之,数字越小,阈值越低,要求越松。阈值的具体含义可以下拉网页有专门的Display Convention and Configuration区域加以描述。
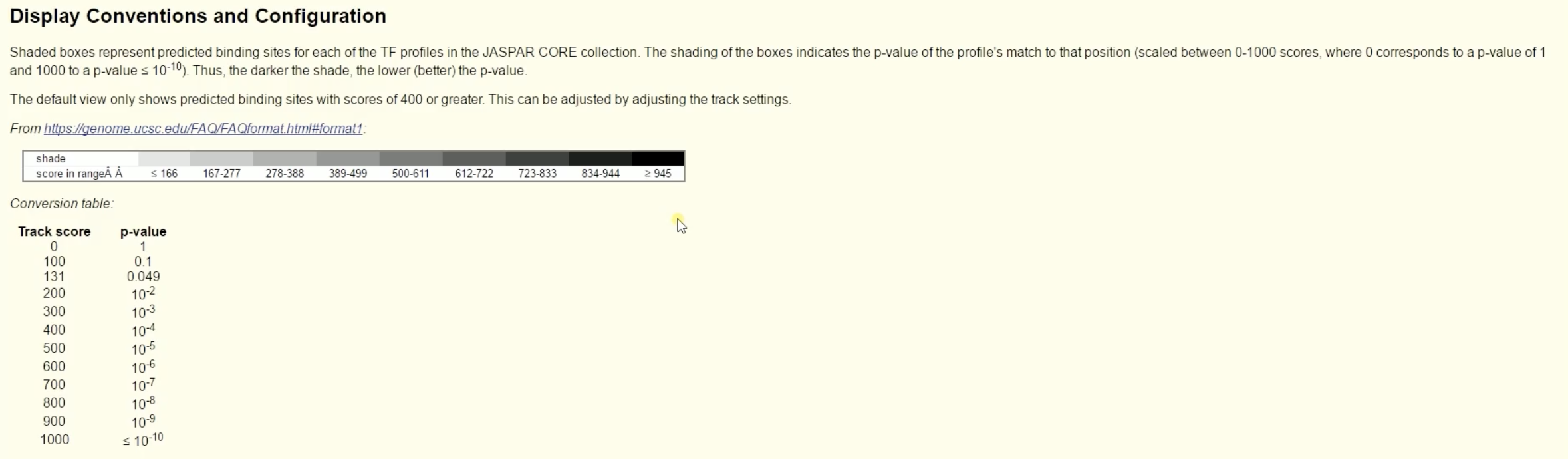
- 转录因子旁边的颜色越浅,表示P值越小,转录因子旁边的颜色越深,表示P值越大;通过Conversion Table可以看到,当选择131的时候P值正好等于0.049。刚刚满足P值小于0.05的临界要求。具有统计学差异。如果阈值低于131,预测结果的意义就不太大了。比如为了获得更多潜在的转录因子候选分子,我们将阈值下调为300,然后点击Submit,这样在图形数据当中所显示的预测结果就会多了很多,不过,虽然预测获得的,可以结合到ABCA1启动子区域的转录因子非常多,而且其中不少是NKX家族的转录因子,甚至是NKX2亚家族的转录因子,例如NKX3-2,3-1,2-5,2-3,2-8等等。但是遗憾的是,预测结果当中并没有NKX2-1,我分析了一下原因,主要还是算法和实验验证的差异,因为根据JASPER算法预测获得的NKX2-1的结合位点,这段序列有16个碱基而实验验证的NKX2-1结合位点只有7个碱基长度上,预测的序列比实验验证的序列长了一倍还多其次,实验验证的两条序列当中只有一条序列,TTGAAAG可以对应预测序列中的9-15位碱基,而实验验证的另一条序列,GAGGAAA完全不能从预测序列中找到对应的序列。这些结果都说明算法预测的结果和实验验证的结果有比较大的差异,也再一次说明生物信息学的预测结果只能作为辅助手段,切勿迷信。
- 另外需要说明的是,在UCSC的图形化数据当中同一个转录因子的结合位点不止一个,一个NKX2-8旁边有一个向左的箭头,而另一个NKX2-8的旁边是一个向右的箭头;箭头向左表示转录因子结合在DNA的负链上;箭头向右表示转录因子结合在DNA的正链上。在选定了转录因子之后,假设我们选定了NKX2-8这个转录因子,如何获得NKX2-8在ABCA1启动子上的结合位点的具体序列呢?这需要我们再回到Jasper网站进行验证。在Jasper的主页上,直接在检索栏输入NKX2-8,点击Search,点选ID序列前的选框,点击网页右侧的Scan按钮,然后把ABCA1启动子序列拷贝到Scan选框内,在Scan选框的下方,有Relative Profile Score Threshold,默认是80%也可以根据需要选择更高的阈值或者降低阈值,最后点击Scan。在ABCA1启动子序列内,总共获得27个潜在的NKX2-8的结合位点,有序列的起始和终止位置信息,还有DNA的结合信息。其中+号表示结合在DNA正链上,-号表示结合在DNA的负链上。由于ABCA1是由DNA负链编码的,因此我们只需要关注结合到负链上的结合位点就可以了。如果觉得候选的结合位点太多,可以提高阈值比如,我们选择90%,然后再点击Scan,这样最终获得了3个NKX2-8的候选结合位点,其中只有2个结合位点是在负链上的。









