使用的文献:Thyroid transcription factor 1 enhances cellular statin sensitivity via perturbing cholesterol metabolism

- 在这一节以及下一节内容当中,我们会介绍和转录因子相关的数据库的使用方法,并进行演示。
一些基础概念
DNA结合结构域以及转录因子结合位点
- DNA结合结构域,DNA binding domain,DBD是一个很重要的概念。转录因子的结构特征之一,就是转录因子可以通过DNA结合结构域和DNA发生结合。有些蛋白分子也可以参与转录的调控,但是分子内部没有DBD,这样的分子只能算作是转录调控因子而不是转录因子。
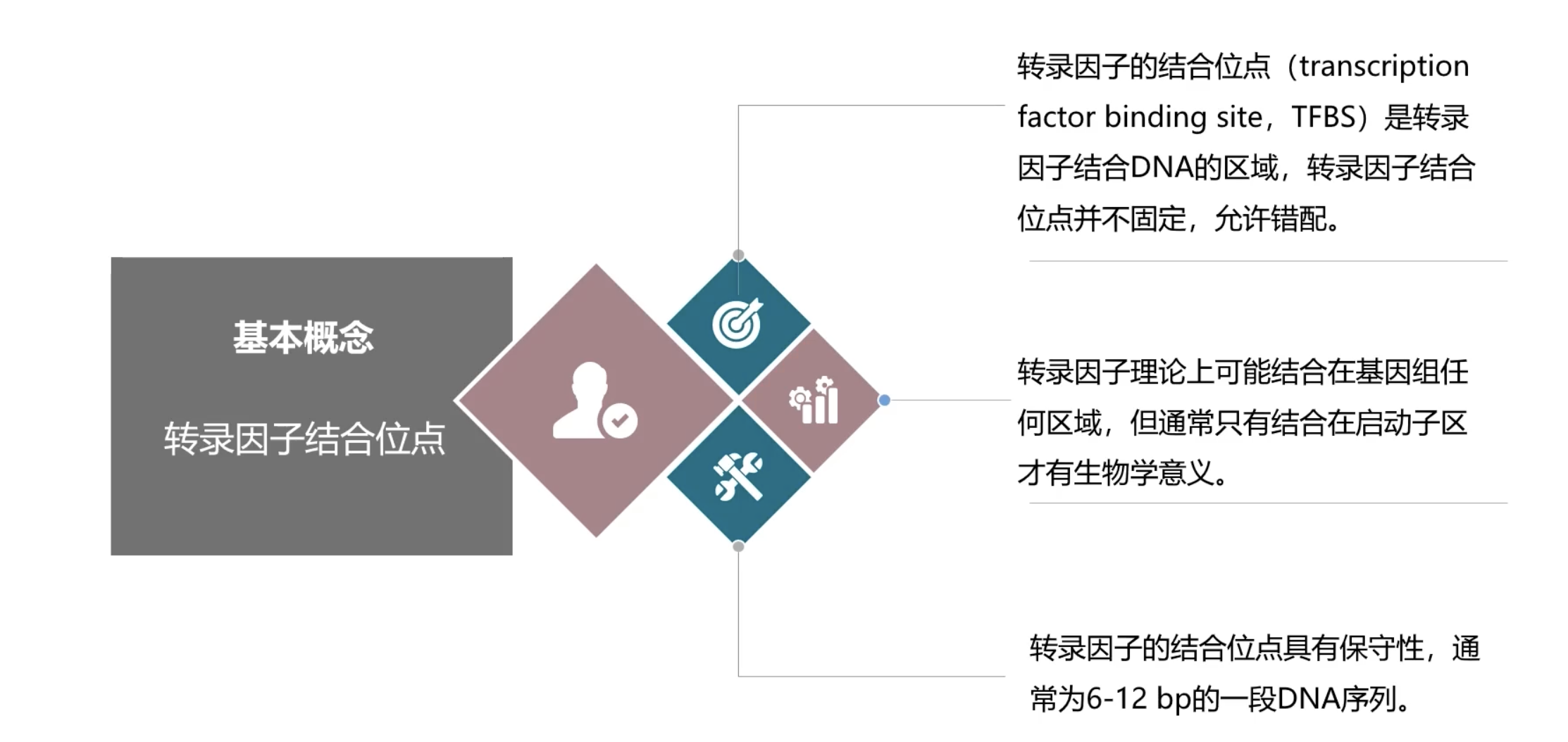
- 和DBD相对应的一个概念是转录因子结合位点,Transcription Factor Binding Site,TFBS。转录因子结合位点是转录因子结合在DNA上的区域,一般这段DNA序列很短长度通常在6到12个bp,有些转录因子的结合位点可能会长一些,一般也不会超过20个bp。而且,某个特定转录因子的结合位点并不是完全固定的,有一些位点具有保守性,而在另一些位点上具有一定的冗余性。
- 如果只是通过生物信息学分析,会发现转录因子可以结合在基因组的任何区域。但是,实际上转录因子只有结合在基因的启动子区域,才能有生物学的意义。DBD只存在于转录因子序列上,而TFBS只存在于DNA序列上。转录因子通过DBD和DNA序列上的TFBS相互结合,因此,DNA结合结构与DBD和转录因子结合位点TFBS是两个很重要的概念。这两个概念既有联系,又有区别。
DNA序列上的转录因子结合位点TFBS有多种表示方式

- 一致性序列consensus sequence:指的是将与同一个转录因子结合的所有DNA片段按照对应位置进行排列,在每个位置上选择最可能出现的碱基,然后把它们组成该转录因子结合位点的一致性序列。在一致性序列中,除了A、T、C、G之外还会出现兼并码,它们代表某个位置上可能出现的碱基组合。一致性序列的优点是此种方法简明易懂,但缺点是不能够反映每个位置上不同碱基出现的概率。
- 序列标识图:在序列标识图中依次汇出Motif中各个位置上可能出现的碱基,每个位置上所有碱基字母的高度总和会反映出该位置上碱基的一致性。每个碱基字母的大小与碱基在该位置上出现的频率成正比。
- 位置频率矩阵Position Frequency Matrix,PFM:实际上位置频率矩阵是转录因子结合位点所使用的序列模体矩阵的一种类型,所谓的序列模体矩阵是根据一系列功能位点的多重比对排列结果,对每一个位置的碱基给出相应的得分值。在位置频率矩阵的表示方式中,假设每一个位置上碱基出现的频率是相互独立,不存在相互影响和干扰,然后通过位置频率矩阵反映出每一个位置上不同碱基出现的概率,矩阵的行表示转录因子结合位点序列上的位置信息,矩阵每一列表示模体相应位置上四种碱基出现的概率。位置频率矩阵可以有两种表示方式一种是数学上的集合论表示方式,另一种是可视化二维表格的表达方式。
常用的转录因子预测网站
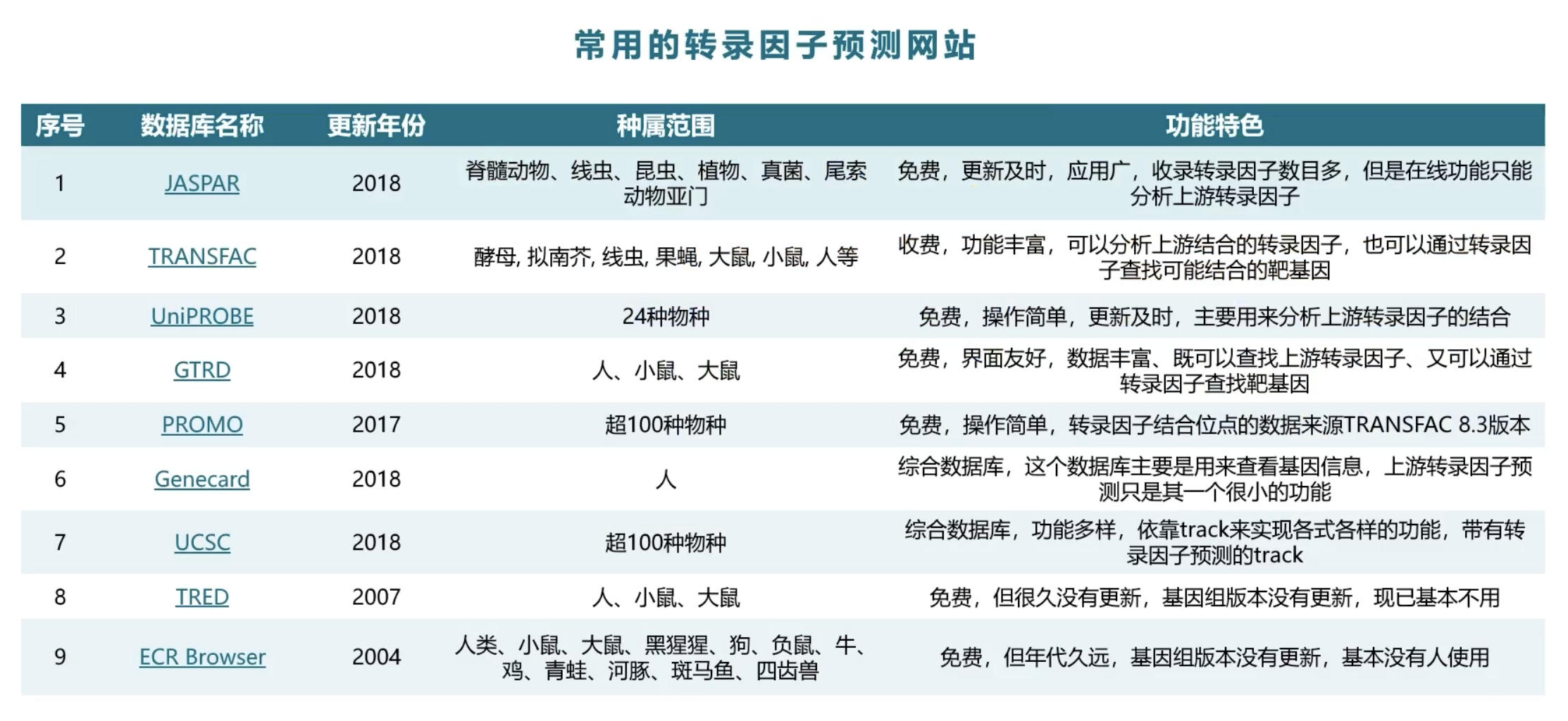
- 能够进行转录因子预测的网站还是有一些的,但是能直接进行上游转录因子预测的网站数目不算很多。在上图中仅列出了其中一部分较为出名的网站,还有一部分网站可以提供chipseq这些数据的下载,后续的分析预测需要比较专业的生物信息学基础,因此没有在网站中列出来。即便是已经在上图当中列出的网站,它们都是拿已经收录在自己数据库内转录因子去分析去预测,如果想要预测数据库中没有收录的新的转录因子,这些网站都不太合适使用。不过好在这些数据库经过那么多年的积累,转录因子的数据库信息比较庞大,常见转录因子都已经包含在内了。由于转录因子相关的数据库比较多,套路课中主要介绍最常用的Jasper数据库以及UCSC数据库的使用。
数据库预测的潜在问题
- 转录因子结合位点预测的原理是基于热力学上的亲和力,而非实际发生在具体细胞内的结合。因此会存在大量假阳性预测位点,也就是说虽然网站预测到了序列上会存在某些转录因子A的结合位点,但在实验当中却完全无法验证。这是目前数据库预测转录因子结合位点所带来的最大的问题,如果对于预测结果不加筛选,直接进行实验验证,往往事倍功半。
- 其次,转录因子结合位点不是完全单一化的序列,而是序列中很多位点包含冗余性;再加上转录因子结合位点预测时允许碱基的错配和冗余性的发生,更加导致了直接预测的结果内存在大量假阳性预测位点。
- 转录因子发挥作用时,它不是一个因子在战斗,而是需要很多转录调控因子,或者组成转录因子复合物的其他分子的协同作用。但是,使用网站预测的时候,算法是没有办法把这些因素考虑在内的。
- 染色体的结构,尤其是组蛋白的乙酰化修饰,会极大的影响转录因子的结合以及下游基因的转录,这也是转录因子预测网站在设计算法时没有办法考虑到的因素。
一些优化改良的方法
- 由于转录因子往往和受到调控的基因具有表达上的显著相关性,因此转录因子结合位点的预测,和转录因子和靶基因之间的表达相关性数据可以进行联合分析,这样会排除很多假阳性的结果。
- 由于不同的网站采用不同的预测算法,虽然每一种算法都会有缺陷,但是如果采用多个网站的不同算法,然后取交集而获得的转录因子预测结果,可以在一定程度上比只采用单个网站的预测结果更加理想。
- 由于转录因子往往会形成转录复合物而发挥作用,而且通常情况下一个转录复合物内会含有多个转录因子,因此如果一个基因的启动子含有的序列可以和同一个转录复合物内包含的不同转录因子都有结合的存在,也可以提高预测的准确性。
演示
- 我们会在本节内容以及下一节内容当中演示:
- 如何获得转录因子内相关结构域的结构信息
- 如何通过Jasper网站获得转录因子结合位点的相关信息
- 如何通过UCSC联合Jasper预测把基因启动子区域内的候选转录因子
- 我们首先演示的是如何获得转录因子相关结构域的结构信息,有两个常用的网站都可以帮助我们获得这些信息。这两个网站分别是NCBI GENE数据库,以及Uniprot数据库。
- 首先我们进入NCBI主页,然后在标签栏内找到GENE这个标签,随后在搜索栏内输入NKX2-1,也就是TTF1转录因子的正式的基因名称,点击搜索。在这里我要特别强调一下,不要在检索框内输入TTF-1,因为有一个分子名叫Transcription Termination Factor 1,也就是转录中止因子1,它的基因正式名称也是TTF-1。所以如果你在GENE数据库当中输入TTF-1,找到的基因其实是转录中止因子1,而不是NKX2-1,在这里的话会误导你的检索结果。
- 为了获得转录因子的结构域信息,我们找到NCBI RefSequence一栏,在网页的右侧有导航栏,直接点击即可。可以看到NKX2-1有两个异构体,Isoform1和Isoform2,我们分别点击两个Isoform的蛋白序列,请大家注意NM开头的是mRNA序列,NP开头的才是蛋白序列。
- 我们先看Isoform1的蛋白序列,其中CDS就是Coding Sequence编码区,是Isoform1蛋白的全序列,这个蛋白长度是401个氨基酸Region就是NKX2-1蛋白中的结构域,点击Region,相应的区域就会进入高亮的状态,这样就很容易获得Region区域的序列。同时,网站还给出了Region的相关信息,这个结构域的名字是Homeo box它也是介导了NKX2-1和DNA相互结合的结构域。
- 类似的,我们也能够获得Isoform2的蛋白序列和相应的结构域信息,NKX2-1的Isoform2全长是371个氨基酸同样含有一个Homeo box,从NCBI数据库上查询这些信息比较容易,但是相对而言,NCBI对于蛋白序列的数据整合度做得不算太好,如果只想知道一些和蛋白结构域相关的简单信息,NCBI可能就够了。但是,如果想要了解更深入的信息,NCBI不合适,相反,Uniprot数据库更加符合要求。
- 我们先登录Uniprot数据库,检索栏直接输入NKX2-1进行检索,找到人的NKX2-1并点击进入相关的页面,在NKX2-1的页面当中首先会对分子的功能做简单的介绍,然后紧接着就是region相关的信息,在这里需要大家注意,因为NKX2-1有两个isoform,Uniprot这里列出的这个region对应的是哪一个isoform需要大家点击进入相应的页面才能够确定。点击Positions进入新的页面,找到Sequence Feature这一栏,然后才能确定是针对371个氨基酸NKX2-1 isoform的。在Uniprot网页当中也有Sequence这一项,在这一栏内同样展示了NKX2-1的两个isoform,但是命名不一样。一个叫isoform1,全长371个氨基酸;而另一个叫isoform3,全长401个氨基酸。其实Uniprot提供的信息和NCBI提供的信息是一样的,只是对于分子的命名不同,不要因为命名的问题就以为它们是不同的异构体。
- 通过Uniprot网站我们所要获取的信息是:NKX2-1关键结构域的结构信息,所以我们要在网站上找到Structure以及3D Structure Databases。
- 我们介绍3D结构的数据库以Protein Model Portal作为一个示例首先点击进入网站,在网页的最上方是Summary,最长的这条暗红色的长条它表示的是371个氨基酸长度的NKX2-1分子,它的下面有很多条线段,鼠标移动到线段之上,相应的数据条就会高亮,比如绿色线条表示大鼠的TTF-1分子的Homeo Domain,再比如深蓝色的线段表示的是来源于SwissModel数据库的3D结构数据。
- 由于3D数据库有好几个,究竟怎么选关键看Sequence Identity,可以把这个参数理解为序列的保守性,大鼠和人的NKX2-1的Homeo Domain序列保守性是100%完全相同,说明物种间的保守性还是比较高的,再看不同的数据库,来源于Swiss Model的Sequence Identity同样也是100%,那么当然我们就更愿意选择Swiss Model的数据库,点击Show按钮。
- 在新的网页中,3D结构图就出现了。这张3D结构的图片可以通过鼠标滚轮放大或者缩小,长按鼠标左键可以拖动,然后就可以360度随意翻转结构。点击鼠标右键可以下载3D结构图,把鼠标移到图片上还可以看具体的氨基酸信息,这些信息都会显示在图片的右下角
- 如果想要了解更多的信息,可以点击图片左侧的Swiss Model按钮,直接进入Swiss Model网站。Swiss Model网站的图形化和界面的友好度是非常让人赞叹的。
- 首先在Swiss Model网站上通过鼠标滚轮可以放大缩小,拖动图形可以360度全景展示和翻转,包括鼠标移到相应的图形上会显示具体氨基酸的信息,这些功能都和Protein Model Portal是一样的,因为本身Protein Model Portal就是引用的Swiss Model的数据。我们再介绍一些Swiss Model独有的特色,首先整个分子画成一个圆,在这个圆形上还有一段彩色的线段,这段彩色线段就是Homeo Domain结构域;在分子的下方还有相应的氨基酸序列,除了这个彩色线段之外,另外有一段紫色的线段和一段蓝色的线段,外加几个橙色的小竖条,大家可以参考右侧的Sequence Features,紫色表示介导蛋白蛋白相互作用的结构域,蓝色表示介导DNA结合的结构域,橙色的小竖条表示自然存在的序列突变。
- 网页的右侧有Homeo Domain的3D结构域,结构图显示Homeo Domain包含了三个α螺旋,而且这个3D结构域的图形也是彩色的,3D的色彩和左侧分子上的线段的彩色是一致的,所以结构域的N-末端就用蓝色表示,结构域的C-末端就用红色表示。这样通过色彩的不同,我们就可以分辨3D结构域的方向,我们也可以选择其他形式表示结构域的3D形态,比如我们选择Lines就是使用线图的方式表示结构域的3D结构,也可以根据需要选择其他的展示形式;还有一个非常有用的功能就是播放键,点击播放键3D结构开始旋转,在旋转的过程中,还可以通过鼠标拖动的方式调整分子的角度,从而获得更好的展示效果。






