GSEA介绍
- Gene Set Enrichment Analysi,即基因富集分析
- 博劳德研究所(Broad Institute)开发的,针对全基因组表达谱芯片数据进行分析的工具
- 综合了现有的,对基因的定位,性质,功能和生物学意义等信息的基础上,构建的分子标签数据库(MSigDB,molecular signature database),在该数据库内,设置了多个功能基因数据的子集
- 在对全基因组表达谱芯片数据进行表达分析分析的时候,首先确定分析的目标,然后从MSigDB中,选择一个或多个功能基因集进行分析,再将经过标准化处理的数据进行表达值大小排序,最后比较这些基因与选择的功能基因的子集的符合程度,从而发现研究的目标基因表达谱数据,是否在选定的功能基因子集中有某种共同表达的趋势,最后给出富集分数
- 可以考虑让专业的数据公司帮我们完成GSEA数据分析的内容,“让专业的人做专业的事”,但通常需要额外收费
GSEA分析的特点
- 分析的是基因集合而不是单个基因(GO)or 少数基因(Pathway),可以获得的信息更加接近细胞内的,真实的生物学状况
- 将基因与预定义的基因集合(MSigDB)进行比较
- 富集分析
GSEA预先设定的基因子集
H-Hallmark Gene Sets
C1-Positional Gene Sets
C2-Curated Gene Sets
- 共识基因集合,数据来源主要是信号通路,文献报道的信息
C3-Motif Gene Sets
C4-Computational Gene Sets
- 计算基因集合
- 主要研究生物信息学的小伙伴会用的非常频繁
C5-GO Gene Sets
C6-Oncogenic Gene Sets
- 癌症特征基因集合
- 直接来源于肿瘤相关的microarray的数据
C7-Immunologic Gene Sets
GSEA分析的关键参数
ES值:富集分数
- GSEA分析得到的原始结果,反映了把全部的杂交数据排序之后,在此序列的前部或者后部,一个功能基因子集的富集程度
- 扫描排序序列时,当出现一个功能基因子集的基因时,ES值上升,反之下降,所以ES值是一个动态变化的数值,在一般的数据图片中,是不会标注ES值的,如果一定要标注,那图片中只会标注出最大ES值
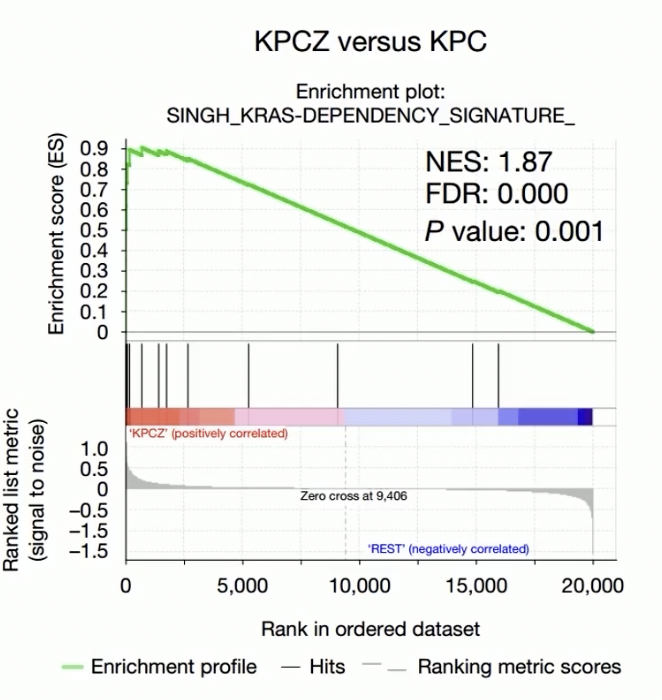
- 上图中绿色的线就是ES值的标准变化曲线,ES值在不同的位置,数值是不一样的
- 对于一张完整的GSEA数据图片,图片的上半部份可以看到是一条ES曲线,中间部份有一根根竖线,这些是这条信号通路当中,出现在芯片数据子集的基因所处的位置,整张图片最下面的部份是杂交数据的排序序列,总共三张图组成
NES值
- normalized enrichment score,标准化的富集分数
- 在比较芯片数据集在不同功能基因子集的富集程度时,需要对ES进行标准化处理,NES是一个数值,是不变的
FDR
- False Discovery Rate,错误发现率
- 描述的是估算的可能性,也就是当一个功能基因子集的NES确定之后,判断其中包含的,错误的阳性发现率
- 举例:FDR=25%表示在特定的ES值下,四次当中就可能出现一次错误
- 在GSEA分析中,越小越好
p值
- 只是名义上的统计显著性
- 通常关注的是p<0.05,但是在GSEA分析中,只看p值会有很大的问题,因为FDR进行了功能子集基因大小,以及多重检验的矫正,而这些矫正在p值中是没有进行的,所以p值仅仅称作名义p值
- 特殊情况:p值很低,FDR值很高,这说明和其他功能基因子集相比,着重研究的基因的富集程度没有想象中那么高
- 基因芯片数据的样本量太小
- 杂交信号弱
- 所选的功能基因子集没能很好反映样本的生物学意义
Anchorage-independent Growth Assay
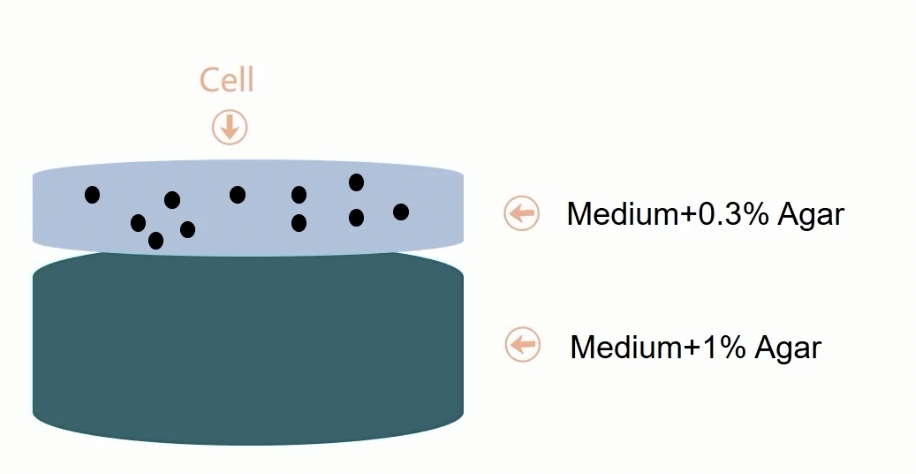
- 非贴壁生长实验/非依赖锚定的增殖实验
- 从原理上讲,就是传统的克隆形成实验的”3D培养版本”
- 对于传统的克隆形成实验,就是把肿瘤细胞的密度种得很低,使细胞和细胞间没有黏附,也没有信号传导,这样单个肿瘤细胞形成的克隆就可以计数,可以评估肿瘤细胞形成克隆的能力
- 肿瘤细胞和正常细胞存在区别:肿瘤细胞可以不依赖黏附就形成增殖,而正常细胞必须依赖黏附,为了区分肿瘤细胞和正常细胞对于黏附的依赖程度,所以就就依赖于传统的克隆形成实验,开发了这种锚定非依赖的克隆形成实验
- 琼脂的特性:比较高的温度下融化,比较低的温度下凝固,1%琼脂区域在凝固后,可以形成比较坚固的结构,为整个实验提到支撑的作用
- 0.3%琼脂的培养基:浓度比较低的培养基,这种培养条件下琼脂呈现出半流质的状态,可以让肿瘤细胞在这种环境中生长、增殖,而且氧气在这种浓度的琼脂培养基内很容易发生渗透,为肿瘤细胞的存活、生长和增殖提供养分
BrdU染色实验
- BrdU是胸腺嘧啶衍生物,在结构上与胸腺嘧啶是非常类似的,在细胞分裂时,BrdU可以替代胸腺嘧啶T,被整合到新合成的DNA链中,并存留在基因组中
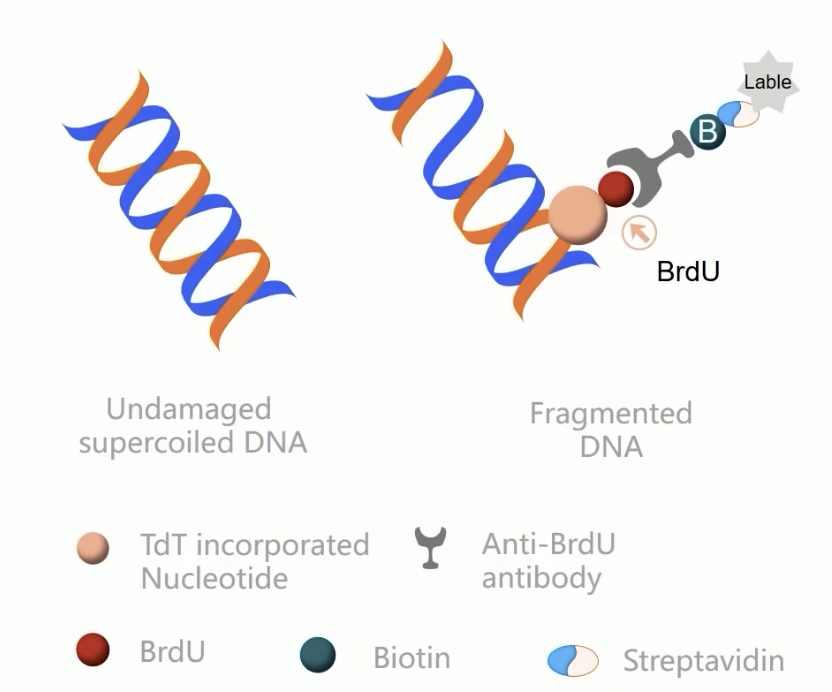
- 在随后的实验中,可以通过抗BrdU抗体,识别这些新合成的,且有BrdU渗入的基因组,最后通过荧光检测,统计BrdU阳性的细胞,提示这些细胞发生过增殖和分裂
荧光素酶报告基因实验Luciferase Assay
- 在这个实验中,首先要构建一个质粒,质粒中含有荧光素酶基因,基因的上游是克隆一段想要探究的转录因子的结合域,在本篇paper中,克隆的是FOXO基因的结合域FOXO Binding Domain,FOXO转录因子可以结合在这段序列上,启动下游的基因转录
- 这样,细胞内就产生了荧光素酶蛋白,这个酶可以催化底物的降解,产生的荧光的强度可以通过酶标仪读出,通过荧光的强度来反映细胞内FOXO和FOXO binding domain结合与调控的强弱
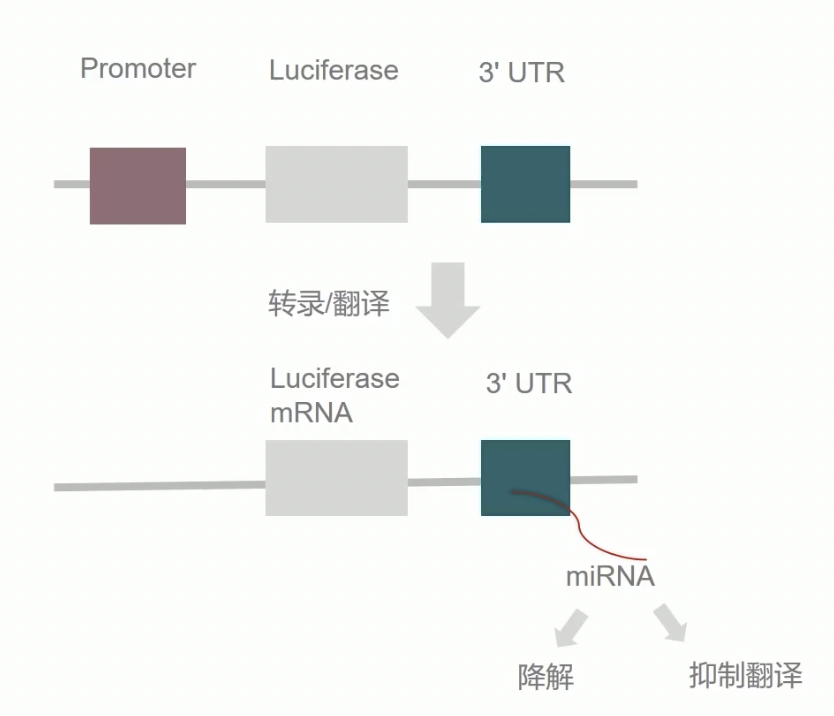
- 对于荧光素酶实验,除了用来研究转录的强弱之外,还可以用来研究microRNA
- 对于microRNA研究而言,研究也从构建质粒开始,但是这个质粒的结构与前面的转录因子研究有差异
- Luciferase mRNA的上游是普通启动子,但下游要把miRNA的潜在靶点3’-UTR序列克隆到荧光素酶基因的下游
- 在没有miRNA的情况下,启动子的启动可以正常开始Luciferase基因的转录和翻译,产生Luciferase蛋白,进而产生荧光;如果细胞内存在miRNA的过表达,Luciferase的mRNA可以正常产生,但其可能被对应miRNA抑制功能或直接分解(因为这条mRNA上有miRNA对应的3’-UTR序列),不管是哪一种机制,都会导致Luciferase蛋白的含量显著降低,导致酶促反映的荧光读值降低,证明miRNA可以与我们引入的某个基因的3’-UTR区域发生特异性结合
- 如果要设计这个实验的rescue实验,就可以把miRNA结合的3’-UTR的序列做几个点突变,让miRNA不能再识别,如果实验作出来荧光的强度恢复了,那么就说明miRNA与target 3’-UTR直接的结合是特异性的






